一.前述
密度聚类是一种能降噪的算法。很多时候用在聚类形状不规则的情况下。
二.相关概念
先看些抽象的概念(官方定义):
1.![]() :对象O的是与O为中心,
:对象O的是与O为中心,![]() 为半径的空间,参数
为半径的空间,参数![]() ,是用户指定每个对象的领域半径值。
,是用户指定每个对象的领域半径值。
2.MinPts(领域密度阀值):对象的![]() 的对象数量。
的对象数量。
3.核心对象:如果对象O![]() 的对象数量至少包含MinPts个对象,则该对象是核心对象。
的对象数量至少包含MinPts个对象,则该对象是核心对象。
4.直接密度可达:如果对象p在核心对象q的![]() 内,则p是从q直接密度可达的。
内,则p是从q直接密度可达的。
5.密度可达:在DBSCAN中,p是从q(核心对象)密度可达的,如果存在对象链,使得![]() ,
,![]() 是
是![]() 从关于
从关于![]() 和MinPts直接密度可达的,即
和MinPts直接密度可达的,即![]() 在
在![]() 的
的
![]() 内,则
内,则![]() 到
到![]() 密度可达。
密度可达。
6.密度相连:如果存在对象![]() ,使得对象
,使得对象![]() 都是从q关于和MinPts密度可达的,则称
都是从q关于和MinPts密度可达的,则称![]() 是关于
是关于![]() 和MinPts密度相连的。
和MinPts密度相连的。
PS:是不是很抽象 ,所以官方定义永远是官方定义确实理解不了。然后再看些非官方定义,其实就大概明白了。
先上图:
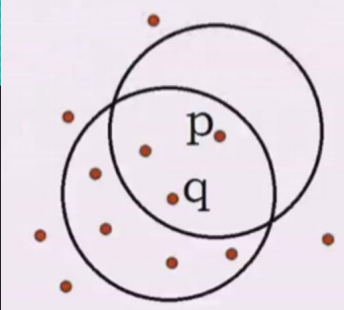
解释下:这里有几个关键的概念。
领域其实就是某一个半径内,假设半径为5,我们先看P点以半径为5画的圆中包含3个点,而q点以半径为5画7个点 7>5,所以q就叫做核心对象。q不是核心对象。理解就是这么简单,再看看什么叫密度可达,见下图:
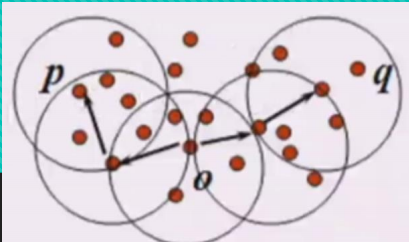
0点以半径为5画圆与p点以半径为5画圆有交集,即O点以半径为5的领域内有以P为中心店半径为5的领域内的点,则O密度可达P,O也密度可达q(在边界交点也算)。
从o点能密度可达p,也能密度可达q,则p,q叫密度相连。
再比如:
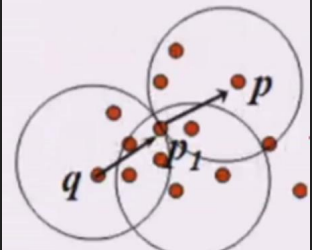
q密度可达p1,p1密度可达p,则q密度可达p(间接的也是密度可达)!!!!
这里需要两个参数注意下:r半径,m阈值,即以r为半径内所包含的点,只有大于m阈值的点才能叫核心对象。
以上理解了这些概念,但跟聚类有什么相连,实际上簇就是密度相连的最大的集合。即一个簇就是最大的密度相连的集合。
如果一个点不是核心对象,也就意味着不能密度可达,所以就是噪声点。(通俗理解就是一个点都不能画圆,怎么会有密度可达呢?)
比如下图:
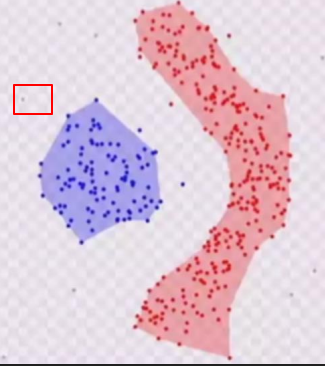
就是噪声点。
PS:总结下规律:
给定的m不够簇就会变多,比如下图:m分别是5,3,2

解释:当是5的时候,圈红的边缘点不是核心对象,所以不能画圆,所以不会密度可达。当是2的时候,半径内的值大于阈值所以是核心对象,那么这堆数据有可能密度相连,形成一个簇。这也就是簇变多的原因。
代码:
# !/usr/bin/python # -*- coding:utf-8 -*- import numpy as np import matplotlib.pyplot as plt import sklearn.datasets as ds import matplotlib.colors from sklearn.cluster import DBSCAN from sklearn.preprocessing import StandardScaler def expand(a, b): d = (b - a) * 0.1 return a-d, b+d if __name__ == "__main__": N = 1000 centers = [[1, 2], [-1, -1], [1, -1], [-1, 1]] data, y = ds.make_blobs(N, n_features=2, centers=centers, cluster_std=[0.5, 0.25, 0.7, 0.5], random_state=0) data = StandardScaler().fit_transform(data) # 数据的参数:(epsilon, min_sample) params = ((0.2, 5), (0.2, 10), (0.2, 15), (0.3, 5), (0.3, 10), (0.3, 15)) matplotlib.rcParams['font.sans-serif'] = [u'SimHei'] matplotlib.rcParams['axes.unicode_minus'] = False plt.figure(figsize=(12, 8), facecolor='w') plt.suptitle(u'DBSCAN聚类', fontsize=20) for i in range(6): eps, min_samples = params[i] model = DBSCAN(eps=eps, min_samples=min_samples) model.fit(data) y_hat = model.labels_ core_indices = np.zeros_like(y_hat, dtype=bool) core_indices[model.core_sample_indices_] = True y_unique = np.unique(y_hat) n_clusters = y_unique.size - (1 if -1 in y_hat else 0) print(y_unique, '聚类簇的个数为:', n_clusters) plt.subplot(2, 3, i+1) clrs = plt.cm.Spectral(np.linspace(0, 0.8, y_unique.size)) print(clrs) for k, clr in zip(y_unique, clrs): cur = (y_hat == k) if k == -1: plt.scatter(data[cur, 0], data[cur, 1], s=20, c='k') continue plt.scatter(data[cur, 0], data[cur, 1], s=30, c=clr, edgecolors='k') plt.scatter(data[cur & core_indices][:, 0], data[cur & core_indices][:, 1], s=60, c=clr, marker='o', edgecolors='k') x1_min, x2_min = np.min(data, axis=0) x1_max, x2_max = np.max(data, axis=0) x1_min, x1_max = expand(x1_min, x1_max) x2_min, x2_max = expand(x2_min, x2_max) plt.xlim((x1_min, x1_max)) plt.ylim((x2_min, x2_max)) plt.grid(True) plt.title(u'epsilon = %.1f m = %d,聚类数目:%d' % (eps, min_samples, n_clusters), fontsize=16) plt.tight_layout() plt.subplots_adjust(top=0.9) plt.show()
r半径太大就会聚类到一起:如下图
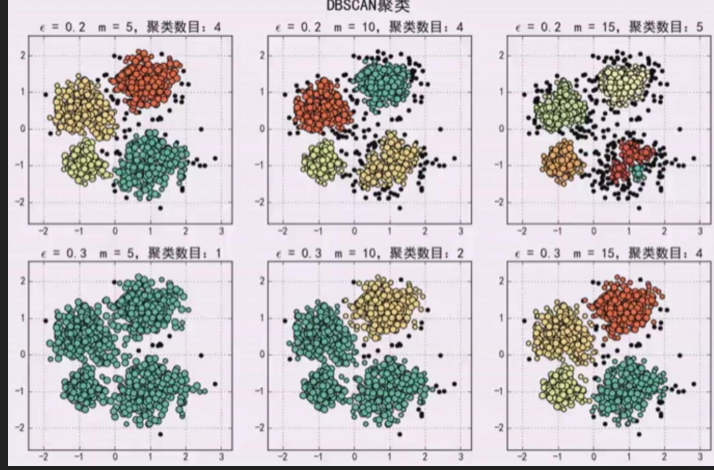
所以Finally总结:要大一起大,要小一起小,参数这是最合适的。比如2,6图是合适的,4个簇。
未完待续,持续更新中。。。。。。。。。。。。