2019年7月31日 大雨
1 已下载eeglab和letswave工具包,在matlab设置好工作路径,将工具包的图形界面调用出来;
2 使用letswave6工具包进行操作。导入数据的时候,我的设备是neuroscan,储存了cnt数据,所以在导入数据的时候,选择了如下图1:
 图1
图1
2.1 如此,便导入了epochs*channels*x*y*z*t的六维数据。在图2红色区域看到此信息:
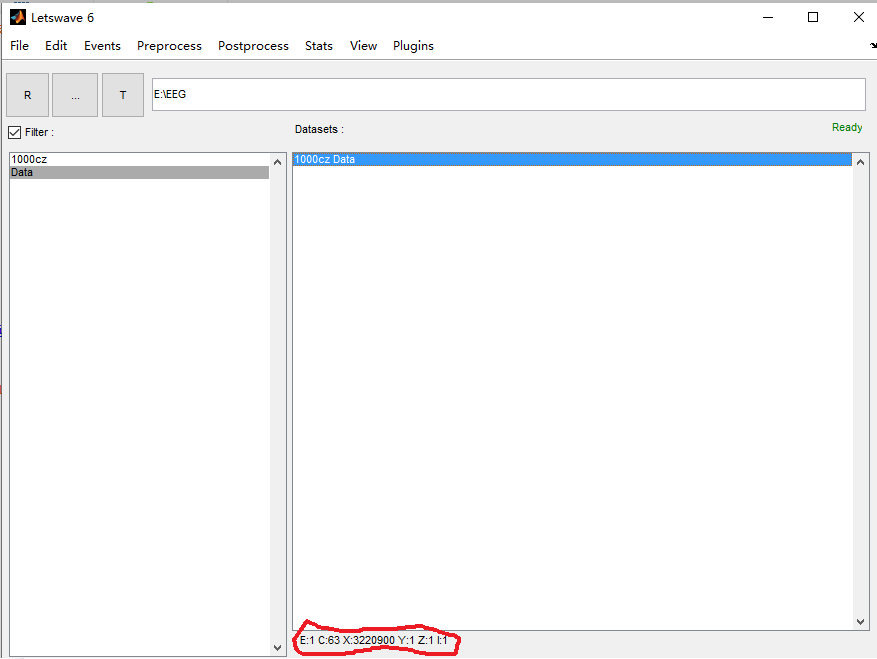 图2
图2
2.2 双击图二蓝色区域“data",即可观看所有通道的信号,如图三:
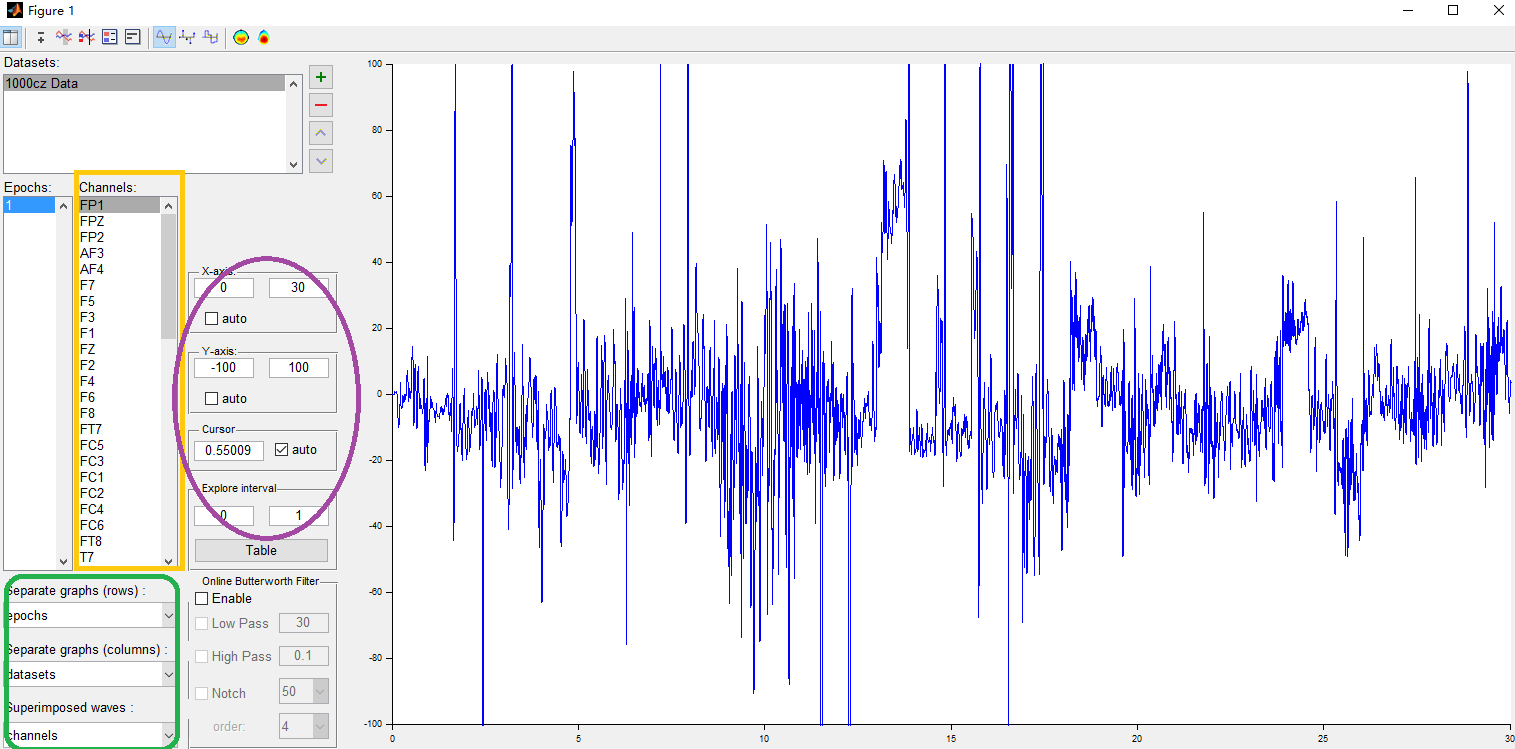 图三
图三
tips1: 可以单独对图三黄色区域的通道信号进行观看,也可以同时对多个通道信号进行观看。借助“shift"和”ctrl“键,选择兴趣通道;
tips2:图三紫色区域的横纵坐标可改变;
tips3:图三绿色区域的横纵坐标可以变换选择,不同选择有不同的信号对比效果,如图四所示:


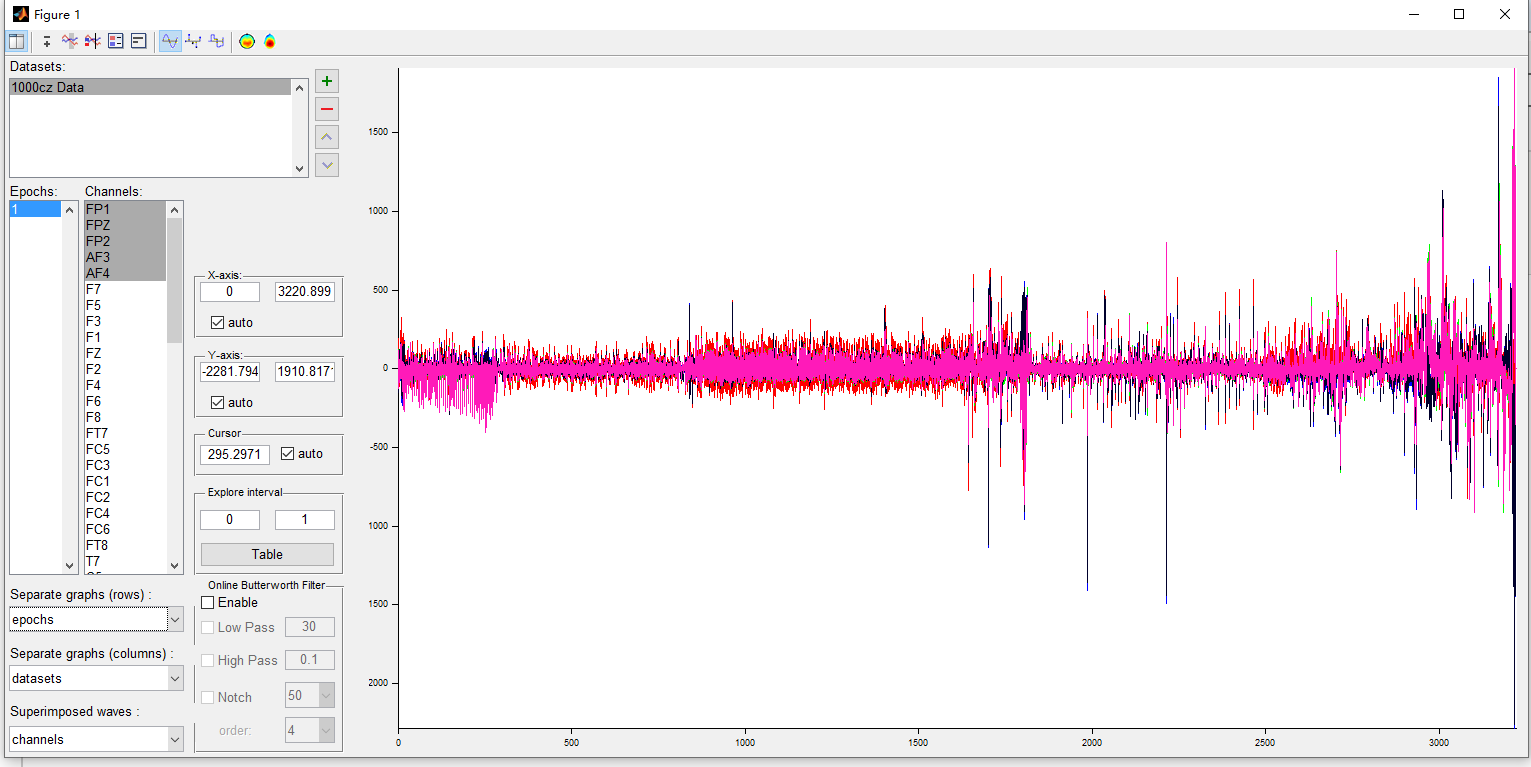 图四
图四
3 数据预处理-电极位置配置和采用率不会,在letswave里不用管(老师说的);
4 去除无用通道流程:2 -> 3 -> 1(以下流程的操作步骤同理展示);将所有通道“select all”移至右边,再将要删除的通道选中,“remove”,可多选。最后,“run”(绿色三角形或最底部)。最后,将生成“sel_cha”开头的新文件。
 图五
图五
5 带通滤波:选中datasets栏,上一步骤生成的“sel_cha”开头的新文件,再 3 -> 6 -> 7(Butterworth.fileter),bandpass参数设置,根据自己的感兴趣频率定。
6 修换通道:(有些通道的信号可能由于前期试验导致收集的数据不好,所以,可以用周围通道的数据进行平均数替代。) 选中上一步骤生成的数据,再
2 -> 1 -> 3。橙色区域点击想要替换的通道,粉色区域是替换通道,紫色是离待替换通道最近的通道个数,可自行选择,然后点击黑色区域,程序即选择了离待替换通道最近的其余通道。最后点击“run”
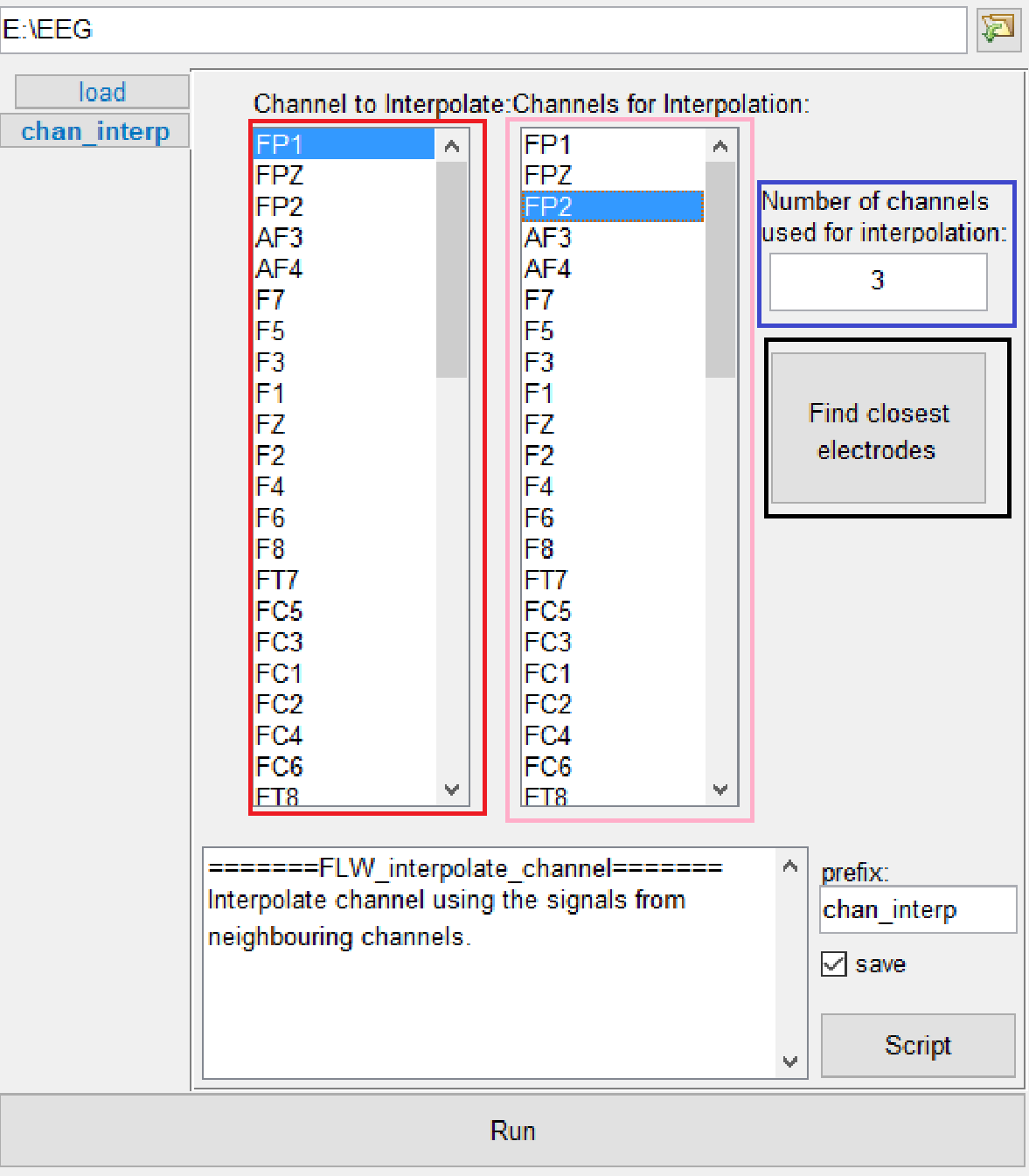 图6
图6
7 去伪迹:界面流程 3 -> 4 -> 1
理论: 在MRI扫描仪中进行电生理记录时,主要有三种伪迹:1)射频脉冲导致的EEG信号,由于其频率在兆赫范围内与EEG信号的频率分布差异很大,通过低通滤波即可消除;2)由于梯度磁场切换引起的梯度伪迹,这种信号的典型波幅是EEG信号的100~1000倍且它的频率在EEG范围之内,因此难以去除,现有方法主要有:自适应伪迹相减法(AAS)及最优基组法(OBS);3)高场强条件下由心跳及主动脉弓处血流冲动产生的伪迹,即心电伪迹,用ICA可消除。
实践:3 -> 4 -> 1后,number of conponents选择第二项“decide by user”,Components numbers手动输入(去除无用通道后的通道数-替换通道个数),“run”
2019年8月1日 阴
8 数据分割:界面流程 3 -> 1 -> 2
理论:针对ERP事件相关数据(要有刺激段的数据),EEG数据记录的是受试者进入磁共振后的一切序列,包括任务态,所以应该分段。
实践:橙色区域为标记marker,选择感兴趣marker进行分段;黄色区域为分段具体信息,“开始(-1)”、“结束(2)”、“持续时间(3)”,代表:从marker开始记录的前1秒到后2秒,总持续时间3秒。“run”
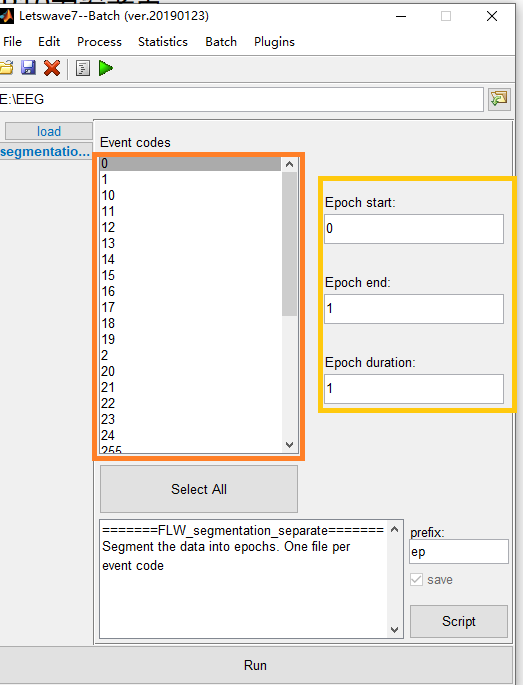 图7
图7
9 重参考:界面流程 3 -> 2 -> 1
通常“new reference”选择双侧乳突(TP9、TP10)作为参考点,"apply reference to"全选,“run”。
10 基线校正:界面流程 3 -> 3 -> 1
疑惑:老师说点进去后参数都不用改,直接run就行。但是这个范围跟数据分割这一步设置的范围不应该一样吗?
11 average:界面流程 3 -> 7 -> 1
直接run
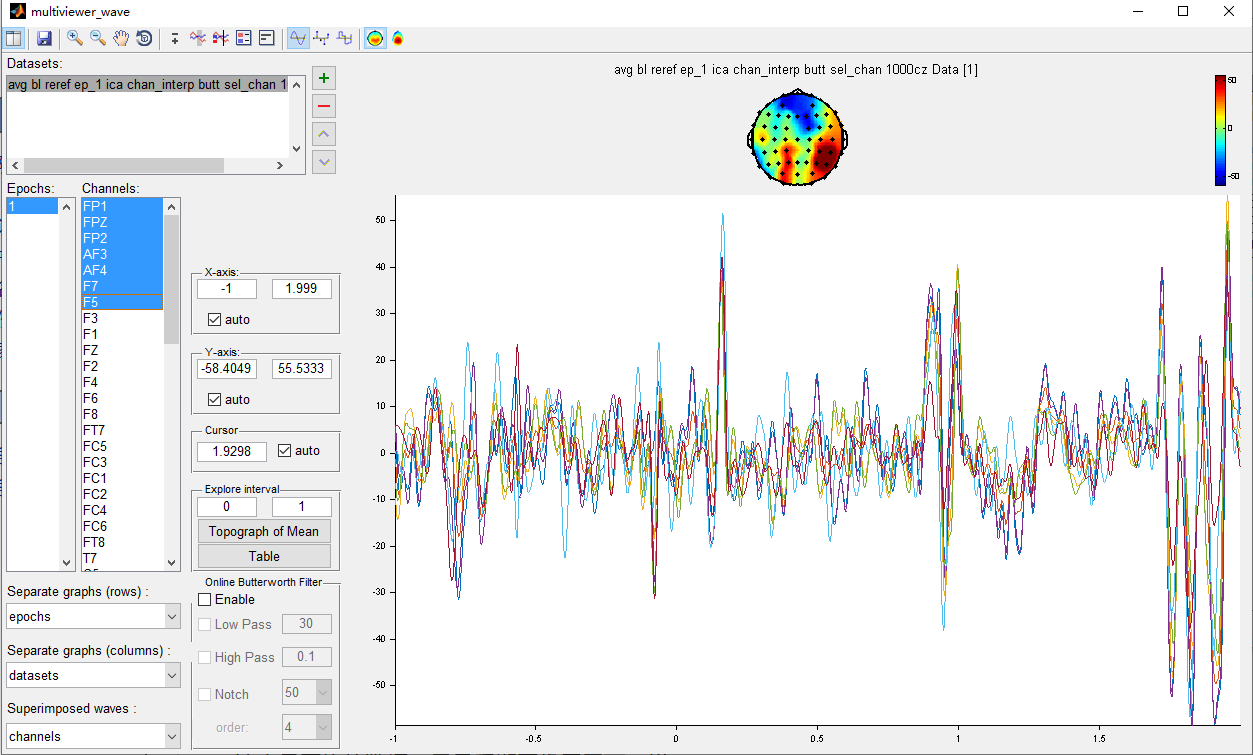 图10
图10
12 cut:选用基线校正数据 then 界面流程 3 -> 3 -> 1
这也是平均的数据,只是后期图像展示不一样。处理好后的数据展示如图9:
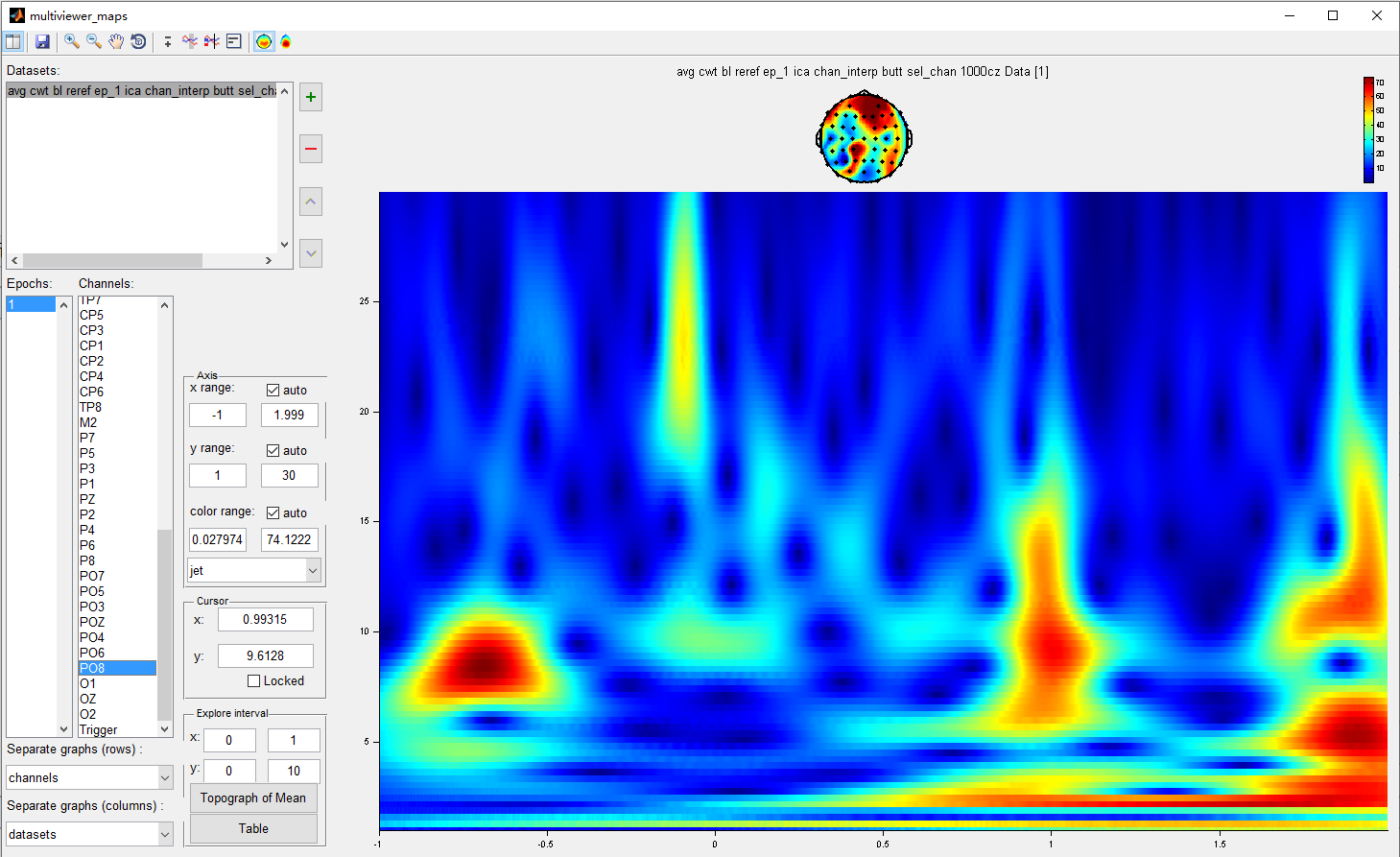 图9
图9
以上步骤只是对单个数据的预处理,如果是要批处理,还是按照以上步骤,每一步进入操作界面,设置好参数,但是不要点击“run”,知道最后一步时全部选好了,再“run”。如图10 标记的红色区域,全部程序好了之后,最终run一把。期间,在进行下一步骤时的操作程序时,需要加载上一步生成数据集时,点击“load”,在弹出框中双击需要文件(系统模拟的,现在并不是真实的有这个数据集)。程序运行过程中,新生成的数据集会逐步保存。
 图10
图10