通常我会用simpleitk来读取dicom文件,主要是为了将dicom文件转换为numpy矩阵,便于输入神经网络,读取dicom文件可分为两种情况,一.单独的dicom文件 二.一系列dicom文件,前者只是一张切片,通常是X光片,后者是很多张切片,合在一起通常代表CT图像。
一.
读取dicom文件
file = sitk.ReadImage(filepath)
获取基本信息,大小,像素间距,坐标原点,方向
file.GetSize()
file.GetOrigin()
file.GetSpacing()
file.GetDirection()
输出信息类似下面这样

还可以获取很多其它信息,这些信息以字典的形式存储,称为元数据
上面是字典的键,具体为什么是这种形式,还不太清楚
字典的值形式如下
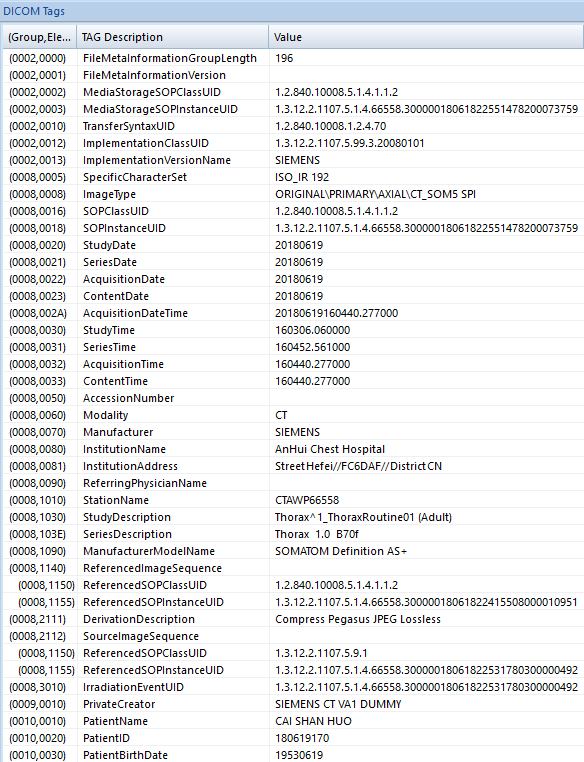
上面输出的只是一小部分信息,利用microDicom软件打开一个dicom文件,可以看到详细的元信息
当然,最最重要的还是像素矩阵,这是我们用来训练模型的原料啊
pixel_array = sitk.GetArrayFromImage(file)#这个file是之前读取出来的文件
二.
读取dicom序列
reader = sitk.ImageSeriesReader() reader.MetaDataDictionaryArrayUpdateOn()#这一步是加载公开的元信息 reader.LoadPrivateTagsOn()#这一步是加载私有的元信息 series_IDs = sitk.ImageSeriesReader.GetGDCMSeriesIDs(directorypath)#根据文件夹获取序列ID,一个文件夹里面通常是一个病人的所有切片,会分为好几个序列 dicom_names = reader.GetGDCMSeriesFileNames( directorypath,series_ID)#选取其中一个序列ID,获得该序列的若干文件名 reader.SetFileNames(dicom_names)#设置文件名 image3D = reader.Execute()#读取dicom序列
image3D是一个三维矩阵,也就是说是一个三维的立体的数据,可以像读取一张切片一样,读取它的原点,像素间隔,方向,等基本信息
但是元信息的读取,不能通过image3D本身,要通过reader
reader.GetMetaDataKeys(slice_index)
reader.GetMetaData(slice_index,key)
通过切片的索引来读取属于该切片的键,然后通过切片索引与键获取相应的值,这里的键值对与单张切片的形式一样