数据集背景介绍
- 2009年的《纽约市基准法律》要求对建筑的能源和水的使用信息进行说明和评分。 涵盖的建筑包括具有单个建筑物的总建筑面积超过50,000平方英尺(平方英尺),和群建筑面积超过100,000平方英尺。指标是由环境保护署的工具ENERGY STAR Portfolio Manager计算的,并且数据由建筑物所有者自行报告。(回归问题)
字段说明
- 目标数据:
- ENERGY STAR Score:指定建筑物类型的1到100百分位排名,在投资组合管理器中根据自我报告计算报告年度的能源使用情况。
数据导入
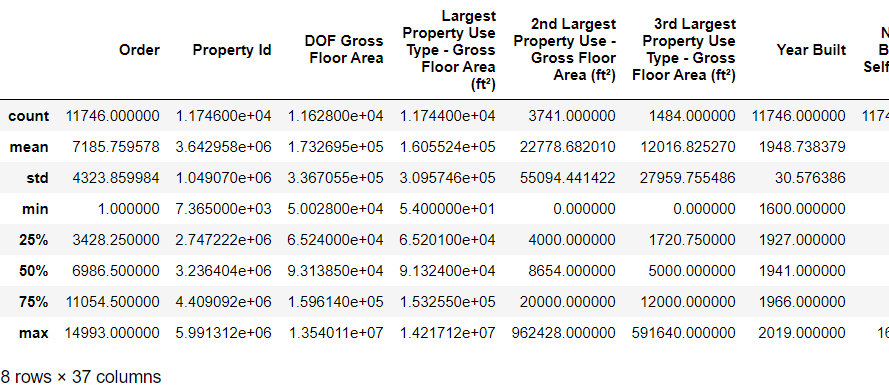
import pandas as pd import numpy as np import matplotlib.pyplot as plt %matplotlib inline import seaborn as sns from sklearn.model_selection import train_test_split data = pd.read_csv('data/Energy_and_Water_Data_Disclosure_for_Local_Law_84_2017__Data_for_Calendar_Year_2016_.csv') data.head(1)
查看数据形状
# 查看数据形状 data.shape # (11746, 60)
查看数据字段类型和是否存在缺失值
# 查看数据字段类型和是否存在缺失值 data.info()
数据集中的字段中存有Not Available值,该值表示无效值可以视为缺失值被处理
# 首先将"Not Available"替换为 np.nan data = data.replace({'Not Available': np.nan})
数据集有的字段显示为数值型数据,但是实际类型为str,再将部分数值型数据转换成float
# 数据集有的字段显示为数值型数据,但是实际类型为str,再将部分数值型数据转换成float for col in list(data.columns): if ('ft²' in col or 'kBtu' in col or 'Metric Tons CO2e' in col or 'kWh' in col or 'therms' in col or 'gal' in col or 'Score' in col): data[col] = data[col].astype(float)
通过 describe 和 matplotlib 可视化查看数据的相关统计量(柱状图)3
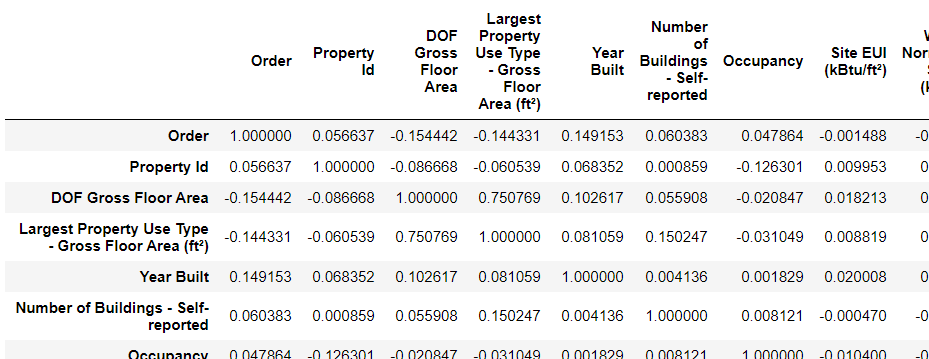
data.describe()
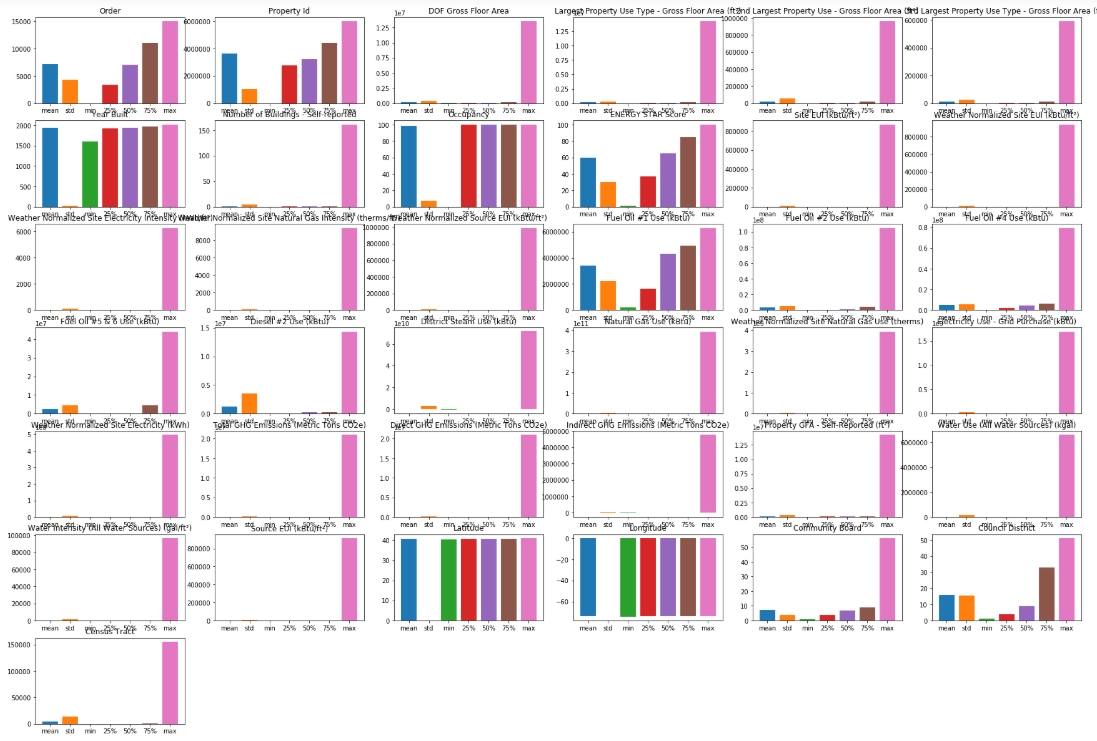
# 通过 describe 和 matplotlib 可视化查看数据的相关统计量(柱状图) data_desc = data.describe() # 查看数据描述 cols = data_desc.columns # 取得列缩影 index = data_desc.index[1:] # 去除count行 plt.figure(figsize=(30, 30)) # 控制画布大小 for i in range(len(cols)): ax = plt.subplot(10,6,i+1) # 绘制10x6的表格,当前数据特征维度为60 ax.set_title(cols[i]) # 设置标题 for j in range(len(index)): plt.bar(index[j], data_desc.loc[index[j], cols[i]]) # 对每个特征绘制describe柱状图 plt.show() # Order的图形比较正常,因为最小值,中位数,最大值是错落分布,正常分布的,且均值和标准差分布也正常 # DOF Gross Floor Area图形可能有问题,显示最大值比其他的值都大很多(离均点,异常值),如果最大值的数据数量较少,则考虑将其删除 # 发现:经度,维度特征的std极低,且数值分布特别均匀,说明这俩列特征对结果影响几乎为0,适当考虑过滤该特征
查看每列的缺失值比例
封装查看每列缺失值比例函数
查看每列的缺失值比例,估计大家有很多种方法可以做,可以给大家提供一个函数,聪明的同学把他当做自己的工具吧,以后你会常用到的
# 查看缺失值 def missing_values_table(df): # 计算每一列缺失值的个数 mis_val = df.isnull().sum(axis=0) # 计算每列缺失值占该列总数据的百分比 mis_val_percent = 100 * df.isnull().sum(axis=0) / data.shape[0] # 将每一列缺失值的数量和缺失值的百分比级联到一起,形成一个新的表格 mis_val_table = pd.concat([mis_val, mis_val_percent], axis=1) # 重新给上步表格的列命名 mis_val_table_ren_columns = mis_val_table.rename( columns = {0 : 'Missing Values', 1 : '% of Total Values'}) # 将百分比不为0的行数据根据百分比进行降序排序 mis_val_table_ren_columns = mis_val_table_ren_columns[ mis_val_table_ren_columns.iloc[:,1] != 0].sort_values( '% of Total Values', ascending=False).round(1) # 打印概述 print ("Your selected dataframe has " + str(df.shape[1]) + " columns. " "There are " + str(mis_val_table_ren_columns.shape[0]) + " columns that have missing values.") # Return the dataframe with missing information return mis_val_table_ren_columns
调用函数查看缺失值及比例
missing_df = missing_values_table(data); missing_df.head(3) Your selected dataframe has 60 columns. There are 46 columns that have missing values. # 下面缺失比例比较大
设置阈值将缺失比例超过百分之50的列删除
# 设置阈值将缺失比例超过百分之50的列删除 # 找出超过阈值的列 missing_df = missing_values_table(data); missing_columns = list(missing_df.loc[missing_df['% of Total Values'] > 50].index) print('We will remove %d columns.' % len(missing_columns)) data = data.drop(columns = list(missing_columns)) Your selected dataframe has 60 columns. There are 46 columns that have missing values. We will remove 11 columns.
中位数填充剩下的空值
# 中位数填充剩下的空值 np.median获取中位数,如果原始数据存在空值就会返回空nan for x in data.columns: # 去除object类型的列(object列不存在中位数) if str(data[x].dtypes) == 'object': continue if data[x].isnull().sum() > 0: # 取出每列非空元素求得中位数进行填充 data[x] = data[x].fillna(value=np.median(data.loc[~data[x].isnull(),x]))
查看目标数据的分布情况
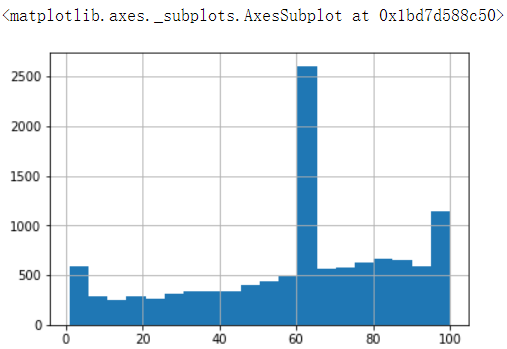
# 查看目标数据的分布情况 data['ENERGY STAR Score'].hist(bins=20)
plt.figure(figsize=(40,20)) plt.scatter(data['ENERGY STAR Score'].index,data['ENERGY STAR Score'].values) # 由图看到集中到60多出现一条线,说明数据集中在60多,没有找到离群数据
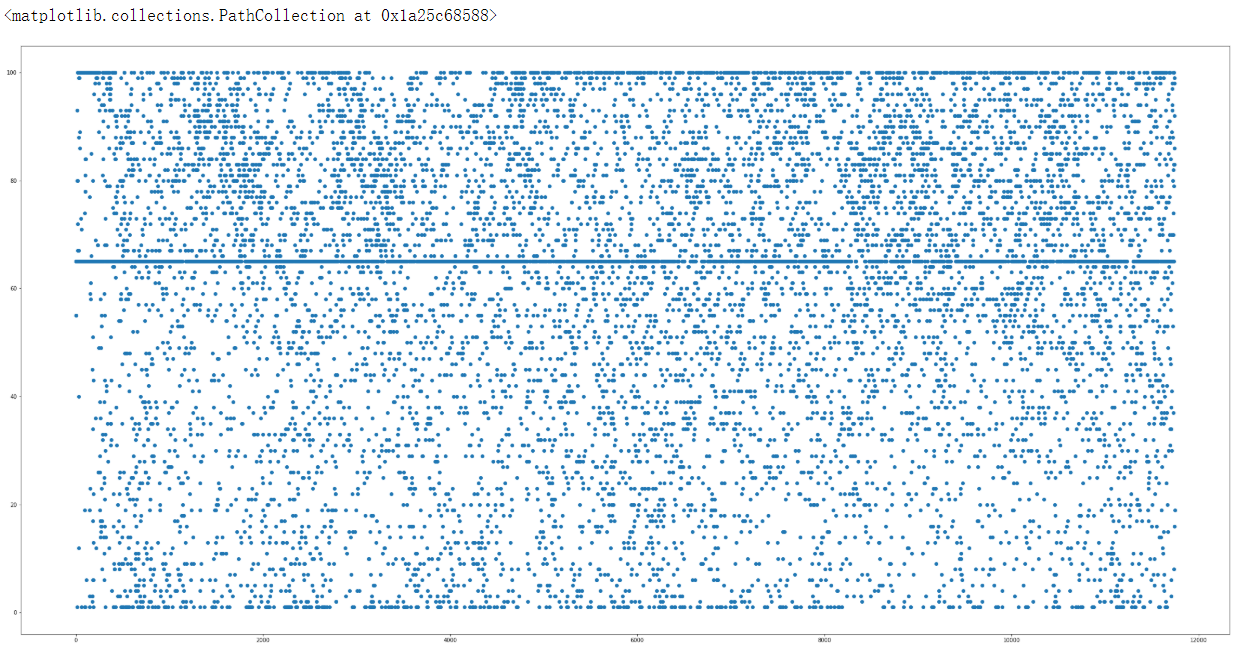
data['ENERGY STAR Score'].value_counts().sort_values().tail(1) 65.0 2216 Name: ENERGY STAR Score, dtype: int64
发现分布在65的数据量是最多的,没有发现离群数据
什么是离群点数据?
存在着一些特别大或者特别小的值,这些可能是离群点或记录错误点,对我们结果会有一些影响的。那我们是需要将离群点数据进行过滤的。
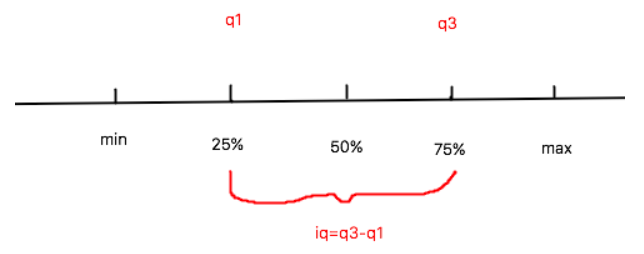
- 离群点:离群点是指一个数据序列中,远离序列的一般水平的极端大值和极端小值,且这些值会对整个数据的分析产生异常的影响。
- 传统的过滤方式:
- Q1 - 3 * IQ:Q1为序列中25%的中位数,IQ为Q3-Q1
- Q3 + 3 * IQ:Q3为序列中75%的中位数,IQ为Q3-Q1
- 离群点判定:
- 极小的离群点数据:x < (q1 - 3 * iq)
- 极大的离群点数据:x > (q3 + 3 * iq)
假设我们的目标数据为Site EUI (kBtu/ft²)
寻找离群点
data['Site EUI (kBtu/ft²)'].hist(bins=20)
plt.figure(figsize=(15,8)) plt.scatter(data['Site EUI (kBtu/ft²)'].index,data['Site EUI (kBtu/ft²)'].values)
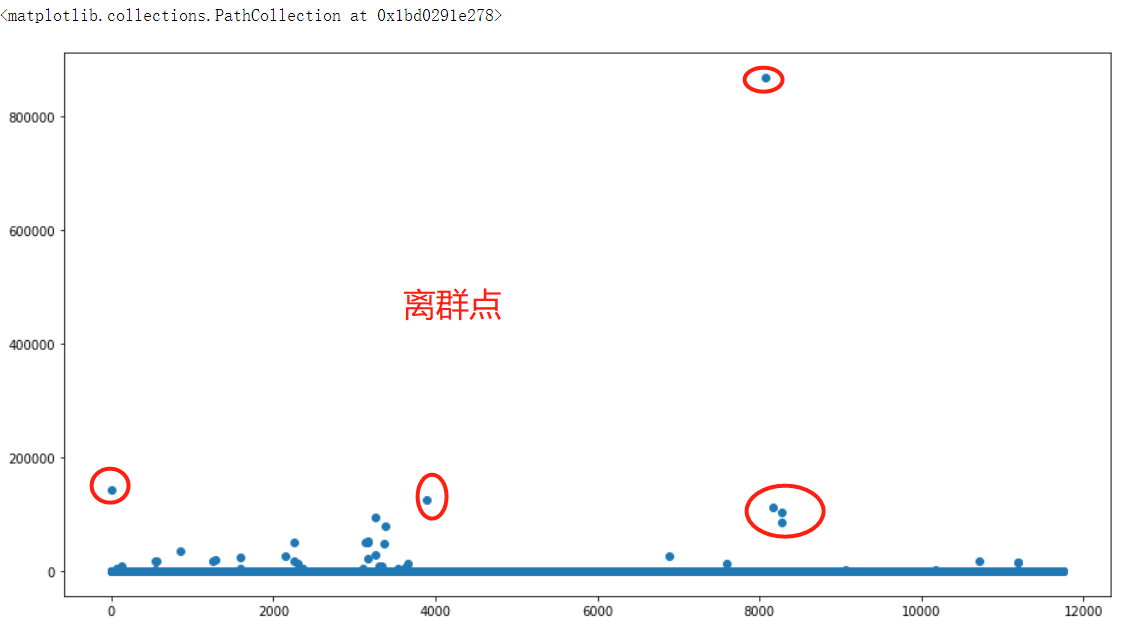
离群点数据过滤
# 离群点数据过滤 q1 = data['Site EUI (kBtu/ft²)'].describe()['25%'] q3 = data['Site EUI (kBtu/ft²)'].describe()['75%'] iq = q3 - q1 # data_copy就是离群的数据 data_copy = data[(data['Site EUI (kBtu/ft²)'] > (q1 - 3 * iq)) & (data['Site EUI (kBtu/ft²)'] < (q3 + 3 * iq))] # 之后我们就可以对离群点做处理,替换replace还是删除 data_copy['Site EUI (kBtu/ft²)'].hist(bins=30)
plt.scatter(data_copy['Site EUI (kBtu/ft²)'].index,data_copy['Site EUI (kBtu/ft²)'].values)
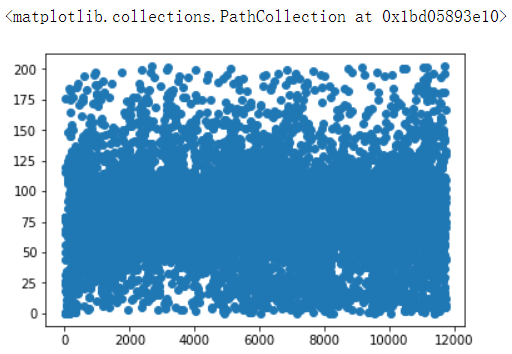
特征数据的分布
- 可以观测到特征的取值范围
- 可以观测到特征不同数值的分布的密度
- 可以观测到特征是连续性还是离散型
查看特征的不同取值数量
# 查看特征的不同取值数量 for x in data.columns: print('*'*50) print(x,data[x].nunique()) # 取值少的可能为类别性数据,取值多的为连续性数据
图形查看所有特征数据分布
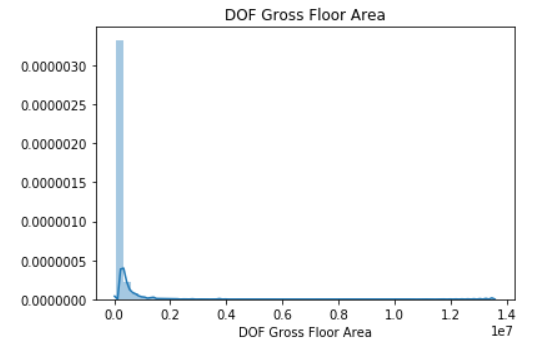
for col in data.columns: if 'int' in str(data[col].dtypes) or 'float' in str(data[col].dtypes): # plt.hist(data[col],bins=50) sns.distplot(data.loc[~data[col].isnull(),col]) plt.title(col) plt.show() # 发现有很多特征都是长尾分布的,需要将其转换为正太或者近正太分布,长尾分布说明特征中少数的数值是离群点数据
长尾分布说明特征中少数的数值是离群点数据,需要将其转换为正太或者近正太分布了,log操作转换为近正太分布
log操作转换为近正太分布
# log操作转换为近正太分布 data['DOF Gross Floor Area'] sns.distplot(np.log(data.loc[~data['DOF Gross Floor Area'].isnull(),'DOF Gross Floor Area']))
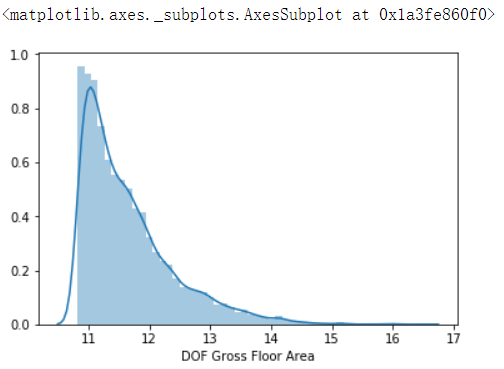
直方图查看数据分布
# 直方图 for col in data.columns: if 'int' in str(data[col].dtypes) or 'float' in str(data[col].dtypes): plt.hist(data[col],bins=50) plt.title(col) plt.show()
类别型特征的特征值化
- 结合着项目的目的和对非数值型字段特征的理解等手段,我们只选取出两个代表性特征Borough和Largest Property Use Type对其进行onehot编码实现特征值化
features = data.loc[:,data.columns != 'ENERGY STAR Score']# 提取特征数据 fea_name = features.select_dtypes('number').columns # 提取数值型特征名称 features = features[fea_name] # 提取数值型特征 # 提取指定的两个类别型特征 categorical_subset = data[['Borough', 'Largest Property Use Type']] categorical_subset = pd.get_dummies(categorical_subset) features = pd.concat([features, categorical_subset], axis = 1)
探索特征之间的相关性
corr查看特征与特征的相关性
features.corr() # corr查看特征与特征的相关性
plt.subplots(figsize=(30,15)) #指定窗口尺寸(单位英尺) features_corr=features.corr().abs() # 返回列与列之间的相关系数 abs求得是绝对值,相关系数与正负无关 # 数据为相关系数,显示数值,显示颜色条 这里需要导入模快 import seaborn as sns 也是一个绘图模块 sns.heatmap(features_corr, annot=True)
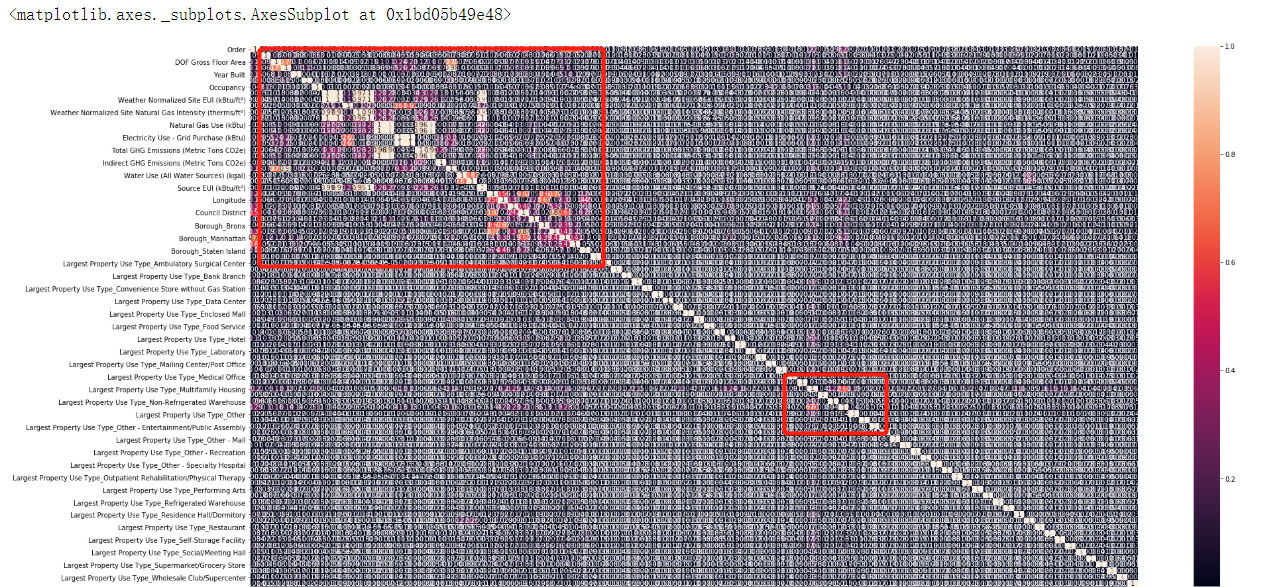
去除相关性强的冗余特征,工具包封装如下
cols = features.columns # 获取列的名称 corr_list = [] size = features.shape[1] high_corr_fea = [] # 存储相关系数大于0.5的特征名称 for i in range(0,size): for j in range(i+1, size): if(abs(features_corr.iloc[i,j])>= 0.5): corr_list.append([features_corr.iloc[i,j], i, j]) # features_corr.iloc[i,j]:按位置选取数据 sorted_corr_list = sorted(corr_list, key=lambda xx:-abs(xx[0])) # print(sorted_corr_list) for v,i,j in sorted_corr_list: high_corr_fea.append(cols[i]) print("%s and %s = %.2f" % (cols[i], cols[j],v)) # cols: 列名
删除相关性强的特征
# 删除特征 features.drop(labels=high_corr_fea,axis=1,inplace=True) features.shape # (11746, 69)
查看特征和目标之间的相关性
- 如果特征和标签之间是存在线性关系的才可以采用如下方式
target = data['ENERGY STAR Score'] target = pd.DataFrame(data=target,columns=['ENERGY STAR Score']) # 级联target&features new_data = pd.concat((features,target),axis=1) # 计算相关性,之后我们就可以选择特征与目标相关性较大的数据进行特征的选取 fea_target_corr = abs(new_data.corr()['ENERGY STAR Score'][:-1]) fea_target_corr
保存数据
# 改名字 new_data = new_data.rename(columns = {'ENERGY STAR Score': 'score'}) new_data.to_csv('./data/eda_data.csv')
建模 (该案例是回归问题)
数据集导入及切分
# Pandas and numpy for data manipulation import pandas as pd import numpy as np import matplotlib.pyplot as plt %matplotlib inline import seaborn as sns from sklearn.preprocessing import MinMaxScaler from sklearn.linear_model import LinearRegression from sklearn.ensemble import RandomForestRegressor from sklearn.svm import SVR from sklearn.neighbors import KNeighborsRegressor from lightgbm import LGBMRegressor # Hyperparameter tuning from sklearn.model_selection import GridSearchCV,train_test_split data = pd.read_csv('./data/eda_data.csv').drop(labels='Unnamed: 0',axis=1) fea_name = [x for x in data.columns if x not in ['score']] feature = data[fea_name] target = data['score'] x_train,x_test,y_train,y_test = train_test_split(feature,target,test_size=0.2,random_state=2020)
选择的机器学习算法(回归问题)
- Linear Regression
- Support Vector Machine Regression
- Random Forest Regression
- lightGBM (忽略)
- xgboost
- 我们只选择其默认的参数,这里先不进行调参工作,后续再来调参。
- 由于模型的调用和评价方式是一样的,则封装如下工具函数
# Function to calculate mean absolute error def mae(y_true, y_pred): return np.mean(abs(y_true - y_pred)) # Takes in a model, trains the model, and evaluates the model on the test set def fit_and_evaluate(model): # Train the model model.fit(x_train, y_train) # Make predictions and evalute model_pred = model.predict(x_test) model_mae = mae(y_test, model_pred) # Return the performance metric return model_mae
尝试各种模型
# 线性回归 lr = LinearRegression() lr_mae = fit_and_evaluate(lr) print('Linear Regression Performance on the test set: MAE = %0.4f' % lr_mae) Linear Regression Performance on the test set: MAE = 20.8585 # svm支向量机 svm = SVR(C = 1000, gamma = 0.1) svm_mae = fit_and_evaluate(svm) print('Support Vector Machine Regression Performance on the test set: MAE = %0.4f' % svm_mae) Support Vector Machine Regression Performance on the test set: MAE = 21.3965 # 随机森林 random_forest = RandomForestRegressor(random_state=60) random_forest_mae = fit_and_evaluate(random_forest) print('Random Forest Regression Performance on the test set: MAE = %0.4f' % random_forest_mae) Random Forest Regression Performance on the test set: MAE = 11.7919
条形图显示mae
plt.style.use('fivethirtyeight') model_comparison = pd.DataFrame({'model': ['Linear Regression', 'Support Vector Machine', 'Random Forest', ], 'mae': [lr_mae, svm_mae, random_forest_mae,]}) model_comparison.sort_values('mae', ascending = False).plot(x = 'model', y = 'mae', kind = 'barh', color = 'red', edgecolor = 'black') plt.ylabel(''); plt.yticks(size = 14); plt.xlabel('Mean Absolute Error'); plt.xticks(size = 14) plt.title('Model Comparison on Test MAE', size = 20);
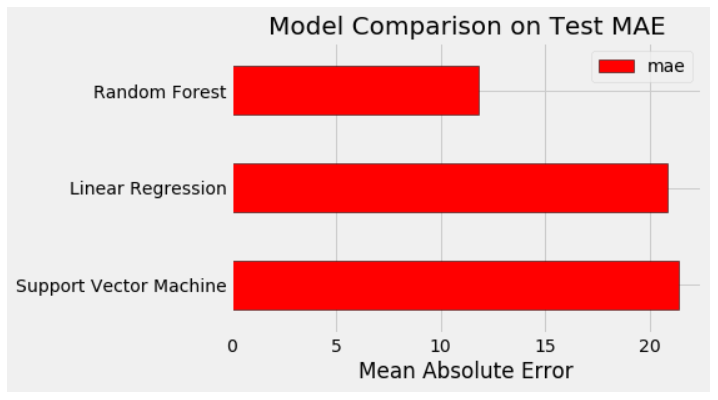
看起来集成算法更占优势一些,这里存在一些不公平,因为参数只用了默认,但是对于SVM来说参数可能影响会更大一些。
模型调参 (这里用网格搜索找寻最佳参数)
- 以随机森林参数调优为例
from sklearn.model_selection import GridSearchCV parameters = { 'n_estimators':[150,200,250] ,'min_samples_split':[2, 3, 5, 10, 15] ,"max_depth":[2, 3, 5, 10, 15] ,'min_samples_leaf':[1, 2, 4, 6, 8] ,'min_samples_split':[2, 4, 6, 10] } model = RandomForestRegressor() GS = GridSearchCV(estimator=model,param_grid=parameters,cv=5,scoring='neg_mean_absolute_error') GS.fit(x_train,y_train) GS.best_params_