共表达网络揭示组织特异性转录调控和剪接
背景
基因共表达网络在基因表达数据中可以捕获生物学上重要的模式,使基因的功能分析,生物标记的发现和遗传变异的解释成为可能。目前大多数的网络分析局限在评估单个组织或少量组织中总体基因表达水平之间的相关性。最近在Genome Research发表的这篇文章通过建立了网络,捕获相对isoform丰度和剪接的调控,以及每一种不同的组织独特的组织特异性关系。
数据
从Genotype Tissue Expression (GTEx)获得50个组织的449个捐献者的RNA-seq数据
结果
1. 在人体组织上重建全转录组网络(TWN)
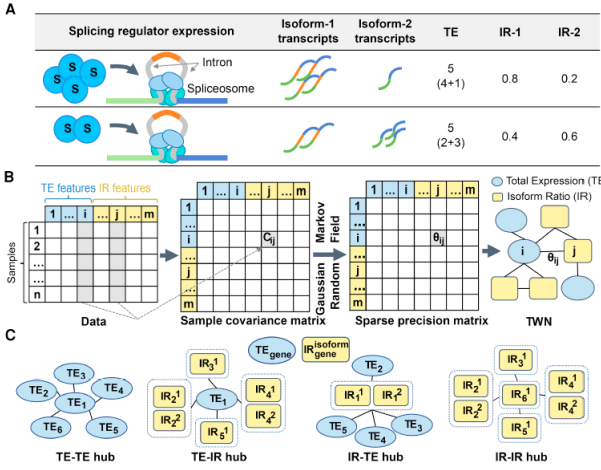
要构建一个TWN,首先对每个样本的每个基因的总体表达水平(TEs)和isoform表达水平都进行量化,然后计算isoform比率(IRs),来代表相对丰度。IRs和TEs都作为网络中的节点。转录调控影响基因的总体表达水平,剪接调控主要影响isoform比率。使用graghical lasso方法估计边的权重,并改进这个方法对不用类型节点之间赋予不同的权重。文章对至少有200个捐献者的16种组织构建了TWN。网络中包含四种类型的hub节点。
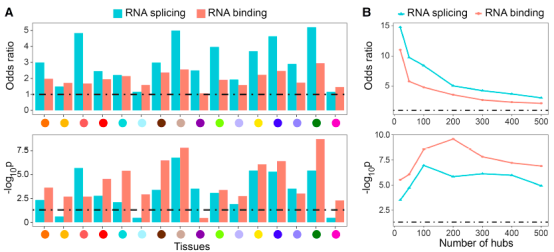
2. TWN中的hub节点富集剪接调控子
对于每种组织,评价了top TE-IR hubs在GO term中的富集,发现它们在RNA剪接基因和RNA结合基因中的显著富集。并且富集对度阈值的选取是鲁棒的。此外,在多组织共享的TE-IR hubs中还观察到RNA剪接和RNA结合的更强的富集。
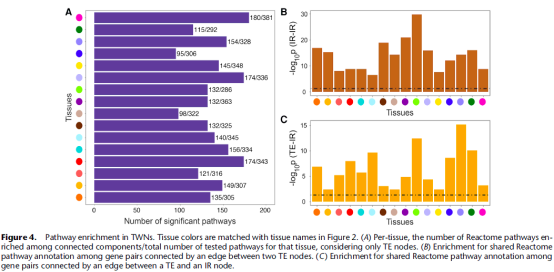
3. 表达和isoform比率的共调控反映生物学通路
在TWN中,观察到了转录因子和已知靶基因之间的边的富集,还观察到紧密连接的基因在Reactome和KEGG通路中更强的富集。
此外,作者还评价了每个TWN中功能相关基因的边的富集情况。发现IR-IR边,TE-IR边,TE-TE边都倾向于参与同一通路。因此,TWN可以用来预测基因功能,基于共调控,包括剪接调控。
4. TWN之间的比较揭示每个组织的hub基因
通过Kendall秩相关的计算,发现TWN的所有hub类型在大多数组织间具有较高水平的相似性。功能相关的组织间有着更高水平的相似性。
为了识别出组织特异的调控基因,作者评价了在相关组织中rank较高而在不相关组织间rank较低的TE-IR hub。发现了几个最高rank的组织特异的hub是已知的功能或相关的基因。
作者评估了确定的hub基因的组织特异性。为了做到这一点,计算了每个组织中没有出现在任何其他组织中的前100个TWN hub。发现了有8%-43%的TE-TE,11%-39%的TE-IR,0%-24%的IR-TE,0%-20%的IR-IR是组织特异的。
5. 组织特异性网络识别组织特有的基因共表达模式
作者为了区分共享的和组织特异的基因共表达关系,使用了BicMix,一种Bayesian二分网络框架,重构了组织特异的网络(TSN)。构建了26个GTEx组织的TSN。TSN中只包含了TWN中少量的边,代表了组织特异的共表达成分。
6. TSN的功能分析
对每个TSN中的基因进行GO富集分析发现26个TSN中的21个显著富集在了组织相关的GO生物学过程中。此外,作者还分析了每个TSN的hub基因,发现它们没有富集在跨组织转录因子或是组织特异性转录因子。这可能是由于转录因子不是组织特异的。此外,作者还对TSN进行了通路富集分析。
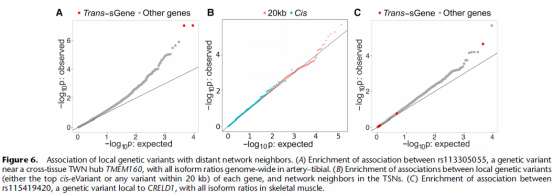
7. 整合网络与调控遗传变异
之前的TWN和TSN只用到了基因表达数据,在这一部分作者将GTEx数据中的基因型信息整合进网络。将网络中的边与eQTL结合,以此增强检测影响基因表达的反式遗传变异的能力。在每个网络中都观察到了遗传变异和基因以及基因的邻居节点之间显著的富集。
总结
本文的结果提供了迄今为止最大的一组组织中最全面的基因调控,剪接和共表达的图谱。这些网络将为解释复杂疾病中遗传变异,差异表达和剪接的全转录组范围的影响以及各种调控基因在人体组织中的影响提供基础。
参考文献
Saha A, Kim Y, Adh G, et al. Co-expression networks reveal the tissue-specific regulation of transcription and splicing[J]. Genome Research, 2016.