-
Illumina的SNP芯片原理
Illumina的SNP生物芯片的优势在于:
第1,它的检测通量很大,一次可以检测几十万到几百万个SNP位点
第2,它的检测准确性很高,它的准确性可以达到99.9%以上
第3,它的检测的费用相对低廉,大约一个90万位点的芯片(每个样本的)检测费用在一、两千人民币
Illumina的生物芯片系统,主要是由:芯片、扫描仪、和分析软件组成。
Illumina的生物芯片,由2部分组成:第1是玻璃基片,第2是微珠。

这个玻璃基片,它的大小和一张普通的载玻片差不多大小,它起到的作用,就是给微珠做容器。
在这个玻璃基片上,通过光蚀刻的方法,蚀刻出许多个排列整齐的小孔。每个小孔的尺寸都在微米级,这些小孔是未来容纳微珠的地方。小孔的大小与微珠正好相匹配,一个小孔正好容纳一个微珠。
微珠是芯片的核心部分,微珠的体积很小,只有微米级。
每个微珠的表面,都各偶联了一种序列的DNA片段。每个微珠上,有几十万个片段,而一个珠子上的片段,都是同一种序列。
这些DNA片段的长度是73个碱基,而这73个碱基又分成2个功能区域。

靠近珠子的这一端的23个碱基的序列,被称为Address序列,它也是DNA片段的5'端。它是标识微珠的标签序列。标签序列,通过碱基的排列组合,得到许多可能,每种序列,就是相应微珠的身份证号码(ID号)。
DNA片段上离珠子远的那一端的50个碱基,也就是3'端的序列,被称作Probe序列,它的作用,是与目标DNA进行互补杂交。
一种Address序列,就对应了一种probe序列。它们之间有着一一对应的关系。

在Illumina生产芯片的过程当中,是把要做芯片的几十万种微珠,按设定的比例进行混合好,撒到玻璃基片上。微珠随机地落入基片的小孔当中,然后,通过检测芯片上每个小孔当中的微珠上的Address序列,就可以知道,这个小孔当中是哪种微珠。 -
又因为Address序列和Probe序列有着一一对应的关系,这样,也就知道了每个小孔当中,有哪种Probe。反过来说,也就知道了每种Probe分布在哪几个小孔中了。
所以,Illumina公司出厂的每一张芯片,都要跟一个“.dmap”文件。这个.dmap文件,标注了每一张芯片上,每一个微孔当中,分别是哪种微珠。
用户做完芯片实验,得到扫描数据后,要从Illumina的网站上下载这张芯片的对应dmap文件,然后才能解读这张芯片。
在一张芯片的一个反应(样本位)当中,每种珠子平均有约15颗或更多。
说完了Address序列的功能,接着我们来说Probe序列的功能。
Illumina的生物芯片扫描仪,是扫描2种(荧光)颜色的:红色和绿色。而碱基有4种:A、C、G、T,要用2种荧光颜色,在一次实验当中,就区分出四种碱基,就需要一些巧妙的设计。
在Illumina的SNP芯片Probe设计上,先把要检测的位点,分成2种情况。
第一种情况是比较简单的,我们先举例来说明。如果一个SNP位点的野生型是“G”,突变型是“A”,那么就设计一个探针。这个探针的3'端的最末一个碱基,就挨着这个SNP位点。在
实验过程当中,目标片段通过互补杂交,结合到这个探针上,然后,加入四种带标记的双脱氧核苷酸。其中A、T两种核苷酸是用DNP(二硝基苯)来进行标记的。C、G两种碱基是用生物素来标记的。同时,加入聚合酶,聚合酶就会在探针的3’末端,加上一个双脱氧核苷酸,并同时捎带连上一个标记物。


接着加入绿色荧光标记的链霉亲合素,红色荧光标记的抗DNP的抗体。绿色荧光标记的链霉亲合素与生物素特异地结合,让带生物素的C、G碱基显出绿色。红色荧光标记的抗DNP的抗体与DNP结合,让带DNP的A、T碱基显出红色。


并且进一步加入生物素标记的抗链霉亲合素的抗体、和DNP标记的,抗异种抗体FC端的抗体。加入这两种抗体的作用,是使荧光信号得到进一步的级联放大。
抗体结合完了之后,经过清洗,把游离的抗体都给洗掉。在扫描仪下进行扫描。
如果发出的光是绿光,就说明这个SNP结合的位点,是个“G”碱基的纯合子。如果发出的是红光,就说明这个SNP位点是个“A”碱基的纯合子。如果既有红光、又有绿光,而且两种颜色的光的光强差不多,就说明这个SNP位点是一个“A”和“G”的杂合子。
说明了上面的道理,那么,A-C、A-G、T-C、T-G,这四种SNP情况都可以理解了。因为它们长出来的碱基,最后都会被染成不同的颜色,所以,可以被轻松地区分。

那么,接下来,你就会想,对于A:T,或者C:G型的SNP位点,该如何来区分。因为“A:T”会有同样的红色荧光,“C:G”也会有同样的绿色荧光。
好,接着我们就来说,这第二种情况的SNP位点的区分方案。
刚才我们说了,第一种SNP位点的区分方案,是把探针设计到紧挨着SNP位点,但留出SNP位点,让下一个延长的碱基,按照互补原则,根据SNP位点的碱基来生长。
那么,这第二种情况的SNP,在设计探针的时候,最后一个碱基,是盖在SNP位点上的。而且是设计2种探针,如果SNP位点是“A”和“T”,那么探针也设计“A”和“T”。并且分别盖在SNP位点上面。
这2种探针,在与目标DNA片段结合的时候,如果最后一个碱基是互补的,那么接下来的延伸反应就会发生,新的带标签的双脱氧核苷酸就会被加到探针链上。再接下来,就会被荧光抗体染色,在激光扫描的过程当中,就会发光。
反之,如果最后一个碱基是不互补的,那么接下来的延伸反应,就不会发生。当然,也就不会有标签加到探针链上,再接下来,荧光抗体也就不会将之染色。在后面的激光扫描当中,就不会发光。

激光扫描的结果,如果末尾是A碱基的探针发光,而末尾是T碱基的探针不发光,那么说明目标SNP位点上是一个“T”的纯合子;反之,则是“A”的纯合子;如果A和T的探针都发光,而且发光强度差不多,那说明SNP位点上是一个“A”和“T”的杂合子。
理解了Illumina的SNP芯片的工作原理,也就理解了它为什么准确率比较高。因为它是通过“红”或“绿”,和“有”或“无”,来区分一个SNP位点到底是哪种碱基的。
2. Affymetrix芯片原理今天,会和大家谈一下 Affymetrix 公司的生物芯片.
Affymetrix 公司是著名的生物芯片公司,它的芯片当中包含了:RNA表达量分析(表达谱芯片)、SNP检测(基因分型)、拷贝数变异(Copy Number Variation,CNV)、small RNA、甲基化等多种芯片。
今天我们会和大家介绍,它应用得最广的两种芯片:表达谱芯片、和SNP分型芯片。
首先,我们介绍一下 Affymetrix 的(生物芯片的)仪器,目前在售的仪器,主要有4个机型,从小到大,分别是:
1. GeneAtlas
2. GeneChipScanner 3000 7G(简称:7G)
3. GeneChipSystem 3000 DX2(简称:DX2)
4. GeneTitan
GeneAtlas 是一个小型系统,它主要可以扫描4张芯片一组的小芯片条。它的特点是:经济、易用。
GeneChip Scanner 3000 7G(7G)和GeneChipSystem 3000 DX2(DX2)。是一款机器的2个版本,其中,“7G”版是科研型版本、 “DX2”是临床版本。其中,DX2已经取得了美国FDA和中国CFDA的认证。
GeneTitan 是新机型,它的通量更大,自动化程度更高。平均到每个样本的检测成本更低。
GeneTitan在生物样本库项目(BioBank)当中,应用很多介绍完仪器,接下来,我们来介绍Affymetrix 芯片的制造过程。
制造原理
Affymetrix 芯片的制造过程,类似半导体芯片的制造过程。是通过光蚀刻来完成的。



它(生物芯片)的基片,是一张大的玻璃片,称为:“Wafer”。
首先,在玻璃基片上加上保护基团,也就是玻璃板上的这些蓝色小帽子。这些保护基团,它可以阻止接下来的DNA的延长反应。同时,这些保护基团是对光敏感的。
一旦受到紫外光的照射,这些保护基团就会从所连着的羟基上掉下来。把羟基给暴露出来。
接下来,进行光刻。我们以玻璃基片上3*2的6个小格子,这样一个小区域来说明光刻的过程。
先用一个光罩(mask 1)来遮住一部分的玻璃板区域,在光罩上,是一系列排列整齐的小方格。
有些小方格是透明的,还有一些小方格是不透明的,可以挡住光。

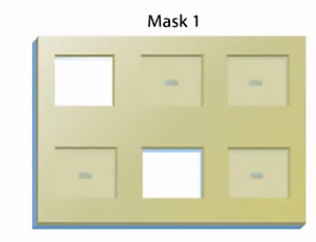
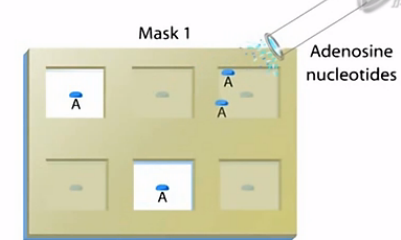

紫外光透过光罩(Mask 1),照到玻璃基片上。这些透明的格子所对应的地方,保护基团被紫外光照射到。
光敏的保护基团就从原来所连的羟基上掉下来,而那些不透明格子所对应的地方,因为没有被光照到,保护基团依然连接在原来的羟基上。
接下来,把要连的碱基底物加到玻璃基片上,这里第一个要加的碱基是“A”碱基,玻璃基片上刚才被光照射过的,去掉了保护基团的地方。
就会与新的A碱基结合,这样,A碱基就连接到玻璃基片上。
而刚才被光罩上不透明的区域所覆盖的,还留有保护基团的地方,就不会与新加入的“A”碱基进行结合。
请注意,这里新加进来的A碱基,也带着一个保护基团。
接着,进行第2轮的光刻,也就是拿第二张光罩盖Mask 2,盖在玻璃基片上。
紫外光再次透过光罩,照到玻璃基片上。但是请注意,这第二张光罩上的透明与不透明格子的分布。
是与第一张光罩不一样的,在第二次光照射之后,玻璃基片上对于应于第二个光罩透明的部分,上面的保护基团就掉了。
然后,我们把第二轮要种的T碱基铺到玻璃基片上,玻璃基片上,那些在第二轮光照射当中,去掉了保护基团的位置,就会长出一个T碱基。
再接着,用Mask 3进行遮盖,进行第3轮的光照射,然后,加上C碱基。
这样,不断地重复这个过程,玻璃基片上不同的位置,就会按原来的设计意图,长出我们想要的DNA链来。

这些DNA链,就是探针,这些探针,会在后面的实验当中,与目标DNA链或者RNA链,进行杂交、结合。
Affymetrix 公司芯片上的的探针,都是3’端连到玻璃基片上的。
Affymetrix 的芯片当中,做表达谱的芯片,也就是测RNA表达量的芯片,探针的长度是25个碱基,而做SNP分型的芯片,探针的长度是30个碱基。
Affymetrix 芯片上,长有相同序列DNA链的一个小点,被称为一个“Feature”。这也就是未来芯片扫描图上的一个光点,一张芯片上最多可以有680万个Feature。



每个Feature上会有几百万条相同序列的DNA探针。
这样一张大的玻璃基片,在种好所有的DNA链之后,就被裁切成一小片一小片的玻璃片,每张小玻璃片上都有一套完整的探针。每一张小的玻璃片,加上了辅助液流的外壳,再打上相应的标识,就成了一张生物芯片。
Affymetrix 公司的芯片,它所有设计的探针,都在确定的位置。在最后的芯片判读过程当中,
也是通过光点的空间位置,来知道测到的是哪个探针。
RNA芯片实验原理
接下来,我们介绍芯片的实验原理。我们先来说,表达谱的实验原理。
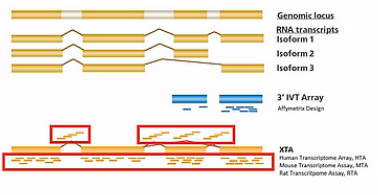
Affymetrix 的表达芯片,分成传统的In Vitro Transcription 芯片,也就是 IVT 芯片(In Vitro Transcription 的缩写),和新一代的 Whole Transcriptome 芯片,也就是 WT 芯片 (Whole Transcriptome 的缩写)。
其中 IVT 芯片是用 Oligo dT 引物和T7 逆转录酶来得到 cDNA 链的,所以,它得到的cDNA主要是靠近mRNA 3’位末端的cDNA。相应地,它的探针也主要针对每个基因的最后一、二个外显子来进行设计。
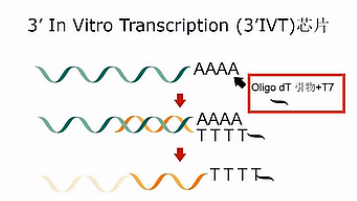
比较著名IVT类的芯片,有经典的 U133芯片,和较为经济的 PrimeView 芯片。
而 WT 芯片是用随机引物和 T7 逆转录酶来得到 cDNA 的,所以,它得到的 cDNA 会覆盖转录本上更多的区域,相应地,它的探针也是针对基因的整个转录本来进行设计的。
WT芯片的好处:
一、是它可以覆盖转录本上更多的区域,实验结果的代表性就会更强。
二、是它可以针对因为差异剪接所形成的不同转录本,分别设计探针,这样,就可以知道不同的转录本的表达量的变化了。
三、是它可以检测到长链非编码RNA(Long Non-CodingRNA, LncRNA)
比较著名的WT芯片有 HTA 2.0、Exon 1.0、Gene 2.0/2.1 等。
实验过程当中,先通过逆转录得到第一链的cDNA,紧接着就合成第二链的cDNA,变成双链cDNA之后,这个双链cDNA就可以作为接下来转录的模板了。
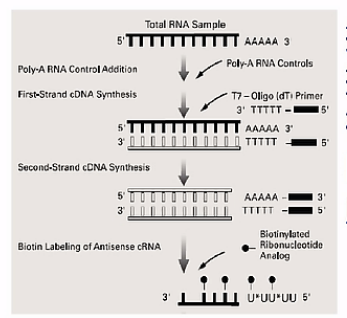

接下来,用掺有生物素标记的UTP的聚合反应底物,也就是ATP、CTP、TTP,再加上生物素标记的UTP,形成的4个单核苷酸的混合物,进行体外转录,转录得到cRNA (comple-mentaryRNA)。
因为转录的原料中含有被生物素标记的UTP,所以转录出来的cRNA片段就是带有生物素标签。
然后拿这些cRNA片段与芯片进行杂交,cRNA与芯片上的探针,依照碱基互补的原则进行杂交,杂交完了之后,用标记了藻红蛋白的链霉亲合素,也就是SAPE,对芯片进行染色(streptavidin-phycoerythrin ,SAPE)。
这其中,(SAPE上的)链霉亲合素会与cRNA上的生物素进行特异地结合;而(SAPE上的)藻红蛋白在激发光的照射下,可以发出红色荧光。
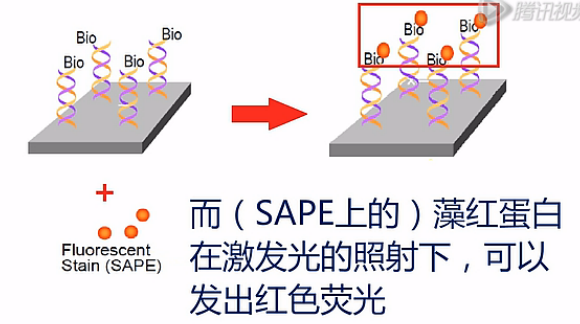

然后,再加入标记了生物素的抗链霉亲合素抗体,抗体就亲合吸附到那些已经吸附在cRNA上的链霉亲合素上。
亲合吸附完成之后,再加入SAPE。对芯片进行二次染色。
SAPE就吸附到抗体上的那些生物素上,通过上述的再次染色,可以把更多的藻红蛋白吸附到目标cRNA片段上,以增加荧光的强度。
化学反应完成之后,就可以把芯片拿到扫描仪上进行激光扫描了。
激光扫描之后,得到一张有着密密麻麻光点的图片,这张图片也就是荧光信号的矩阵,光点的X、Y轴的位置,也就是探针的ID号。
光点的(光)强度,也就对应着被杂交到的cRNA的量,而这个cRNA的量,就反映了对应基因特定mRNA转录本的表达量。

SNP分型芯片实验原理
说完了表达谱芯片,我们接下来说基因分型芯片,也就是SNP分型芯片。
Affymetrix 公司的SNP分型芯片有两种实验原理:新的是Axiom芯片,是基于连接反应的;而老的卡式芯片,是基于目标DNA片段与探针序列进行杂交。看序列是否完全配对。
我们先来说新的Axiom方法,Axiom方法中,有两种探针在起作用。


第一种探针是芯片上的捕获探针,它是30个碱基的长度。它起到的作用是把目标DNA片段,固定到芯片表面。
第二种探针是显色探针,它负责对SNP芯片进行显色。
我们先来看显色探针的设计,显示探针共分成四组,A、C、G、T各一组探针。它们都是9个碱基的长度,它们的3’末端的第一个碱基是特异的,而从第二个碱基到第9个碱基都是简并的。
这其中,3’端是C、或者G的,设计成5’端带一个生物素标签。也就是最后会被染成红色荧光。
而3’端是A、或者T的,5’端被设计成带另外一种标签,最后会被染色绿色荧光。
接下来,我们以一个“G:T”型的SNP位点为例,来进行说明。
在设计这个SNP位点的探针的时候,所设计的捕获探针,是正好到SNP位点旁边的一个碱基。
实验过程进行两轮杂交。
第一轮杂交,是目标DNA与芯片进行杂交,结果是芯片上的捕获探针会抓到相匹配的目标DNA片段,接着加入显色探针,进行第二轮杂交。
这一轮杂交,把显色探针,杂交到目标DNA片段上。
然后用连接酶进行连接,因为连接酶会对连接位点的前后几个碱基进行识别。
只有前后几个碱基都完全匹配,连接反应才会发生。
所以,利用连接酶的这种识别作用,让只有与目标DNA片段互补的显色探针,才会被连接酶连接到捕获探针上去。连接反应完成之后,把游离的显色探针都给洗掉。
再用带荧光标记的染色试剂进行染色,刚才连到捕获探针上的生物素标签,就在这个染色过程中被染上红色荧光(染料)。
反之,如果目标DNA片段上,这个位点是个“T”碱基,相应地,它的标签基团就会被染上绿色荧光基团。

染色完成之后,就可以用在激光扫描下对芯片进行扫描了,扫描过程当中,如果看到这个探针上所发出的光是单纯的红色,就可以判断这个位点的SNP型是“G”型纯合子。如果发出的荧光是单纯的绿光,那么就可以判断这个SNP位点是个“T”型纯合子,如果发出的光,既有红光,又有绿光;而且红光、和绿光的光强差不多,则可以判断这个SNP位点是个“G”和“T”的杂合子。
同样的道理,对于A:C、 A:G、 T:C、 T:G,这4种SNP情况,因为不同的基因型会发出不同颜色的荧光,所以只要看荧光的颜色、和荧光的光强,就可以分辨SNP型了。
那么对于"A:T"或者"C:G"型的SNP位点,就需要用不同的检测方案,因为 A / T 探针都是绿色荧光,而 C / G 探针都是红色荧光。
Affymetrix 对这2种SNP型,另外设计了相应的检测方案。我们拿“ A:T” 型的SNP来说明。
它针对“A”设计一个探针,再对“T”。设计一个探针。而这里设计的探针,它是盖到SNP位点上的。
请注意,这与之前第一种情况所设计的探针(颜色差异探针)不同,之前的探针是设计到SNP位点的旁边,而不是盖到SNP位点上。
这里,我们以一个5’端最后一个碱基为“A”的捕获探针为例,来看它上面所发生的化学反应。在经过第一轮的捕获杂交后,目标DNA片段与之发生杂交。
如果目标DNA片段的SNP位置上是一个“T”碱基,那么捕获探针与目标DNA片段完美匹配,接下来经过第二轮的杂交,一个显色探针杂交到它的旁边。


再经过连接反应,显色探针上的标签就连到了这个捕获探针上。反之,如果目标DNA片段上这个SNP位置上是一个“A”,那么它与捕获探针上的“A”是不匹配的。
那么在第二轮的杂交过程当中,虽然会有显色探针会停在它的旁边,但是,连接反应过程当中,因为连接酶要求严格的碱基匹配,所以连接反应不会发生。
停在它旁边的显色探针因为不能共价地连到捕获探针上。所以在接下来的洗脱过程当中,就会被洗掉。
这样,在激光扫描过程当中,如果这个探针上发出荧光,则说明对应的SNP位点上有“T”碱基。如果这个探针上不发出荧光,则说明对应的SNP位点上没有“T”碱基。
然后,再来看芯片上另一个对应的,5’端最后一个碱基为“T”的捕获探针:如果它发光,则说明SNP位点上有“A”;如果它不发光,则SNP位点上没有“A”。
把两个探针的发光情况综合来看,如果两个探针都发光,则说明这个SNP位点是一个“A”和“T”的杂合子。如果5’位末端是A碱基的探针有上荧光,而5’位末端是T碱基的探针上没有荧光,则可以判断这个SNP位点上是一个“T”的纯合子。反之,这个SNP位点是一个“A”的纯合子。
如果理解了A:T型SNP的区分原理。当然也就很容易理解C:G型SNP的区分原理了。
上述就是Axiom的检测原理,归纳一下:就是通过连接酶,对连接位点上碱基匹配的情况进行识别。只有碱基匹配,连接反应才可以发生。
如果碱基不匹配,则连接反应不能发生。
在Axiom的芯片当中,CHB1和CHB2是两款很常用的、针对中国人的SNP分型芯片。
它们有130万个SNP位点,而(Affymetrix公司的)卡式SNP芯片的原理,与Axiom的检测原理,是略有不同的。
卡式芯片不是以连接反应是否发生,作为检测的依据。
而是检测目标DNA片段,与捕获探针,之间的杂交结果。
在探针设计当中,对SNP的两种情况都设计相应的探针。
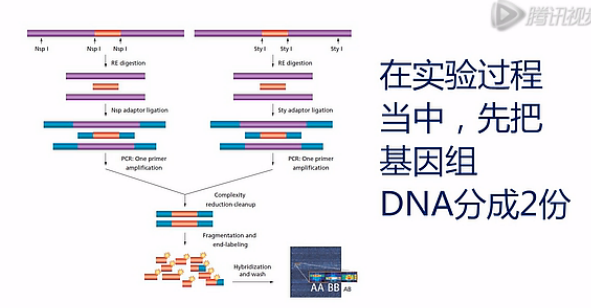
在实验过程当中,先把基因组DNA分成2份。一份用Nsp I酶进行消化;另一份用Sty I酶进行消化。
基因组DNA被消化成片段,之后,在两头连上接头。
进行PCR扩增,PCR扩增完了。之后,会得到长度主要分别在 200BP ~1100BP 之间的扩增片段。
然后再用酶把 PCR 扩出来的DNA片段进行(再次)片段化。片段化完了之后,所得到的,应该是平均长度小于 180BP片到的的片段。
接着用末端核苷酸转移酶(Terminal Deoxynucleotidyl Transferase),把带有生物素的单核苷酸,加到目标片段上。
然后,把这些带了生物素标签的目标DNA片段。与芯片进行杂交,再染色、扫描。
目标DNA片段与捕获探针杂交的,过程当中,遵循碱基互补原则。如果完全匹配,则杂交效率高。杂交到捕获探针上的目标片段就会多。反之,如果有一个碱基是不匹配的,那么杂交效率就会低许多,杂交到捕获探针上的目标片段,也就会少许多。
接下来,再经过染色,染色完了之后,进行激光扫描。
扫描过程当中,能发出荧光的探针,说明样本当中有对应基因形的DNA,如果探针不能发出荧光信号,或者发出的荧光信号很弱,则说明样本当中没有对应基因型的DNA。
如果一个SNP的两种荧光探针都发光,而且发光强度差不多,则说明样本在这个位点是一个杂合子。
以上就是卡式SNP芯片的检测原理,在卡式SNP芯片当中,“SNP 6.0”是一款很经典的芯片。
它上面有90多万个SNP位点的探针,并且同时还有94万个拷贝数变异探针。

软件
Affymetrix 分析表达谱的软件。
主要是用的 Transciptome under galtetede软件,简称TAC软件分析基因分型的软件,主要是用 Genotyping Console软件。
除了表达谱芯片、和基因分型芯片之外。
Affymetrix 公司还提供:microRNA芯片、基因调控芯片、拷贝数变异芯片
分子细胞遗传学芯片、药物遗传学芯片等,多种芯片。并且提供客户定制化服务。
3.Agilent生物芯片原理
今天,会和大家谈一下Agilent公司(安捷伦公司)的生物芯片。
Agilent的生物芯片(系统)和别的公司的生物芯片(系统)一样,同样由:扫描仪、生物芯片、分析软件,三部分组成。
Agilent的芯片扫描仪,叫SureScan DX。SureScan DX已经取得了欧洲的CE认证,和中国的CFDA认证,可以应用于临床。
生产工艺
接下来,我们介绍Agilent的芯片。首先,我们来看Agilent的芯片合成工艺。
Agilent芯片的基片是一个玻璃片。它的大小和一张标准的病理载玻片一样大小。
它的芯片制作过程,是用和喷墨打印一样的技术来进行制作的。喷墨打印机,是在墨盒里面是装了“红、黄、蓝、黑”四种颜色的墨水。而Agilent打印生物芯片的墨盒里面,是用带保护基团的A/C/G/T四种碱基底物,来代替了颜色墨水。
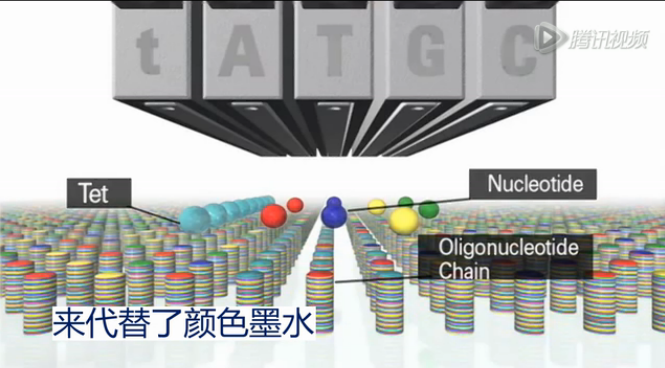
分别含有4种碱基底物的小液滴,被按照设计的探针序列,依次、层叠地喷到玻璃板的确定的位置上。
在每一个碱基的延伸过程当中都有3个步骤,分别是“脱保护基团、偶联、氧化”。

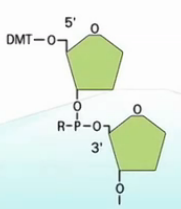
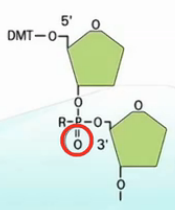

先把一个碱基,喷到玻璃板上,然后,再喷上第二个碱基,让两个碱基之间发生偶联。
接下来,进行氧化,把亚磷酸基团氧化成磷酸基团。
然后,把连在第二个碱基5’位羟基上的DMT保护基团给去掉。这样,留下一个自由的5‘位羟基。有了这个羟基,就可以进行下一步的延伸反应了。
不断重复这个过程,DNA链就会不断地延长。
Agilent的这个DNA链合成技术,每一步的合成效率都非常高,可以达到99%以上。这让Agilent可以在芯片上,得到很长的DNA链。最长,可以达到300个碱基的长度。

Agilent的这个方法得到的DNA链,它是3’端连到玻璃基片上的。
Agilent的这个基于打印原理的芯片合成技术,给予Agilent公司在制作不同序列的探针的时候,有着极大的灵活性。只要更换芯片探针的设计文件,就可以轻松地制作出一张全新序列的芯片来。因此,Agilent公司在接受客户定制化芯片的时候,可以接受少到“1张”芯片的定制化订单。
Agilent(目前)生产的芯片,可以根据点阵密度的不同,分成密度较低的HD芯片、和高密度的G3芯片。
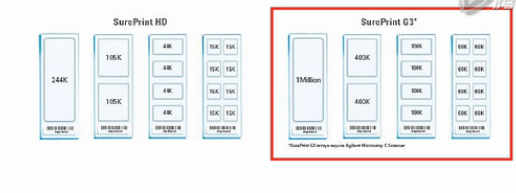
HD芯片,一张芯片上最多可以有24万4千个点,高密度的G3芯片,一张芯片上最多可以有1百万个点。
而一张芯片上,又可以根据点阵的分区的情况,区分成:1个区的、2个区的、4个区的、和8个区的。分的区越多,则一张芯片上,可以同时检测的样本数就越多。但是分区越多,每个样本可以检测的数据点就越少。
CGH芯片
说完了芯片制造的过程和大体规格,接下来,我们介绍Agilent芯片的应用。
我们先来说CGH芯片,也就是“Comparative GenomicHybridization”芯片。翻成中文,就是“比较基因组杂交”芯片。
CGH芯片,主要是检测:杂合性缺失(LOH)、单亲二染色体(UPD)、和拷贝数变异(CNV)。
先说一下,什么叫“杂合性缺失”。它的英文是“Loss Of Heterozygosity”,简称“LOH”。
正常情况下,常染色体上的一个区段,都会有来自于父亲、和母亲的各一个拷贝。
当发生杂合性缺失(LOH)的时候,两个染色体上的同一个区段,都是来自于或者父亲、或者母亲的一方,而把另一方的对应区段给丢失了。这就叫杂合性缺失(LOH)。


杂合性缺失是肿瘤发病的重要病因。
单亲二染色体,也就是“Uniparental Disomy”,简称“UPD”,是杂合性缺失的一种特殊形式。也就是一对染色体,都是来自于父亲、或者母亲中的一方,而把另一方的对应染色体,全部给缺失了。
这种变异的危害,和杂合性缺失的道理是一样的。只是因为它丢的是一整个染色体,所以致病的可能性会更高。
拷贝数变异,Copy Number Variation,简称“CNV”,是指一小段染色体片段的缺失,或者额外增加。

CGH芯片,主要就是检测这三种突变:“LOH”、“UPD”、和“CNV”。
在生物芯片检测方法出来之前,染色体变异,主要是通过核型分析来做的。但是核型分析的分辨率是较低的,大约只能看到10M以上片段的缺失、或者增加。对于小于10M的片段缺失、或者增加,则无法在核型分析中发现。


而用生物芯片的方法,则可以极大地提高检测上述突变的分辨率、和灵敏度。分辨率最高可以达到发现一个外显子的增加、或者缺失。
Agilent CGH生物芯片工作的原理,就是把样本的DNA片段化,标上红色荧光素“Cy5”;同时,再把来自几十个正常人的基因组DNA,混成一个标准DNA样本,取同样的DNA量,同样片段化,标上绿色荧素“Cy3”。

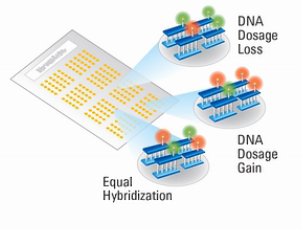

然后,把这两种标了荧光素的DNA片段,混合在一起,在同一张芯片上进行杂交。
接下来进行激光扫描,比较红光荧光与绿光荧光的光强。
所得到的光强比值,换算成以2为底的Log值。
如果在一个探针上,Log值接近于“0”,也就是说,红光与绿光的荧光光强差不多,那么,可以基本断定,在这个位置,样本中是有2个基因拷贝。
如果在一个点上,Log值大约等于“1”,也就是说红光的光强,是绿光的2倍,那么说明,样本在这个位置的拷贝数,可能是标准品的2倍。也就是说,样本在这个位置,可能有4个基因拷贝,比正常情况多出了2个拷贝。
同样道理,在一个点上,如果Log值小于等于“-2”,也就是说,红光的强度只有绿光强度的“1/4”,甚至更低,那么说明,样本在这个位置的2个拷贝,可能都丢失了。


因为来自一个点的荧光的光强变化,可能会带有一定的偶然性,所以,一般是看染色体空间位置上相邻的三个点(或者更多的点),如果这三个点的荧光比值,都发生同一个方向的偏离,就可以作为判断这一段有拷贝数变异的证据。
说完了拷贝数变异,我们进一步来看LOH和UPD的情况。因为LOH(杂合性缺失)和UPD(单亲二染色体),并不会改变某一区段的基因拷贝数,所以,就有必要加入SNP分析,来探测LOH和UPD。
如果染色体的一个区段内,同时有大量的杂合子存在,那么一般可以判断,这个区域没有发生LOH;反之,一个区段内,如果都是纯合子,那么,很可能这个区段内是发生了LOH(杂合性缺失)。
Agilent的CGH芯片,区分SNP位点的方法,是通过“酶切+杂交”。
把基因组DNA,用Alu I和Rsa I两种限制性内切酶进行消化。
我们以Alu I这种酶为例,它切的位点是“AGCT”。
如果基因组上的这个位点是CGCT的纯合子,那它就不会被酶切断。在后面杂交的过程当中,因为DNA链保持了完整的长链,所以与探针的吸附能力就强,最后,(在激光扫描中),就得到高强度的荧光信号。
如果基因组上这个位点,是CGCT和AGCT的杂合子,那么AGCT就会被切断,而CGCT保持完整。在后面与探针的杂交过程当中,保持完整长链的链,可以杂交到探针上去,而被切断链,它与探针杂交的序列就变短了,这样,它与探针的吸附力就减弱,在后面的洗脱过程当中,这个短链就会被洗掉。这样,2个等位基因链中,只那个没有被切断的长链会留在探针上,所以,最后的(激光扫描得到的)荧光强度,只有中等的强度。
如果基因组上这个位点是一个AGCT的纯合子,那它就会被酶完全切断。并且在杂交(洗脱)过程当中,它就会被洗脱掉,最后,探针上就没有荧光,或者荧光强度非常低。
这样,通过“酶切+杂交”,CGH芯片就可以分辨出基因组上的SNP位点,并且进一步判断是否有LOH或者UPD发生。
Agilent的最常卖的CGH芯片,是它的8*60K(SurePrint G3 Human CGH Microarray Kit,8x60K)、和4*180K的芯片(SurePrint G3Human CGH Microarray Kit, 4x180K)。
Agilent分析CGH芯片数据的软件,是CytoGenomics软件。
表达谱芯片
接下来,我们说Agilent的表达谱芯片。
Agilent表达谱芯片的检测原理,是IVT原理,也就是“In Vitro Transcription”原理。
首先,用带有T7启动子序列的Poly(T)引物,与mRNA的Poly(A)尾巴结合,(逆)转录出第一链的cDNA。然后,再转录出第二链的cDNA,这样,就得到了双链cDNA。
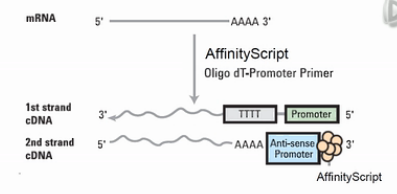

这个双链的cDNA是带有T7启动子的,接下来,就用这个T7启动子转录出cRNA来。cRNA是complementary RNA,也就是“互补RNA”。
在转录出cRNA的过程当中,所用的底物是特殊的。在4种碱基当中,C碱基不是天然的CTP,而是用标有Cy3荧光基团的合成CTP。这样,Cy3荧光基团就在体外转录过程当中,被带入到新合成的cRNA链当中去了。
接下来,把这个cRNA链与芯片进行杂交,杂交完了之后,在激光扫描仪下看每个点的荧光强度。根据每个探针点的荧光强度,反推出对应基因的RNA表达量来。
Agilent的表达谱芯片有以下几个特点:
第一,它的cRNA链上,标的直接就是Cy3荧光基团,而不是生物素,所以,它在与芯片杂交、洗脱完了之后,就可以直接上扫描仪进行检测。而不像别的用生物素进行标记的芯片平台,还要经过几步的荧光染色过程,才能进行激光扫描。所以Agilent的芯片处理过程,会比别的用生物素做标签的芯片平台更快。一般Agilent只要2天就可以完成样本处理、和芯片杂交过程,而别的芯片平台,可能会要3天时间。
第二,它的检测线性范围更大,可以达到10的5次方。相比之下,别的做表达谱的芯片平台的检测范围,一般只有10的3次方。也就是说,对于低表达的基因,安捷伦芯片的灵敏度更高。它可以检测到比别的平台低10倍的低表达量,对于高表达量的基因,Agilent芯片的检测量程更宽。它可以比别的平台,更准确地测到高10倍的高表达量。

第三,Agilent的表达谱芯片上的探针,是60个碱基。因为有了比别的芯片平台更长的探针,所以它的检测特异性会更好。
Agilent公司最常卖的人表达谱芯片,是:SurePrint G3 HumanGene Expression v3 8*60K MicroArray Kit。
安捷伦分析表达谱的软件,是GeneSpring软件。
Agilent公司除了提供:CGH芯片、和表达谱芯片之外,还提供:microRNA芯片、甲基化芯片、ChIP芯片、基因合成用的Oligo Library Synthesis芯片。有兴趣的同学,可以向Agilent公司咨询(www.agilent.com.cn)。