原文链接:http://tecdat.cn/?p=23061
原文出处:拓端数据部落公众号
数据集信息:
这个数据集可以追溯到1988年,由四个数据库组成。克利夫兰、匈牙利、瑞士和长滩。"目标 "字段是指病人是否有心脏病。它的数值为整数,0=无病,1=有病。
目标:
主要目的是预测给定的人是否有心脏病,借助于几个因素,如年龄、胆固醇水平、胸痛类型等。
我们在这个问题上使用的算法是:
-
二元逻辑回归
-
Naive Bayes算法
-
决策树
-
随机森林
数据集的描述:
该数据有303个观察值和14个变量。每个观察值都包含关于个人的以下信息。
-
年龄:- 个人的年龄,以年为单位
-
sex:- 性别(1=男性;0=女性)
-
cp - 胸痛类型(1=典型心绞痛;2=非典型心绞痛;3=非心绞痛;4=无症状)。
-
trestbps--静息血压
-
chol - 血清胆固醇,单位:mg/dl
-
fbs - 空腹血糖水平>120 mg/dl(1=真;0=假)
-
restecg - 静息心电图结果(0=正常;1=有ST-T;2=肥大)
-
thalach - 达到的最大心率
-
exang - 运动诱发的心绞痛(1=是;0=否)
-
oldpeak - 相对于静止状态,运动诱发的ST压低
-
slope - 运动时ST段峰值的斜率(1=上斜;2=平坦;3=下斜)
-
ca - 主要血管的数量(0-4),由Flourosopy着色
-
地中海贫血症--地中海贫血症是一种遗传性血液疾病,会影响身体产生血红蛋白和红细胞的能力。1=正常;2=固定缺陷;3=可逆转缺陷
-
目标--预测属性--心脏疾病的诊断(血管造影疾病状态)(值0=<50%直径狭窄;值1=>50%直径狭窄)
在Rstudio中加载数据
heart<-read.csv("heart.csv",header = T)header = T意味着给定的数据有自己的标题,或者换句话说,第一个观测值也被考虑用于预测。
head(heart)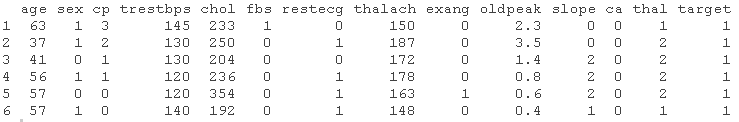
当我们想查看和检查数据的前六个观察点时,我们使用head函数。
tail(heart)
显示的是我们数据中最后面的六个观察点
colSums(is.na(heart))
这个函数是用来检查我们的数据是否包含任何NA值。
如果没有发现NA,我们就可以继续前进,否则我们就必须在之前删除NA。
检查我们的数据结构
str(heart)
查看我们的数据摘要
summary(heart)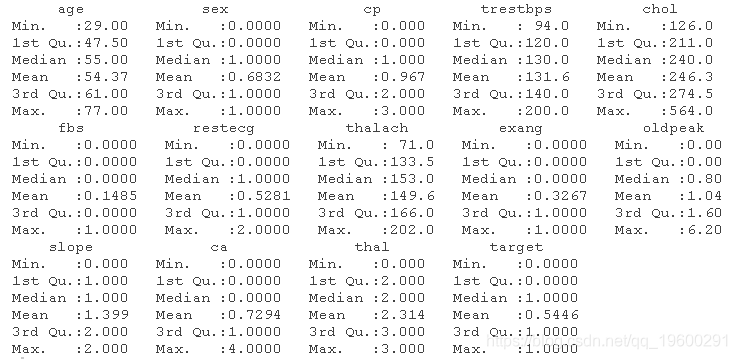
通过观察以上的总结,我们可以说以下几点
-
性别不是连续变量,因为根据我们的描述,它可以是男性或女性。因此,我们必须将性别这个变量名称从整数转换为因子。
-
cp不能成为连续变量,因为它是胸痛的类型。由于它是胸痛的类型,我们必须将变量cp转换为因子。
-
fbs不能是连续变量或整数,因为它显示血糖水平是否低于120mg/dl。
-
restecg是因子,因为它是心电图结果的类型。它不能是整数。所以,我们要把它转换为因子和标签。
-
根据数据集的描述,exang应该是因子。心绞痛发生或不发生。因此,将该变量转换为因子。
-
斜率不能是整数,因为它是在心电图中观察到的斜率类型。因此,我们将变量转换为因子。
-
根据数据集的描述,ca不是整数。因此,我们要将该变量转换为因子。
-
thal不是整数,因为它是地中海贫血的类型。因此,我们将变量转换为因子。
-
目标是预测变量,告诉我们这个人是否有心脏病。因此,我们将该变量转换为因子,并为其贴上标签。
根据上述考虑,我们对变量做了一些变化
-
#例如
-
sex<-as.factor(sex)
-
levels(sex)<-c("Female","Male")
检查上述变化是否执行成功
str(heart)
summary(heart)
EDA
EDA是探索性数据分析(Exploratory Data Analysis)的缩写,它是一种数据分析的方法/哲学,采用各种技术(主要是图形技术)来深入了解数据集。
对于图形表示,我们需要库 "ggplot2"
-
library(ggplot2)
-
ggplot(heart,aes(x=age,fill=target,color=target)) + geom_histogram(binwidth = 1,color="black") + labs(x = "Age",y = "Frequency", title = "Heart Disease w.r.t. Age")
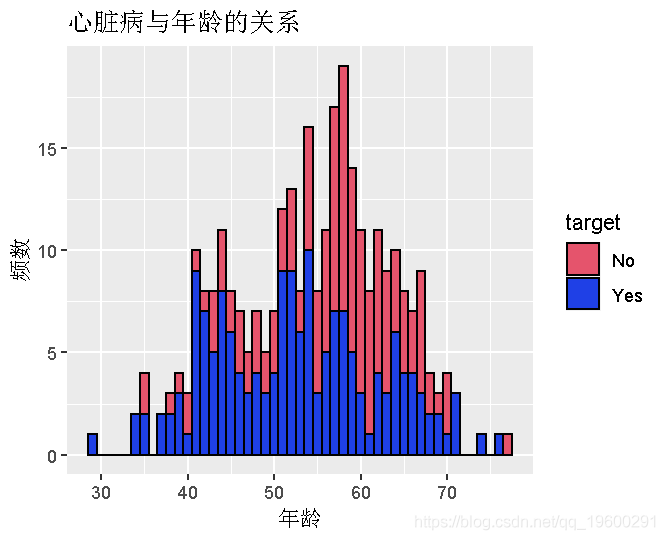
我们可以得出结论,与60岁以上的人相比,40至60岁的人患心脏病的概率最高。
-
table <- table(cp)
-
-
pie(table)
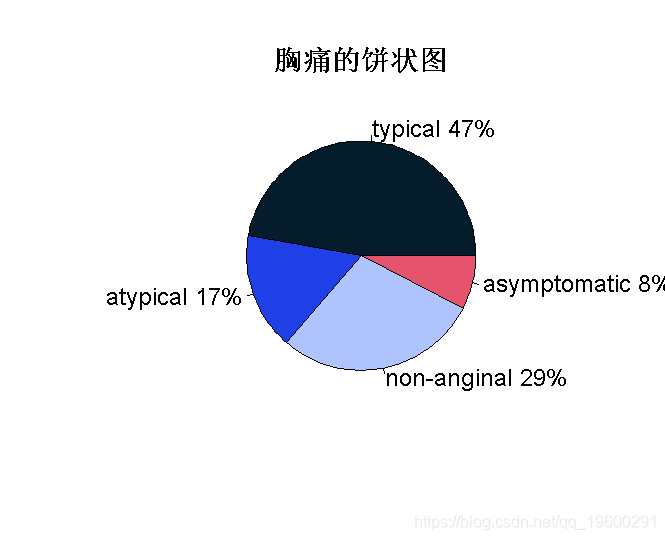
我们可以得出结论,在所有类型的胸痛中,在个人身上观察到的大多数是典型的胸痛类型,然后是非心绞痛。
执行机器学习算法
Logistic回归
首先,我们将数据集分为训练数据(75%)和测试数据(25%)。
-
set.seed(100)
-
#100用于控制抽样的permutation为100.
-
index<-sample(nrow(heart),0.75*nrow(heart))
在训练数据上生成模型,然后用测试数据验证模型。
-
glm(family = "binomial")
-
# family = " 二项式 "意味着只包含两个结果。
为了检查我们的模型是如何生成的,我们需要计算预测分数和建立混淆矩阵来了解模型的准确性。
-
pred<-fitted(blr)
-
# 拟合只能用于获得生成模型的数据的预测分数。

我们可以看到,预测的分数是患心脏病的概率。但我们必须找到一个适当的分界点,从这个分界点可以很容易地区分是否患有心脏病。
为此,我们需要ROC曲线,这是一个显示分类模型在所有分类阈值下的性能的图形。它将使我们能够采取适当的临界值。
-
pred<-prediction(train$pred,train$target)
-
perf<-performance(pred,"tpr","fpr")
-
plot(perf,colorize = T,print.cutoffs.at = seq(0.1,by = 0.1))
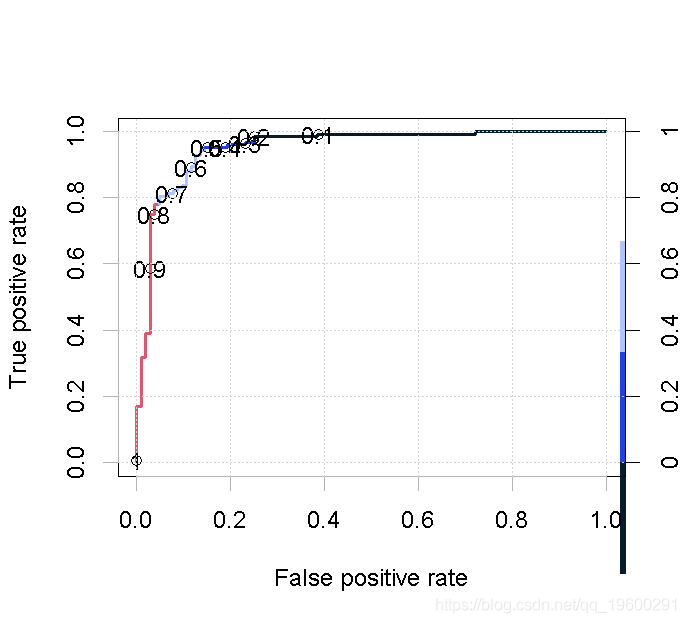
通过使用ROC曲线,我们可以观察到0.6具有更好的敏感性和特异性,因此我们选择0.6作为区分的分界点。
pred1<-ifelse(pred<0.6,"No","Yes")

-
# 训练数据的准确性
-
acc_tr
![]()
从训练数据的混淆矩阵中,我们知道模型有88.55%的准确性。
现在在测试数据上验证该模型
-
predict(type = "response")
-
## type = "response "是用来获得患有心脏病的概率的结果。
-
head(test)

我们知道,对于训练数据来说,临界点是0.6。同样地,测试数据也会有相同的临界点。
confusionMatrix((pred1),target)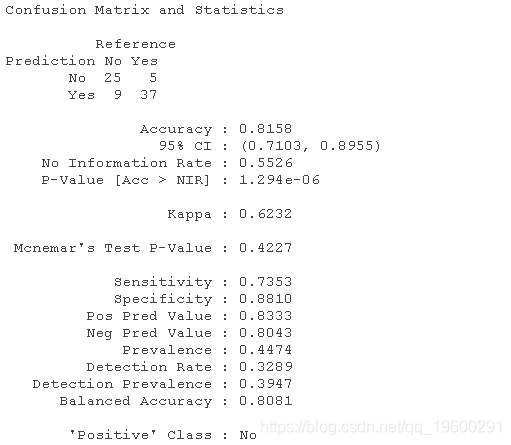
#测试数据的准确性.
![]()
检查我们的预测值有多少位于曲线内
auc@y.values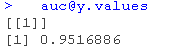
我们可以得出结论,我们的准确率为81.58%,90.26%的预测值位于曲线之下。同时,我们的错误分类率为18.42%。
Naive Bayes算法
在执行Naive Bayes算法之前,需要删除我们在执行BLR时添加的额外预测列。
-
#naivebayes模型
-
nB(target~.)
用训练数据检查模型,并创建其混淆矩阵,来了解模型的准确程度。
-
predict(train)
-
confMat(pred,target)

![]()
我们可以说,贝叶斯算法对训练数据的准确率为85.46%。
现在,通过预测和创建混淆矩阵来验证测试数据的模型。
Matrix(pred,target)
![]()
我们可以得出结论,在Naive Bayes算法的帮助下生成的模型准确率为78.95%,或者我们也可以说Naive Bayes算法的错误分类率为21.05%。
决策树
在实施决策树之前,我们需要删除我们在执行Naive Bayes算法时添加的额外列。
train$pred<-NULL
rpart代表递归分区和回归树
当自变量和因变量都是连续的或分类的时候,就会用到rpart。
rpart会自动检测是否要根据因变量进行回归或分类。
实施决策树
plot(tree)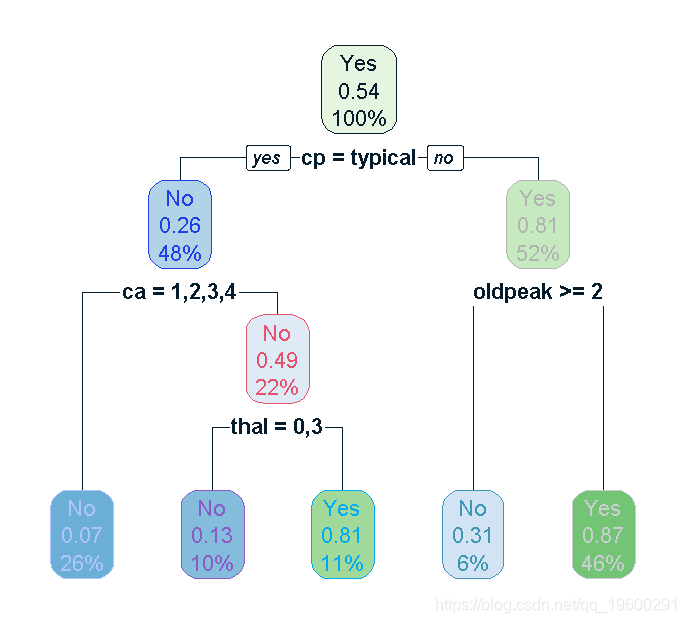
在决策树的帮助下,我们可以说所有变量中最重要的是CP、CA、THAL、Oldpeak。
让我们用测试数据来验证这个模型,并找出模型的准确性。
conMat(pred,targ)
![]()
我们可以说,决策树的准确率为76.32%,或者说它的错误分类率为23.68%。
随机森林
在执行随机森林之前,我们需要删除我们在执行决策树时添加的额外预测列。
test$pred<-NULL在随机森林中,我们不需要将数据分成训练数据和测试数据,我们直接在整个数据上生成模型。为了生成模型,我们需要使用随机森林库
-
# Set.seed通过限制permutation来控制随机性。
-
-
set.seed(100)
-
model_rf<-randomForest(target~.,data = heart)
-
model_rf

在图上绘制出随机森林与误差的关系。
plot(model_rf)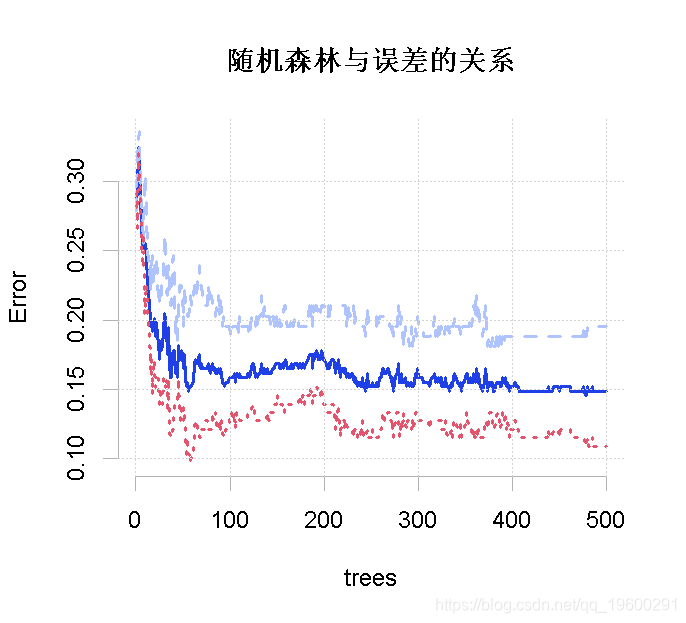
红线代表没有心脏病的MCR,绿线代表有心脏病的MCR,黑线代表总体MCR或OOB误差。总体误差率是我们感兴趣的,结果不错。
结论
在进行了各种分类技术并考虑到它们的准确性后,我们可以得出结论,所有模型的准确性都在76%到84%之间。其中,随机森林的准确率略高,为83.5%。

最受欢迎的见解
3.matlab中的偏最小二乘回归(PLSR)和主成分回归(PCR)
5.R语言回归中的Hosmer-Lemeshow拟合优度检验