原文链接:http://tecdat.cn/?p=22956
原文出处:拓端数据部落公众号
贝叶斯网络(BN)是一种基于有向无环图的概率模型,它描述了一组变量及其相互之间的条件依赖性。它是一个图形模型,我们可以很容易地检查变量的条件依赖性和它们在图中的方向。
在这篇文章中,我将简要地学习如何用R来使用贝叶斯网络。
本教程旨在介绍贝叶斯网络学习和推理的基础知识,使用真实世界的数据来探索图形建模的典型数据分析工作流程。关键点将包括:
- 预处理数据;
- 学习贝叶斯网络的结构和参数。
- 使用网络作为预测模型。
- 使用网络进行推理。
- 通过与外部信息的对比来验证网络的有效性。
快速介绍
贝叶斯网络
定义
贝叶斯网络(BNs)的定义是:
- 一个网络结构,一个有向无环图
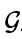 , 其中每个节点
, 其中每个节点 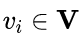 对应于一个随机变量
对应于一个随机变量 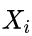 ;
; - 一个全局概率分布
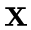 (带参数
(带参数 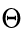 ), 它可以根据图中存在的弧被分解成更小的局部概率分布。
), 它可以根据图中存在的弧被分解成更小的局部概率分布。
网络结构的主要作用是通过图形分离来表达模型中各变量之间的条件独立性关系,从而指定全局分布的因子化。
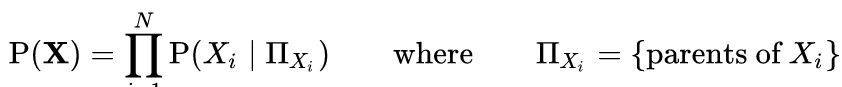
每个局部分布都有自己的参数集 ![]() ; 而⋃
; 而⋃ ![]() 要比
要比![]() 小得多,因为许多参数是固定的,因为它们所属的变量是独立的。
小得多,因为许多参数是固定的,因为它们所属的变量是独立的。
R实现了以下学习算法。
基于约束的:PC, GS, IAMB, MMPC, Hilton-PC
基于分数的:爬山算法、Tabu Search
配对的:ARACNE, Chow-Liu
混合:MMHC, RSMAX2
我们使用基于分数的学习算法,希尔算法。首先,我们将先为本教程生成简单的数据集。
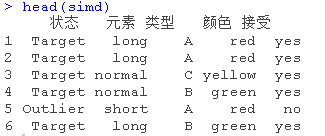
在这个数据集中,'状态'与'元素'和'接受'列有关系。而'类型'与'颜色'列有关系。当你创建一个带有分类数据的数据框时,列应该是一个因子类型。否则,该数据框不能用于BN结构的创建。

接下来,我们将创建学习结构。

我们可以在一个图中看到结构。
> plot(hc_simd)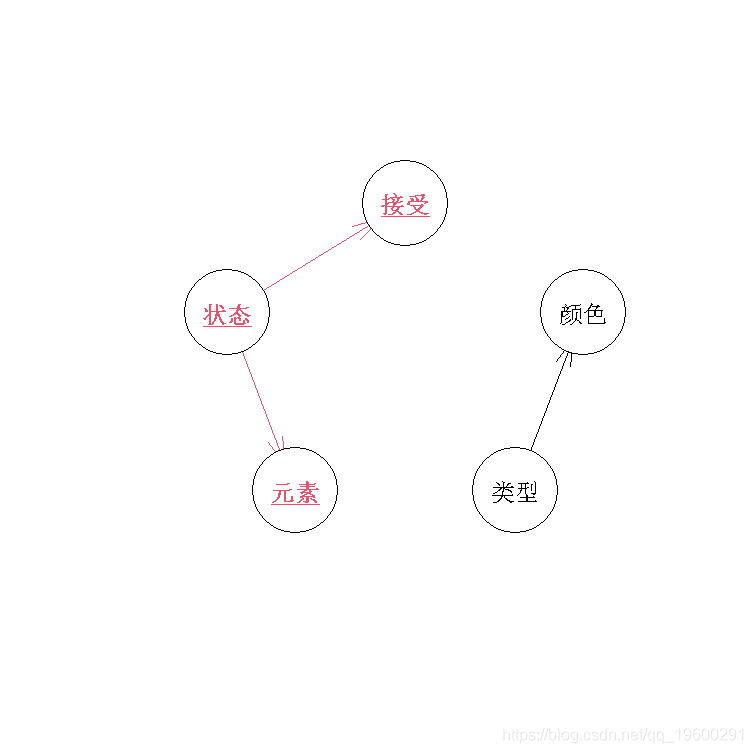
在这个图中,状态、元素、接受、类型和颜色被称为节点。节点之间的方向用弧线描述,弧线是一个包含从元素到元素方向数据的矩阵。
如上弧线显示,在我们的数据中存在'类型'到'颜色',以及'状态'到'接受'和'元素'的关系。'类型'和'状态'是两个独立的组,它们之间不存在相互依赖关系。
接下来,我们将用数据来拟合模型。
simd_fitted


基于上述训练数据,我们可以进行条件概率查询。
我们检查 "Outlier "和 "Target "的状态概率。
![]()
该样本成为 "离群 "的概率为51%。

状态成为 "目标 "的概率是0%。
错颌畸形数据的贝叶斯网络分析
问题:受第三类错牙合畸形影响的患者(以下牙弓突出为特征),其骨骼不平衡在生命早期就产生,在青春期和骨骼成熟前会变得更加明显。在单个III类患者中早期预测治疗的成功或失败,使其更容易矫正,但仅从少量的形态决定因素中预测是很难做到的。原因是III类错颌畸形很少是单一颅面部件异常的结果,所以单个的临床和放射学测量值可能不如测量值本身的相互作用具有指示性。
任务:
- 我们学习一个BN,并使用它来确定和可视化在成长和治疗过程中各种III类错位颌面特征之间的相互作用。
- 我们通过验证一些普遍接受的关于这些骨骼不平衡演变的假说来检验其一致性。
- 我们表明,与接受快速上颌扩张和面罩治疗的正畸患者相比,未经治疗的受试者形成了不同的III类颅面生长模式。
- 在接受治疗的患者中,CoA段(上颌骨长度)和ANB角(上颌骨与下颌骨的前后关系)似乎是接受治疗的主要影响的骨骼亚空间。
数据
我们将使用的数据集包含143名患者,在T1和T2年龄段有两组测量数据(以年为单位),用于以下变量。
治疗:未经治疗(NT),治疗后效果不好(TB),治疗后效果好(TG)。生长:一个二元变量,数值为好或坏。ANB:唐氏点A和B之间的角度(度)。IMPA:门牙-下颌平面角(度)。PPPM:腭平面-下颌平面的角度(度)。CoA:上颌骨从髁状突到唐氏点A的总长度(mm)。GoPg:下颌体从齿龈到齿龈的长度(mm)。CoGo:下颌骨的长度,从髁状突到齿状突(mm)。
所有的测量都是通过X射线扫描得出的,使用一套参考点建立的图,如以下。
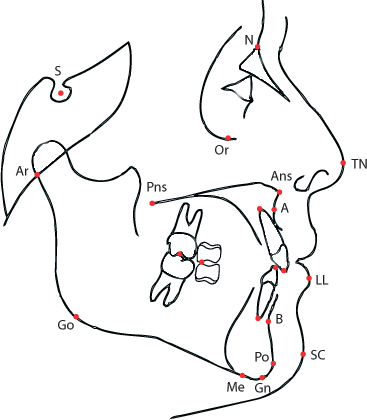
> str(data)

预处理和探索性数据分析
首先,我们创建一个数据框架,其中包括所有变量的差异以及增长和治疗。
生长和治疗变量带有关于病人预后的冗余信息,这一点从TB和TG之间生长良好的病人比例的差异中可以看出。
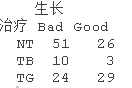
为了避免在模型中包括这两个变量所导致的混杂,我们将治疗重新编码为一个二元变量,0表示NT,1表示TB或TG。同样地,我们对成长进行重新编码,0表示坏,1表示好。

由于我们将使用高斯BN进行分析,检查这些变量是否是正态分布;从下面的图来看,似乎并非所有的变量都是如此。
-
-
+ hist(x, prob = TRUE )
-
+ lines(density(x), lwd = 2 )
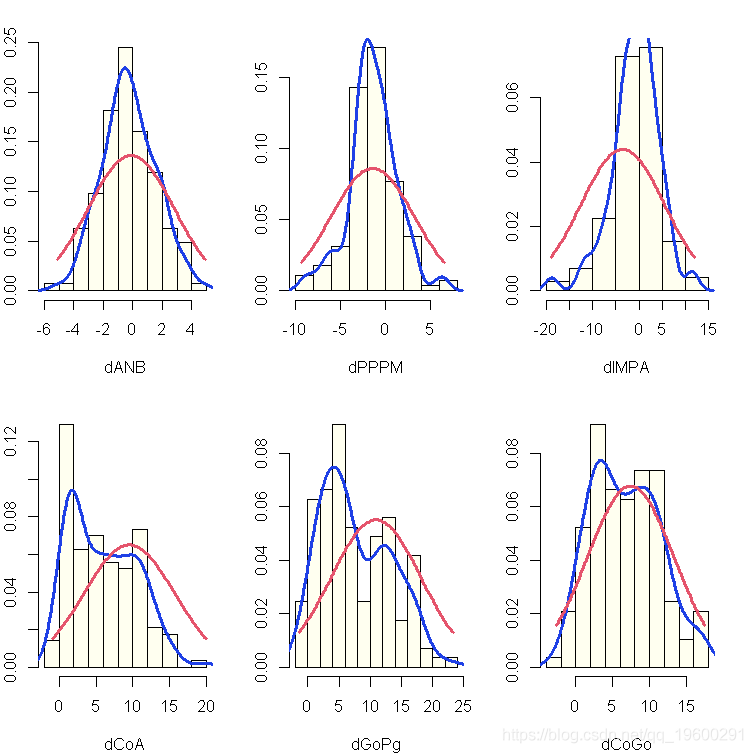
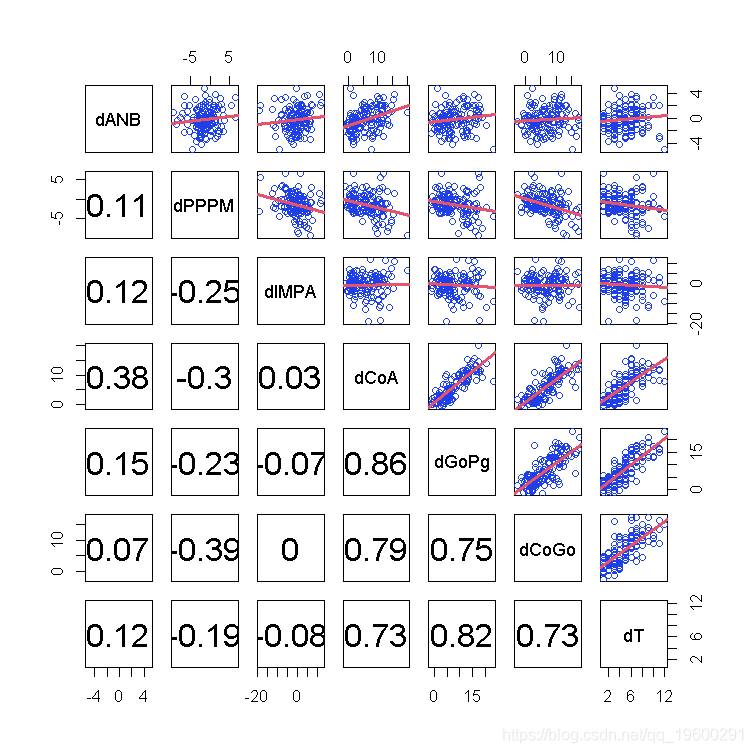
这些变量是通过线性关系联系起来的吗?其中一些是,但不是全部。
> pairs(diff[, setdiff(names(diff)
最后,我们可以看看这些变量是否以任何方式聚在一起,因为聚在一起的变量更有可能在BN中发生联系。
-
-
> heatmap(rho)

我们可以在热图中看到两个集群:第一个集群包括dCoGo、dGoPg和dCoA,第二个集群包括Treatment、dANB和dCoA。第一个聚类在临床上很有意思,因为它包括治疗和两个都与唐氏A点有关的变量,这为治疗的主要效果提供了一些线索。
plot(ug )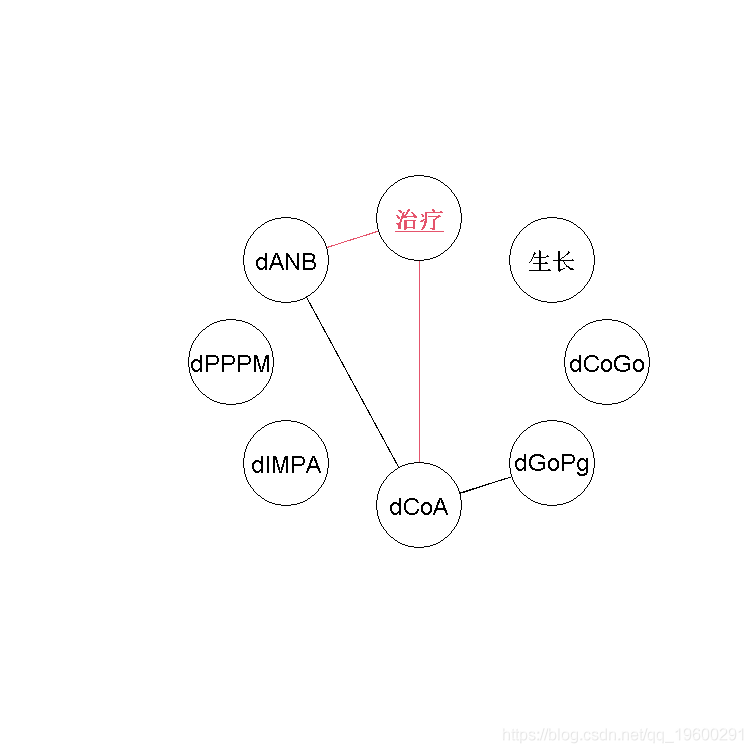
模型#1:作为差异模型的静态贝叶斯网络
在这里,我们使用保存在diff中的差异来为数据建模,而不是原始值;我们将使用GBN处理,因为所有变量都是数字。对差异进行建模会导致局部分布,其形式为回归模型
![]()
其中![]() 对于其他回归因子,以此类推。我们可以将这种回归改写为
对于其他回归因子,以此类推。我们可以将这种回归改写为
这是一组微分方程,对变化率进行建模,其关系被假定为很好地近似于线性关系。然而,这种表述仍然意味着原始值随时间线性变化,因为变化率取决于其他变量的变化率,但不取决于时间本身。要有一个非线性的趋势,我们需要
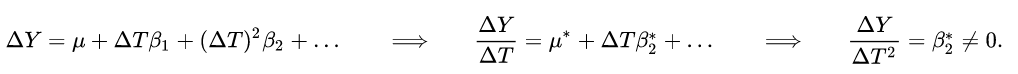
此外,包括增长变量意味着我们可以有以下形式的回归模型
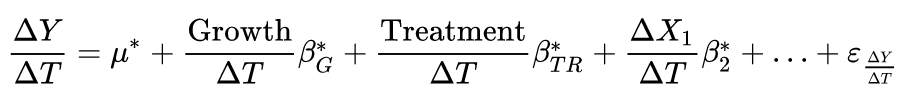
从而允许不同的变化率,这取决于病人是否在畸形中表现出积极的发展,以及他是否正在接受治疗。
学习贝叶斯网络
学习结构
学习BN的第一步是学习其结构,即DAG ![]() . 我们可以使用数据(来自不同的数据框架)结合先验知识来做这件事;结合后者可以减少我们必须探索的模型空间,并生成更强大的BN。一个直接的方法是将那些编码我们知道不可能/真实的关系的弧列入黑名单; 并将那些编码我们知道存在的关系的弧列入白名单。
. 我们可以使用数据(来自不同的数据框架)结合先验知识来做这件事;结合后者可以减少我们必须探索的模型空间,并生成更强大的BN。一个直接的方法是将那些编码我们知道不可能/真实的关系的弧列入黑名单; 并将那些编码我们知道存在的关系的弧列入白名单。
黑名单只是一个矩阵(或一个数据框),其中有from和to两列,列出了我们不希望在BN中出现的弧。
- 我们把任何指向正畸变量中的dT、治疗和生长的弧列入黑名单。
- 我们将从dT到Treatment的弧列入黑名单。这意味着一个病人是否被治疗不会随时间而改变。
- 我们将从生长到dT和治疗的弧线列入黑名单。这意味着病人是否接受治疗不会随时间变化,而且显然不会因预后而变化。

白名单的结构与黑名单相同。
- 我们将依赖结构dANB → dIMPA ← dPPPM列入白名单。
- 我们将从dT到Growth的弧线列入白名单,这使得预后可以随时间变化。

一个简单的学习 ![]() 方法是在整个数据上找到具有最佳拟合度的网络结构。例如,使用hc()与默认分数(BIC)和整个diff数据框架。
方法是在整个数据上找到具有最佳拟合度的网络结构。例如,使用hc()与默认分数(BIC)和整个diff数据框架。

至于绘图,关键函数是plot()。
plot(dag, , highlight )
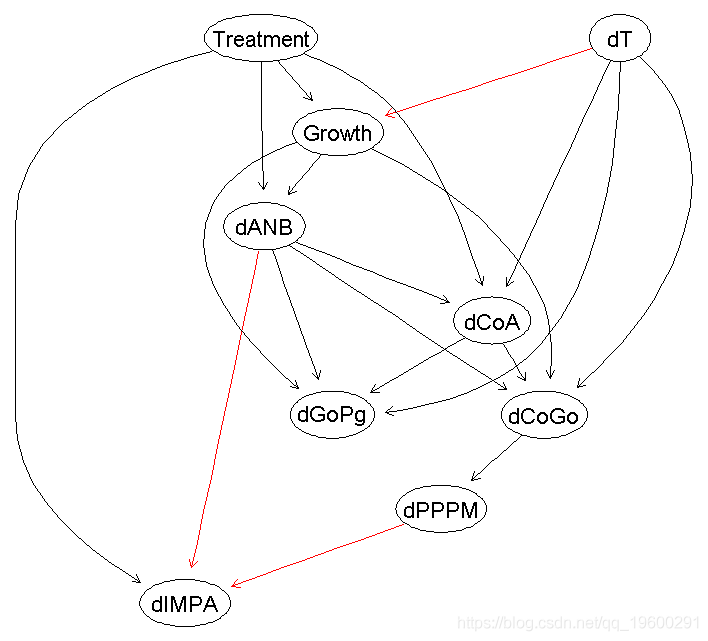
然而,dag的质量关键取决于变量是否是正态分布,以及连接它们的关系是否是线性的;从探索性分析来看,并不清楚所有的变量都是如此。我们也不知道哪些弧线代表强关系,也就是说,它们能抵抗数据的扰动。我们可以用boot来解决这两个问题。
- 使用bootstrap对数据重新取样。
- 从每个bootstrap样本中学习一个单独的网络。
- 检查每个可能的弧在网络中出现的频率。
- 用出现频率较高的弧构建一个共识网络。
-
booth(diff, R = 200)
-
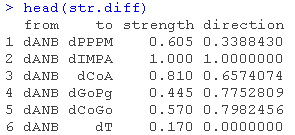
boot.strength()的返回值包括,对于每一对节点,连接它们的弧的强度(例如,我们观察到dANB → dPPPM或dPPPM → dANB的频率)及其方向的强度(例如,当我们观察到dANB和dPPPM之间有弧时,我们观察到dANB → dPPPM的频率)。
attr( "threshold")
![]()
因此,averaged.network()取所有强度至少为0.585的弧,并返回一个平均的共识网络,除非指定不同的阈值。
> avg.diff = averaged.network(str.diff)纳入我们现在拥有的关于弧线强度的信息。
> strength.plot(avg.diff, str.diff, shape = "ellipse", highlight = list(arcs = wl)) 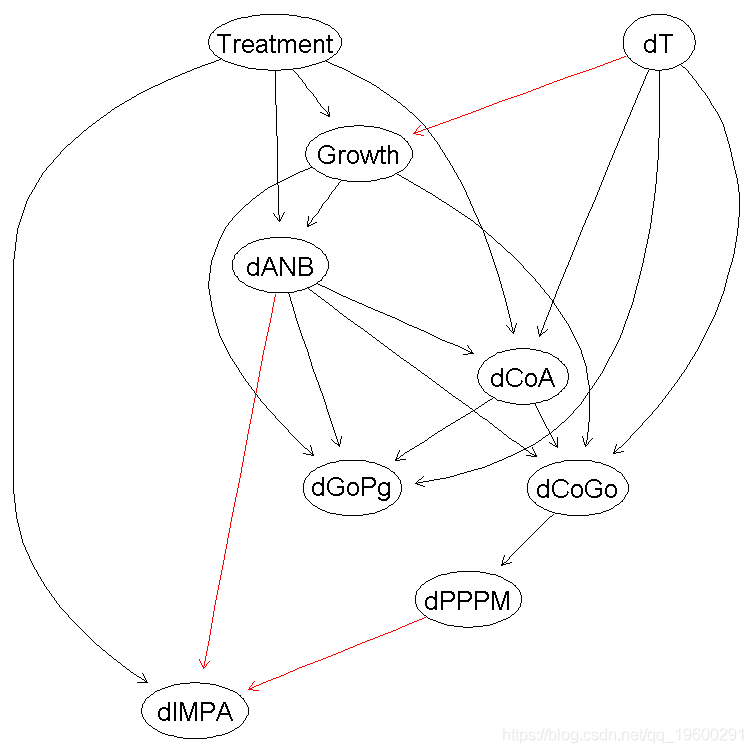
我们如何将平均的网络(avg.diff)与我们最初从所有数据中学习到的网络(dag)进行比较?最定性的方法是将两个网络并排绘制,节点位置相同,并突出显示一个网络中出现而另一个网络中没有的弧,或者出现的方向不同的弧。
-
> par(mfrow = c(1, 2))
-
> graphviz.compare(avg.diff, dag, shape = "ellipse", main = c("averaged DAG", "single DAG"))
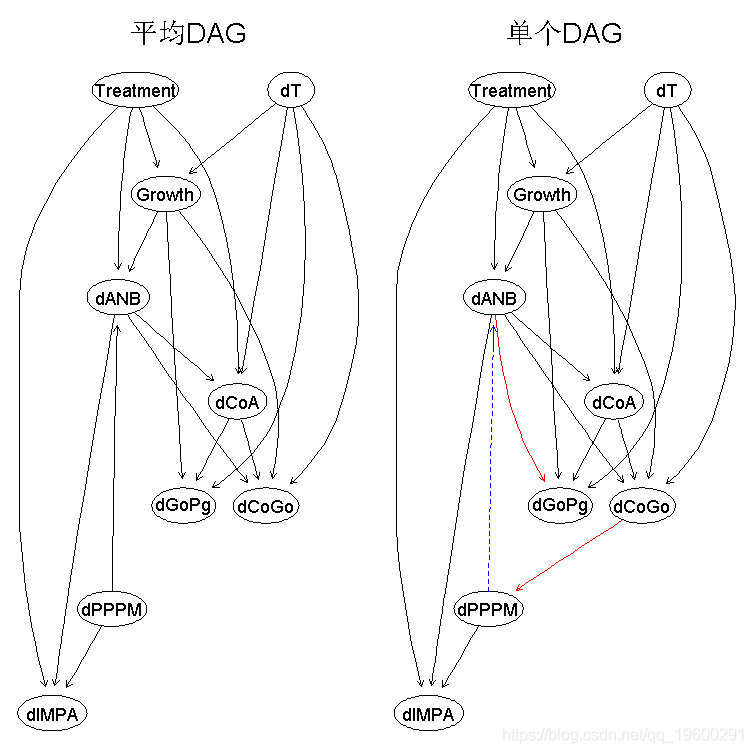
我们可以看到,Treatment→dIMPa、dANB→dGoPg和dCoGo→dPPPM这些弧线只出现在平均网络中,而dPPPM→dANB只出现在我们从所有数据中学到的网络中。我们可以假设,前三个弧被数据的噪声加上小样本量和偏离常态的情况所隐藏。编程可以返回真阳性(出现在两个网络中的弧)和假阳性/阴性(只出现在两个网络中的一个的弧)的数量。
> compare

或弧=TRUE。

但是,考虑到网络是用BIC学习的,而BIC是等价的,那么所有的弧线方向是否都很确定?看一下dag和avg.diff的CPDAGs(并考虑到白名单和黑名单),我们看到没有无方向的弧。所有弧的方向都是唯一的。
最后,我们可以结合来进行原则性的比较,如果两个弧被唯一确定为不同,我们就说它们是不同的。

也可以看一下关于弧长分布的阈值:平均的网络是相当密集的(9个节点有17个弧),很难阅读。
-
> plot(str.diff)
-
> abline(v = 0.75, col = "tomato", lty = 2, lwd = 2)
-
> abline(v = 0.85, col = "steelblue", lty = 2, lwd = 2)
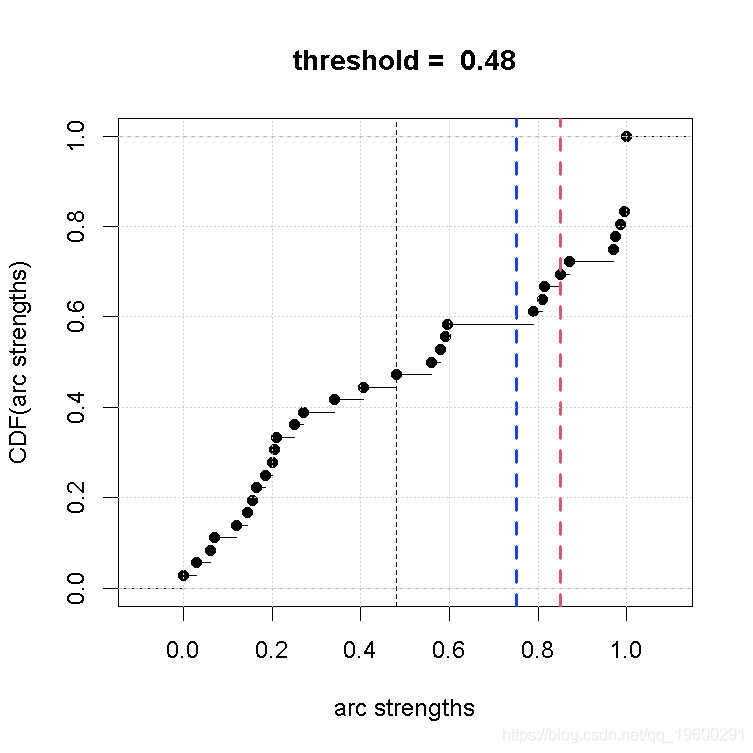
因此,把阈值提高一点,多剔除几个弧就好了。看一下上面的图,由于弧长分布的差距,较高的阈值的两个自然选择是0.75(红色虚线)和0.85(蓝色虚线)。
-
> nrow( strength > "threshold" direction > 0.5, ])
-
[1] 18
-
trength > 0.75 & direction > 0.5
-
[1] 15
-
strength > 0.85 & direction > 0.5
-
[1] 12
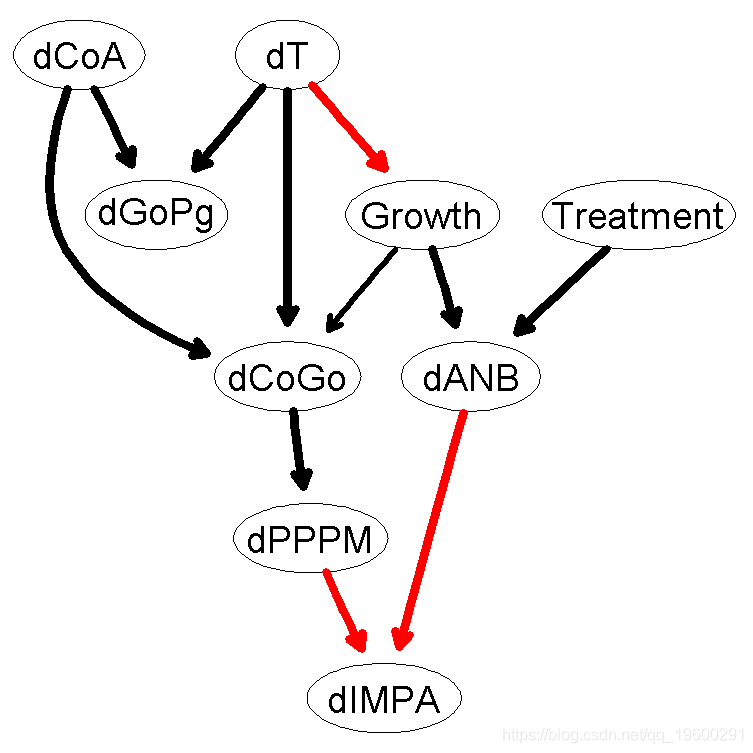
我们通过在 network()中设置阈值=0.85得到的更简单的网络如下所示;从定性的角度来看,它当然更容易推理。
-
> avg.simpler = averaged.network(str.diff, threshold = 0.85)
-
> strength.plot(avg.simpler, str.diff, shape = "ellipse", highlight = list(arcs = wl))
学习参数
在学习了结构之后,我们现在可以学习参数。由于我们正在处理连续变量,我们选择用GBN来建模。因此,如果我们使用最大似然估计来拟合网络的参数,我们就会发现每个局部分布都是一个典型的线性回归。
fit(avg, diff)



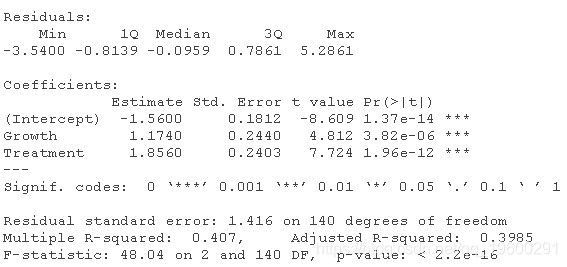
我们可以通过比较bn.fit()和lm()产生的模型,例如dANB,很容易确认这是事实。
-
-
> summary(lm(dANB ~ Growth + Treatment, data = diff))

我们会不会有共线性的问题?理论上是可能的,但在实践中,从数据中学习的网络结构大多不是问题。原因是,如果两个变量 ![]() 和
和![]() 是共线性的,在增加(比如说)Xi←Xj之后,那么Xj←Xk将不再显著提高BIC,因为Xj和Xk(在某种程度上)提供了关于Xi的相同信息。
是共线性的,在增加(比如说)Xi←Xj之后,那么Xj←Xk将不再显著提高BIC,因为Xj和Xk(在某种程度上)提供了关于Xi的相同信息。
-
-
> # 逐渐增加解释变量之间的关联性。
-
> for (rho 5)) {
-
-
+ # 更新相关矩阵并生成数据。
-
+ R = R = rho
-
+ data = as.data.frame(mvrnorm(1000))
-
+ # 比较线性模型
-
+ cat( " BIC:",
-
-
+ }
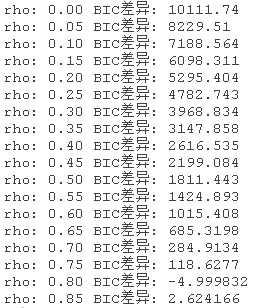
比较线性模型
如果参数估计因任何原因出现问题,我们可以用一组新的、来自不同方法的估计值来取代它们。
-
-
dANB
-
-
dANB = penalized( dANB)
-
dANB


模型验证
有两种主要的方法来验证一个BN。
- 只看网络结构:如果学习BN的主要目标是识别弧和路径,当BN被解释为因果模型时,通常是这种情况,我们可以进行本质上的路径分析和研究弧的强度。
- 将BN视为一个整体,包括参数:如果学习BN的主要目标是将其作为一个专家模型,那么我们可能想。
- 根据其他一些变量的值,预测新个体的一个或多个变量的值;以及
- 将CP查询的结果与专家知识进行比较,以确认BN反映了关于我们正在建模的现象的最佳知识。
- 根据其他一些变量的值,预测新个体的一个或多个变量的值;以及
预测准确性
我们可以用通常的方法来衡量我们所选择的学习策略的预测准确性,即交叉验证。实现了:
- k-fold交叉验证;
- 指定的k进行交叉验证;
- hold-out 交叉验证
对于:
- 结构学习算法(结构和参数都是从数据中学习的)。
- 参数学习算法(结构由用户提供,参数从数据中学习)。
首先,我们检查Growth,它编码了错牙合畸形的演变(0表示坏,1表示好)。我们检查它,把它转回离散变量并计算预测误差。
-
cv(diff)
-
-
> for (i in 1:10) {
-
-
+ err[i] = (sum(tt) - sum(diag(tt))) / sum(tt)
-
-
+ }
-
>
-

其他变量是连续的,所以我们可以估计它们的预测相关性来代替。
-
-
> for (var in names(predcor)) {
-
-
+ xval = cv(diff)
-
-
+ predcor[var] = mean(sapply(xval, function(x) attr(x, "mean")))
-
-
+ }
![]()
![]()
在这两种情况下,我们使用损失函数的变体,它使用从所有其他变量计算的后验期望值进行预测。基本的损失函数(cor, mse, pred)仅仅从它们的父代来预测每个节点的值,这在处理很少或没有父代的节点时是没有意义的。
用专家知识进行确认
确认BN是否有意义的另一种方法是把它当作的工作模型,看看它是否表达了关于关键事实,这些事实在学习过程中没有作为先验知识使用。否则,我们将只是拿回我们放在先验中的信息)。一些例子。
- "CoGo的过度增长应该会引起PPPM的减少"。
我们通过为存储在 fitted.simpler中的BN生成dCoGo和dPPPM的样本,并假设没有发生任何处理,来测试这个假设。随着dCoGo的增加(这表明增长越来越快),DPPPM变得越来越负(这表明假设角度最初是正的,则角度会减少。-
> sim = dist(fitted.simpler)
-
> plot(sim )
-
> abline(v = 0, col = 2, lty = 2, lwd = 2)

-
- "CoGo的小幅增长应该会引起PPPM的增长。"
从上图来看,CoGo的负增长或空增长(dCoGo ⋜ 0)对应于PPPM的正增长,概率为≈0.60。对于CoGo的小幅增长(dCoGo∈[0, 2]),不幸的是,dPPPM ⋜0,概率≈0.50,所以BN不支持这一假设。-
> nrow(sim[( dCoGo <= 0) & ( PPPM > 0), ]) / nrow(sim[( dCoGo <= 0), ])
-
[1] 0.6112532
-
> nrow(sim[( dCoGo > 0) & ( dCoGo < 2) & ( dPPPM > 0), ]) /
-
+ nrow(sim[( CoGo) > 0 & ( dCoGo < 2), ])
-
[1] 0.4781784
-
- "如果ANB减少,IMPA就会减少以进行补偿。"
像以前一样通过模拟测试,我们正在寻找与IMPA(相同)的负值相关的dANB的负值(这表明假设角度最初是正的,就会减少)。从下图中可以看出,dANB与dIMPA成正比,所以其中一个的减少表明另一个的减少;两者的平均趋势(黑线)同时为负。-
-
> plot(sim )
-
-
> abline(coef(lm(dIMPA ~ dANB ))

-
- "如果GoPg强烈增加,那么ANB和IMPA都会减少。" 如果我们从BN中模拟dGoPg、dANB和dIMPA,假设dGoPg>5(即GoPg在增加),我们估计dANB>0(即ANB在增加)的概率为≈0.70,dIMPA<0的概率仅为≈0.58。
-
-
> nrow(sim[(dGoPg > 5) & (dANB < 0), ]) / nrow(sim[(dGoPg > 5), ])
-
[1] 0.695416
-
> nrow(sim[(dGoPg > 5) & (dIMPA < 0), ]) / nrow(sim[(dGoPg > 5), ])
-
[1] 0.5756936
-
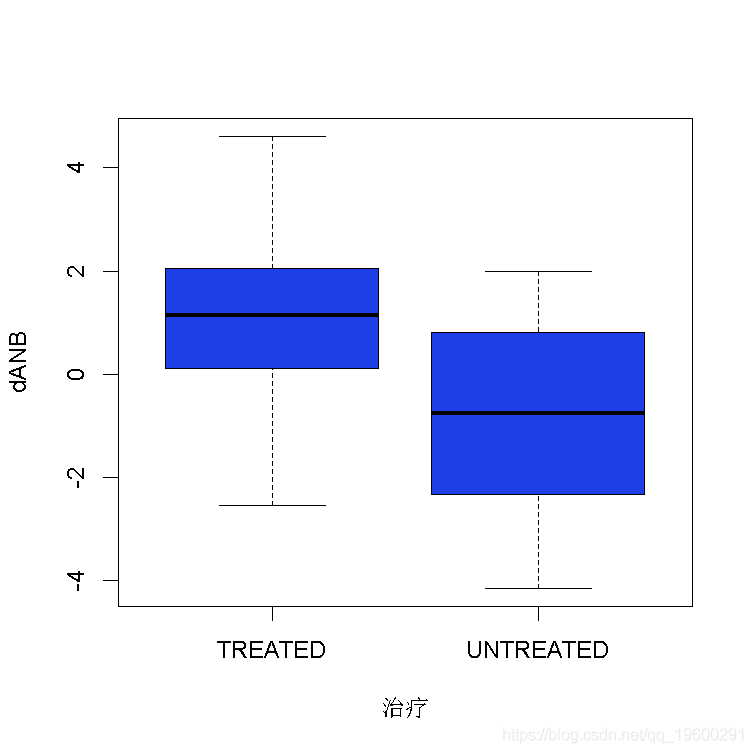
- "治疗试图阻止ANB的减少。如果我们固定ANB,治疗过的病人和未治疗过的病人是否有区别?"
首先,我们可以检查在没有任何干预的情况下,dANB≈0的病人的治疗和增长之间的关系(即使用我们从数据中得知的BN)。-
dist(fitted )
-
table(TREATMENT = Treatment < 0.5, GOOD.GROWTH = Growth > 0.5)
-

-
- 估计的P(GOOD.GROWTH ∣ TREATMENT)对于接受治疗和未接受治疗的病人是不同的(≈0.65对≈0.52)。
如果我们模拟一个正式的干预措施(如Judea Pearl),并从外部设置dANB=0(从而使其独立于其父母,并删除相应的弧),我们就会发现GOOD.GROWTH对于接受治疗和未接受治疗的病人来说实际上具有相同的分布,从而变得与TREATMENT无关。这表明,有利的预后确实是由防止ANB的变化决定的,而治疗的其他成分(如果有的话)就变得不重要了。table(TREATMENT = Treatment < 0.5, GOOD.GROWTH = Growth > 0.5)
- "治疗试图阻止ANB的减少。如果我们固定ANB,治疗和未治疗的病人之间是否有区别?"
评估的方法之一是检查在保持GoPg固定的情况下,A点和B点之间的角度(ANB)是否在治疗和未治疗的病人之间发生变化。
假设GoPg不发生变化,对于接受治疗的病人来说,A点和B点之间的角度会增加(强烈的负值表示水平不平衡,所以正的变化率表示不平衡的减少),而对于未接受治疗的病人来说则会减少(不平衡会随着时间慢慢恶化)。
-
Treatment = c("UNTREATED", "TREATED")[(Treatment > 0.5) + 1L]
-
boxplot(dANB ~ Treatment)
模型#2:动态贝叶斯网络
动态贝叶斯网络在预测方面的效果不如1号模型好,同时更加复杂。这是动态贝叶斯网络所固有的,即模拟随机过程的贝叶斯网络:每个变量都与被模拟的每个时间点的不同节点相关。(通常情况下,我们假设过程是一阶马尔可夫,所以我们在BN中有两个时间点:t和t-1。)然而,我们探索它的目的是为了说明这样一个BN可以被学习并用于bnlearn。
我们用于这个模型的数据是我们在分析开始时存储到正交的原始数据。然而,我们选择使用治疗变量而不是生长变量作为变量来表达受试者可能正在接受医疗的事实。原因是生长变量是一个衡量第二次测量时的预后的变量,它的值在第一次测量时是未知的;而治疗变量在两次测量时都是相同的。
学习结构
首先,我们将变量分为三组:时间为t2的变量,时间为t1=t2-1的变量,以及与时间无关的变量,因为它们在t1和t1取值相同。
-
-
> t2.variables
-
然后我们引入一个黑名单,其中。
- 我们将所有从临床变量到T1、T2和治疗的弧线列入黑名单,因为我们知道,年龄和治疗不是由临床测量决定的。
- 我们将所有进入Treatment和t1时间段的所有变量的弧列入黑名单,因为我们假设t1时间段的变量之间的弧与t2时间段的相应变量是一样的,两次学习它们是没有意义的。
- 我们将所有从t2到t1的弧列入黑名单。
-
grid(from = setdiff(names(ortho), c("T1", "T2")),
-
to = c("T1", "T2"))
相比之下,我们只对T1→T2的弧线进行白名单,因为第二次测量的年龄显然取决于第一次测量的年龄。
> data.frame(from = c("T1"), to = c("T2"))最后我们可以用bl和wl来学习BN的结构。
> dyn.dag
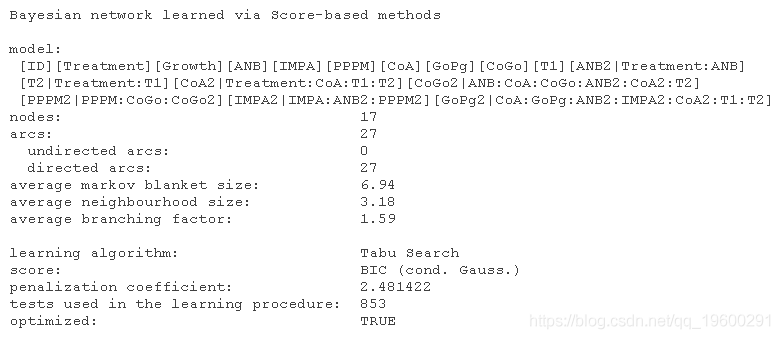
很明显,这个BN比前一个更复杂:它有更多的节点(16对9),更多的弧(27对19),因此有更多的参数(218对37)。
绘制这个新模型的最好方法是用plot()开始。
plot(dyn, render = FALSE)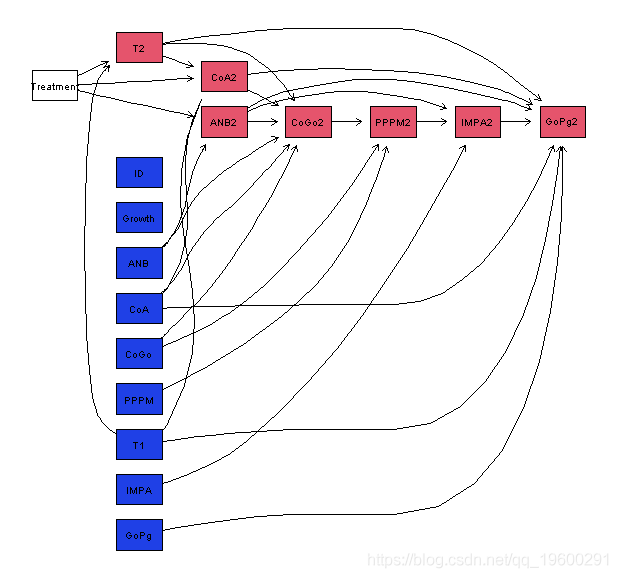
然后,我们对变量进行分组,以方便区分const、t1.variables和t2.variables;我们选择从左到右而不是从上到下绘制网络。
-
-
+ attrs = list(graph = list(rankdir = "LR")))
-
-
> Graph(gR)
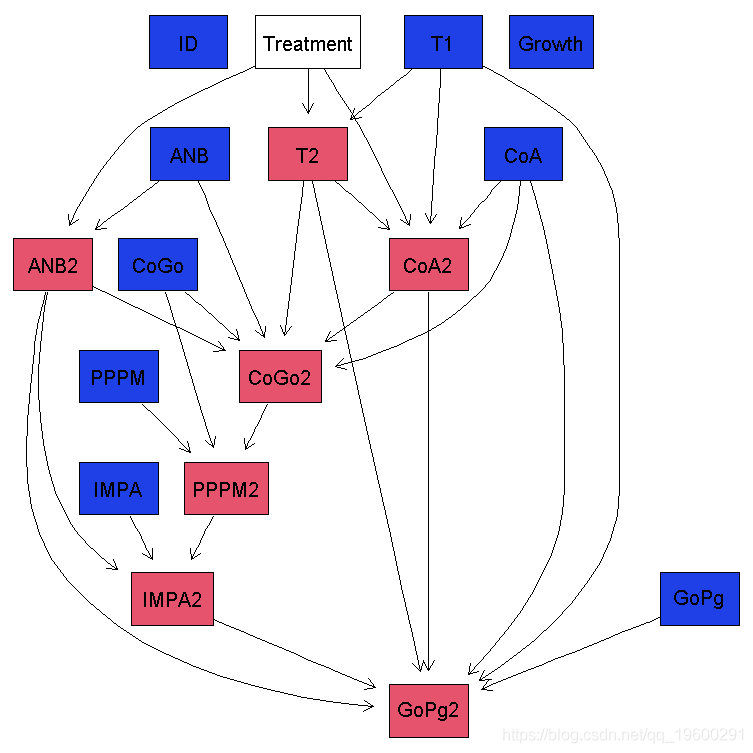
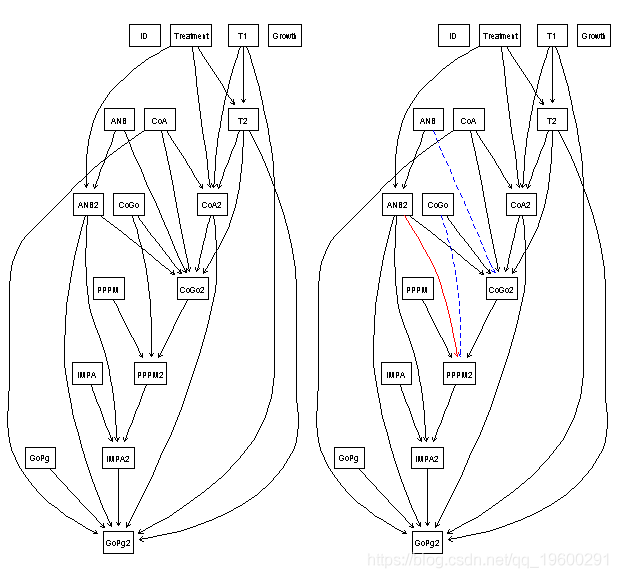
与前一个模型一样,治疗作用于ANB:从治疗出去的唯一弧是治疗→ANB2和治疗→CoA2。同样,这两个子节点都与Down的A点有关。
结构学习中的模型平均化
我们想评估这个动态BN结构的稳定性,就像我们之前对静态BN所做的那样,我们可以再次做到这一点。
-
> boot (ortho )
-
> plot(dyn)
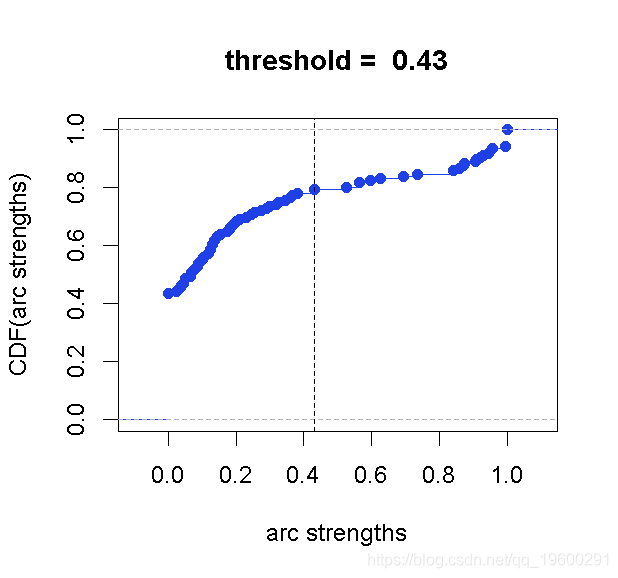
avernet(dyn.str)

平均下来的avg和dag几乎是一样的:它们只相差两道弧。这表明结构学习产生了一个稳定的输出。
-
compare(dag, avg)
-
tp fp fn
-
26 1 1
学习参数
由于Treatment是一个离散变量,BN是一个CLGBN。这意味着以Treatment为父节点的连续节点的参数化与其他节点不同。
fit(dynavg)

我们可以看到,ANB2取决于ANB(所以,在前一个时间点的同一变量)和治疗。 ANB是连续的,所以它被用作ANB2的回归因子。 治疗变量是离散的,决定了线性回归的成分。
模型验证和推理
我们可以对这个新模型提出另一组问题
- "在不同的治疗下,ANB从第一次测量到第二次测量的转变程度如何?"
我们可以用cpdist()生成一对(ANB, ANB2),条件是治疗方法等于NT、TB和TG,并观察其分布。-
data.frame(
-
diff = c(nt[, 2] - nt[, 1], tb[, 2] - tb[, 1], tg[, 2] - tg[, 1]),
-
-
> by(effect$diff, effect$treatment, FUN = mean)

density(~ diff, groups = treatment)
我们知道,治疗试图阻止ANB的下降;这与NT的分布是在TB的左边,而TB是在TG的左边这一事实相一致。未经治疗的病人病情继续恶化;治疗无效的病人没有真正改善,但他们的病情也没有恶化;而治疗有效的病人则有改善。
-
相比之下,这是一个未经治疗的病人在相同初始条件下的模拟轨迹。
对CoA的模拟轨迹是比较现实的:它随着年龄的增长而减慢。这与ANB不同,它的发生是因为CoA2同时取决于T1和T2。(ANB2则两者都不依赖)。
-
-
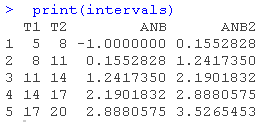
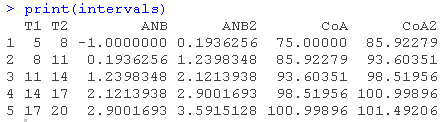
> for (i in seq(nrow(interv)) {
-
+ # 进行联合预测,目前用predict()无法实现。
-
+ dist(dyn.fitted, nodes = c(),
-
+ intervals[i,] = weighted.mean(ANB2, weights)
-
+ intervals[i,] = weighted.mean(CoA2, weights)
最受欢迎的见解
4.R语言中的block Gibbs吉布斯采样贝叶斯多元线性回归