快捷键:
1. Ctrl + Shift + F10 to restart your R session
2. Ctrl +O:打开文件
3. Ctrl + Shift+C :加标记#
4. Ctrl + Shift+N :创建新文本簿
1.r语言中= ,<-功能是相同,有时候=会出错,所以用<-。但是从字面上解释,通常“<-”被认为是赋值,”=”是传值。
2.在函数调用中,func(x=1)与func(x<-1)是有区别的,前者调用完后变量x不会被保留,而后者会在工作区里保留变量x=1。
3.初始化:
初始化
3.1使用data.frame函数就可以初始化一个Data Frame。比如我们要初始化一个student的Data Frame其中包含ID和Name还有Gender以及Birthdate,那么代码为:
student<-data.frame(ID=c(11,12,13),Name=c(“Devin”,”Edward”,”Wenli”),Gender=c(“M”,”M”,”F”),Birthdate=c(“1984-12-29”,”1983-5-6”,”1986-8-8”))
3.2另外也可以使用read.table() read.csv()读取一个文本文件,返回的也是一个Data Frame对象。读取数据库也是返回Data Frame对象。
查看student的内容为:
4.数据标准化:
scale(data, center=T,scale=T) #数据标准化
1)center和scale默认为真,即T或者TRUE
2)center为真表示数据中心化
3)scale为真表示数据标准化
读取数据:
library(xlsx)
ray = read.xlsx('F:/R编辑器/student.xlsx',1,encoding="UTF-8")
文件中含有中文,必须加上:encoding="UTF-8"。不然会出现乱码。
聚类分析:对样品或指标进行分类的一种分析方法,依据样本和指标已知特性进行分类。本节主要介绍层次聚类分析,一共包括3个部分,每个部分包括一个具体实战例子
r语言使用dist(x, method = "euclidean",diag = FALSE, upper = FALSE, p = 2) 来计算距离。其中x是样本矩阵或者数据框。method表示计算哪种距离。method的取值有:
euclidean 欧几里德距离,就是平方再开方。
maximum
切比雪夫距离
manhattan 绝对值距离
canberra Lance
距离
minkowski 明科夫斯基距离,使用时要指定p值
binary 定性变量距离.
定性变量距离: 记m个项目里面的 0:0配对数为m0 ,1:1配对数为m1,不能配对数为m2,距离=m1/(m1+m2);
diag 为TRUE的时候给出对角线上的距离。upper为TURE的时候给出上三角矩阵上的值。
r语言中使用scale(x, center =
TRUE, scale = TRUE) 对数据矩阵做中心化和标准化变换。
如只中心化 scale(x,scale=F) ,
r语言中使用sweep(x, MARGIN, STATS, FUN="-", ...) 对矩阵进行运算。MARGIN为1,表示行的方向上进行运算,为2表示列的方向上运算。STATS是运算的参数。FUN为运算函数,默认是减法。下面利用sweep对矩阵x进行极差标准化变换
|
1 2 3 |
>center <- sweep(x, 2, apply(x, 2, mean)) #在列的方向上减去均值。 >R <- apply(x, 2, max) - apply(x,2,min) #算出极差,即列上的最大值-最小值 >x_star <- sweep(center, 2, R, "/")#把减去均值后的矩阵在列的方向上除以极差向量 |
|
1 2 3 |
>center <- sweep(x, 2, apply(x, 2, min)) #极差正规化变换 >R <- apply(x, 2, max) - apply(x,2,min) >x_star <- sweep(center, 2, R, "/") |
2)层次聚类法
层次聚类法。先计算样本之间的距离。每次将距离最近的点合并到同一个类。然后,再计算类与类之间的距离,将距离最近的类合并为一个大类。不停的合并,直到合成了一个类。其中类与类的距离的计算方法有:最短距离法,最长距离法,中间距离法,类平均法等。比如最短距离法,将类与类的距离定义为类与类之间样本的最段距离。。。
r语言中使用hclust(d, method = "complete",
members=NULL) 来进行层次聚类。
其中d为距离矩阵。
method表示类的合并方法,有:
single 最短距离法
complete 最长距离法
median 中间距离法
mcquitty 相似法
average 类平均法
centroid 重心法
ward 离差平方和法
?
|
1 2 3 4 5 6 7 8 |
> x <- c(1,2,6,8,11) #试用一下 > dim(x) <- c(5,1) > d <- dist(x) > hc1 <- hclust(d,"single") > plot(hc1) > plot(hc1,hang=-1,type="tirangle") #hang小于0时,树将从底部画起。 #type = c("rectangle", "triangle"),默认树形图是方形的。另一个是三角形。 #horiz TRUE 表示竖着放,FALSE表示横着放。 7 |
1、常规聚类过程:
一、首先用dist()函数计算变量间距离
dist.r = dist(data, method=" ")
其中method包括6种方法,表示不同的距离测度:"euclidean", "maximum", manhattan",
"canberra", "binary" or "minkowski"。相应的意义自行查找。
二、再用hclust()进行聚类
hc.r = hclust(dist.r, method = “ ”)
其中method包括7种方法,表示聚类的方法:"ward", "single",
"complete","average", "mcquitty",
"median" or "centroid"。相应的意义自行查找。
三、画图
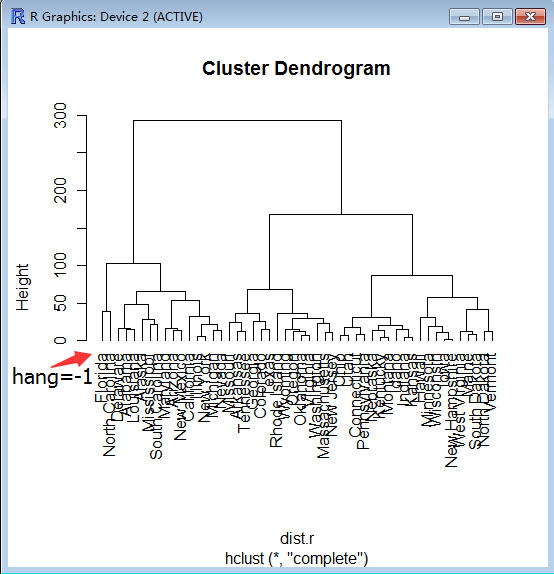
plot(hc.r, hang = -1,labels=NULL) 或者plot(hc.r, hang = 0.1,labels=F)
hang 等于数值,表示标签与末端树杈之间的距离,
若是负数,则表示末端树杈长度是0,即标签对齐。
labels 表示标签,默认是NULL,表示变量原有名称。labels=F
:表示不显示标签。
实例介绍
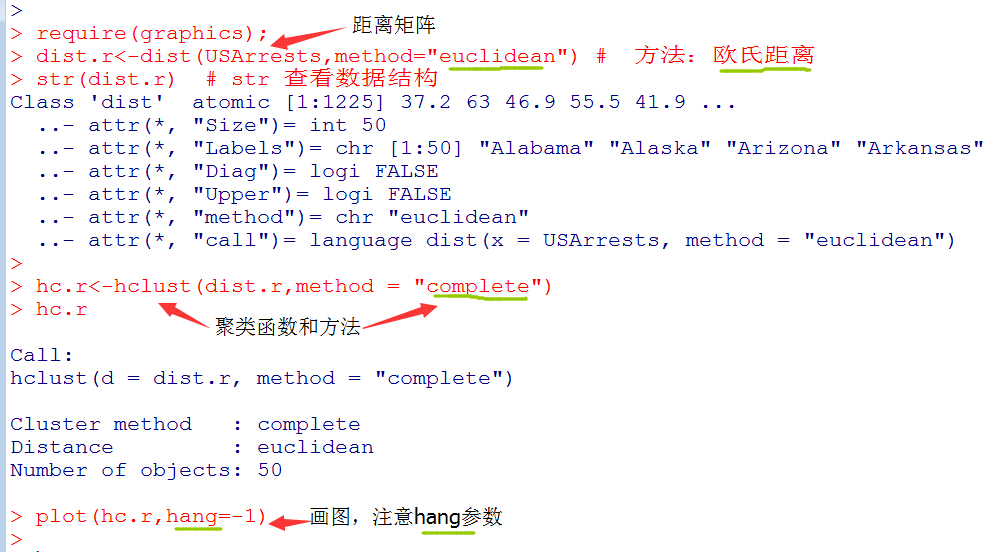
特殊情况用法:
当用已知距离矩阵进行聚类时,即变量间的距离已经计算完,只是想用
已知的距离矩阵进行聚类。这时,需将距离矩阵转成dist类型。
然后再执行hclust()聚类和plot()画图。
# mydata作为距离矩阵,且为正方矩阵
mydata<-matrix(1:25,ncol=5);
class(mydata);
# 把mydata变成dist类型
mydist<-as.dist(mydata);
class(mydist);
[1] "dist"
myhc<-hclust(mydist,method="complete");
myhc;
Call:
hclust(d = mydist, method ="complete")
Cluster method : complete
Number of objects: 5
plot(myhc,hang=0.1)
2、热图聚类过程:
一、首先用dist()函数计算变量间距离
dist.r = dist(data, method=" ")
二、用heatmap()函数进行热点图聚类
对于heatmap中具体参数,这里不做过多介绍,可在帮助文档中找说明。除此heatmap函数之外,gplots包中的heatmap.2()函数,也可以做热点图聚类。
其中参数不做过多描述。若有需求,请分享并回复:heatmap.2
即可得到答案。
实战例子:
require(graphics);
dist.r<-dist(USArrests,method="euclidean") # 方法:欧氏距离
dist.r
#聚类并画图
heatmap(as.matrix(dist.r))
3、多维标度和聚类的结果
MDS方法对距离矩阵进行降维,用不同的颜色来表示聚类的结果。
另一种聚类效果展示。
例子:

系统聚类:
library(xlsx)
mydata=read.xlsx('F:/R编辑器/student.xlsx',1,encoding="UTF-8")
head(mydata)
rownames(mydata)=mydata$科目
mydata=mydata[,-1]
head(mydata)
result=dist(mydata, method = "euclidean")
m=c("complete")
h=list();par(mfrow=c(2,4))
for(i in 1:length(m)){
h[[i]]=hclust(dist(scale(mydata)),method =m[i])
plot(h[[i]],lables=rownames(mydata),cex=2,
main=paste("Method: ",m[i]))
}
5、对普通的画图为:
plot(number, count, yaxt="n", xaxt="n",xlab="x 轴名字",type="b")
上述表示为:yaxt xaxt都是将数据轴的标度和标度值完全去掉,但是可以设置轴的名字,后面设置标度和标度值使用:
axis(side=1, at=c(1,3,5,6,7), lables=(ni, hao, a , man, go));等均可匹配,at表示刻度值范围。
但是有个问题就是太拥挤的不可见怎么缩小y轴的尺寸,而缩小尺寸值是在axis()命令设置at这个参数的时候就设置,使用cex.axis=0.5就表示缩小一半。而当直接使用默认的坐标刻度的时候,那么就在plot中直接放cex.axis=0.5即可。同样两个地方可以用的有:cex.lab=0.5 只缩放坐标轴的名称。 cex.main=0.只缩放主标题 cex.sub只缩放副标题。
当然上述除了axis要设置at值,还有par函数,但是该函数主要是作用于整个画布环境,设置完后,不会对当前已经显示的影响,而只会影响后面重新生成的图片。可以设置,par(cex.axis=0.5, )
其中设置画布大小的为:par(pin(11,7))是目前自己试出来的最大画布。
runif()函数,随机数的产生,均匀分布随机数
R语言生成均匀分布随机数的函数是runif()
句法是:runif(n,min=0,max=1) n表示生成的随机数数量,min表示均匀分布的下限,max表示均匀分布的上限;若省略参数min、max,则默认生成[0,1]上的均匀分布随机数。
正态分布随机数的生成函数是 rnorm()
句法是:rnorm(n,mean=0,sd=1) 其中n表示生成的随机数数量,mean是正态分布的均值,默认为0,sd是正态分布的标准差,默认时为1;
五、apply()函数
apply函数族是R语言中数据处理的一组核心函数,通过使用apply函数,我们可以实现对数据的循环、分组、过滤、类型控制等操作。
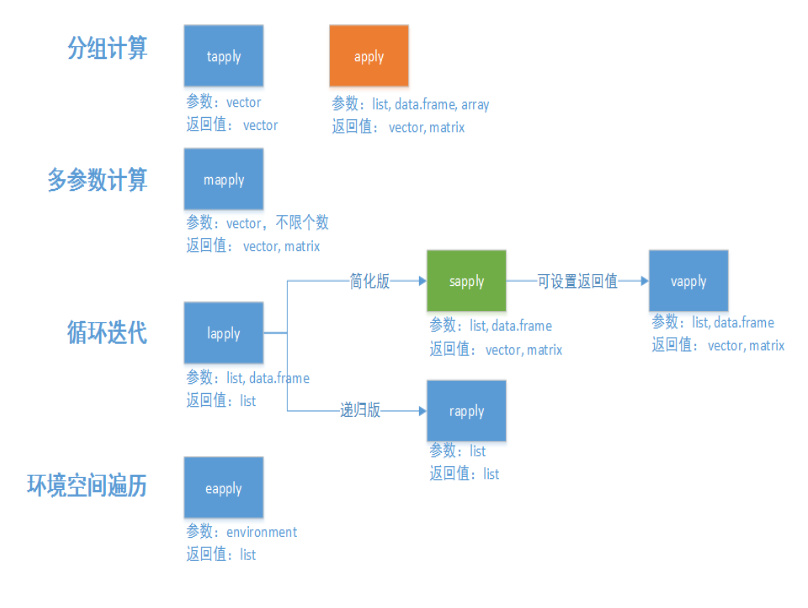
apply函数是最常用的代替for循环的函数。apply函数可以对矩阵、数据框、数组(二维、多维),按行或列进行循环计算, 对子元素进行迭代,并把子元素以参数传递的形式给自定义的FUN函数中,并以返回计算结果。
apply(X, MARGIN, FUN, ...)
参数列表:
- X:数组、矩阵、数据框
- MARGIN: 按行计算或按按列计算,1表示按行,2表示按列
- FUN: 自定义的调用函数
- …: 更多参数,可选.例:apply(X,2,sum)