这篇文章很不错:https://blog.csdn.net/u013082989/article/details/53792010
为什么数据处理之前要进行归一化???(这个一直不明白)
这个也很不错:https://blog.csdn.net/u013082989/article/details/53792010#commentsedit
下面是复现一个例子:
# -*- coding: utf-8 -*-
#来源:https://blog.csdn.net/u013082989/article/details/53792010
#来源:https://blog.csdn.net/hustqb/article/details/78394058 (这里有个例子)关于降维之后的坐标系问题,???结合里面的例子
#用库函数实现的过程:
#导入需要的包:
import numpy as np
from matplotlib import pyplot as plt
from scipy import io as spio
from sklearn.decomposition import pca
from sklearn.preprocessing import StandardScaler
import matplotlib.pyplot as plt
#归一化数据,并作图
def scaler(X):
"""
注:这里的归一化是按照列进行的。也就是把每个特征都标准化,就是去除了单位的影响。
"""
scaler=StandardScaler()
scaler.fit(X)
x_train=scaler.transform(X)
return x_train
#使用pca模型拟合数据并降维n_components对应要降的维度
def jiangwei_pca(x_train,K): #传入的是X的矩阵和主成分的个数K
model=pca.PCA(n_components=K).fit(x_train)
Z=model.transform(x_train) #transform就会执行降维操作
#数据恢复,model.components_会得到降维使用的U矩阵
Ureduce=model.components_
x_rec=np.dot(Z,Ureduce) #数据恢复
return Z,x_rec #这里Z是将为之后的数据,x_rec是恢复之后的数据。
if __name__ == '__main__':
X=np.array([[1,1],[1,3],[2,3],[4,4],[2,4]])
x_train=scaler(X)
print('x_train:',x_train)
Z,x_rec=jiangwei_pca(x_train,2)
print("Z:",Z)
print("x_rec:",x_rec) #如果有时候,这里不能够重新恢复x_train,一个原因可能是主成分太少。
print("x_train:",x_train)
## 这里的主成分为什么不是原来的两个。
## 还有一个问题是,如何用图像表现出来。
## 还有一个问题就是如何得到系数,这个系数是每个特征在主成分中的贡献,要做这个就需要看矩阵,弄明白原理:
或许和这个程序有关:pca.explained_variance_ratio_
摘自:https://blog.csdn.net/qq_36523839/article/details/82558636
这里提一点:pca的方法explained_variance_ratio_计算了每个特征方差贡献率,所有总和为1,explained_variance_为方差值,通过合理使用这两个参数可以画出方差贡献率图或者方差值图,便于观察PCA降维最佳值。
在提醒一点:pca中的参数选项可以对数据做SVD与归一化处理很方便,但是需要先考虑是否需要这样做。
关于pca的一个推导例子:

、、
根据最后的图形显示来看,一共有五个样本点。而从下面的矩阵看,似乎不是这样???
有点纠结。
从对矩阵X的求均值过程可以知道,是对行求均值的。然后每行减掉均值。
(这样的话,也就是说:每一行是一个特征???,就不太明白了。)
应该写成这样比较清楚:(每一列是一个特性)
[
[1,1]
[1,3]
[2,3]
[4,4]
[2,4]
]
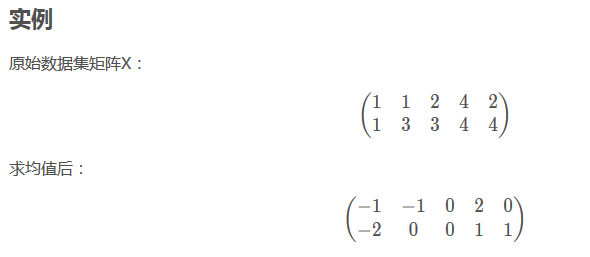
、、
从下面看出这里除的是5,也就是说5是m,也就是行数。???
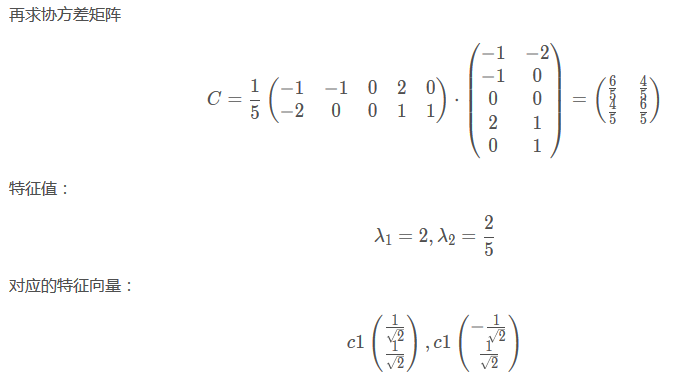
、、
最后降维到一个特征::
下面图片中P的部分,是两个数,也就是两个特征的系数。代表着特征的系数。。。
关键是用的别人的库,但是怎么弄???

、、
上面
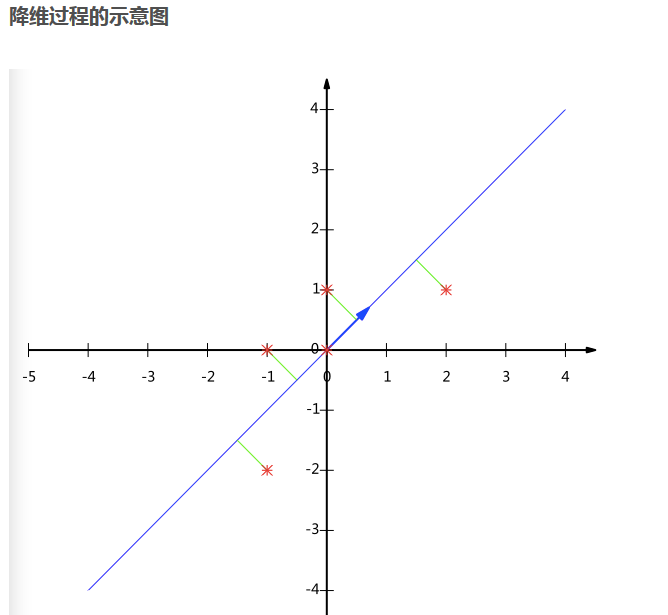
#、、、、、、、、、、、、、、、、、、、、、、、、、、、、、、、、、、、、、、、、、、、、、、、、、、、、
下面我们来分析另一个例子:这个例子是官方给出的:
程序如下:
# -*- coding: utf-8 -*-
"""
测试
这里是Python的pca主成分分析的一个测试程序
"""
import numpy as np
from sklearn.decomposition import PCA
X = np.array([[-1, -1], [-2, -1], [-3, -2], [1, 1], [2, 1], [3, 2]])
pca = PCA(n_components='mle') #这里是让机器决定主成分的个数,我们也可以自行设置。
pca = PCA(n_components=2) #这里设置主成分为,这里不能设置成3,因为这里的特征本身只有两个。
pca.fit(X)
print("这里是X:")
print(X)
Z=pca.transform(X) #transform就会执行降维操作
print('这里是Z:')
print(Z)
# Z = np.dot(X, self.components_.T)
# PCA(copy=True, n_components=2, whiten=False)
print(pca.explained_variance_ratio_)
然后运行程序输出的结果:
这里是X:
[[-1 -1]
[-2 -1]
[-3 -2]
[ 1 1]
[ 2 1]
[ 3 2]]
可能是系数的东西: 这里有可能是没个主成分中包含各个特征的权重系数。
你有没有感觉到这个矩阵有一定的特性,有点对角线对称的样子。
[[-0.83849224 0.54491354]
[-0.54491354 -0.83849224]]
这里是Z: 这里的Z实际上主成分的意思。主成分也就是综合特征
[[ 1.38340578 0.2935787 ]
[ 2.22189802 -0.25133484]
[ 3.6053038 0.04224385]
[-1.38340578 -0.2935787 ]
[-2.22189802 0.25133484]
[-3.6053038 -0.04224385]]
[0.99244289 0.00755711]
要捋清一个问题,我们想要得到的是什么?
我们想要得到的是每个主成分前面包含特征的系数。
主成分1=权重11*特征1+权重12*特征2+权重13*特征3···
主成分2=权重21*特征1+权重22*特征2+权重23*特征3···
[[-0.83849224 0.54491354]
[-0.54491354 -0.83849224]]
主成分1=(-0.83849224) *特征1+(-0.54491354)*特征2···
主成分2=(0.54491354) *特征1+(-0.83849224)*特征2···
就是上面这种系数,
我还是有一点疑问?为什么?这个系数矩阵是对称的,这样有点不是很科学??