同模拟退火算法一样,都是现代优化算法之一。模拟退火是在一定接受程度的情况下仍然接受一个比较差的解。
遗传算法,是真真正正的和大自然的遗传进化有着非常紧密的联系的,当然遗传进化的只是在生物学中已经讲过了,8个字,物竞天择,适者生存。
-
简介
《物种起源》,有兴趣可以看看达尔文的著作。
物竞天择,适者生存,这两句话,也可以说是对遗传算法过程的伪代码描述了,物竞天择,就是我们的目标函数,只有越满足我们的目标函数的个体才会留下来,适者生存,就是我们在算法的过程中要淘汰一些个体。
-
基因编码方式
生物学里面告诉我们,遗传,变异都是以种群为研究对象的,怎么表示一个解呢?用他的基因表示,嘿嘿,组成这个解的步骤表示,第一步干什么,第二步干什么,怎么在程序中编码呢?
常用两种编码方式:二进制编码,浮点数编码。
二进制编码:一定精度的二进制只能表示一定精度的浮点数。栗子,要求精确到6位小数,而区间是 [-1,2],至少要把区间划分为3*10^6等分。编码也就需要22位。这里就涉及到一个二进制串转换到一个区间为 [-1,2] 的实数,两者相互转换。比如得到的十进制数为 x,那么对应的 [-1,2] 区间的浮点数就是
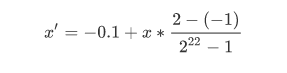
浮点数编码:为改善遗传算法的复杂度,提出来浮点数编码
-
适应评分及选择函数
适应评分函数就是用来衡量哪个个体应该被淘汰。但是也不能说,取值差点的个体一定就被淘汰了,这里会有一个概率存在,怎么建立一种概率关系呢?常用的方法是轮盘法。假设种群数为 n,某个个体 i 的适应度为 fi,那么个体 i 被选择的概率是:
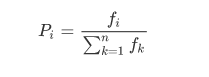
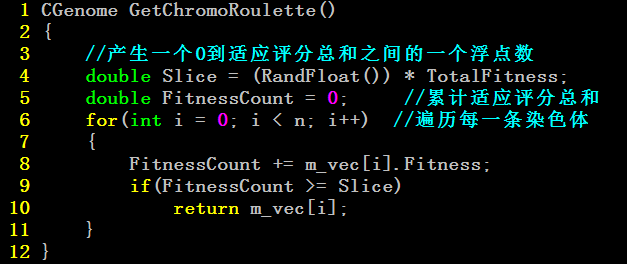
So,遗传算法中的自然选择过程:
适应评分函数——求出各个体的适应评分值
轮盘选择——个体被选择的概率

-
基因重组与基因突变
在生物学中,基因重组有两种情况。
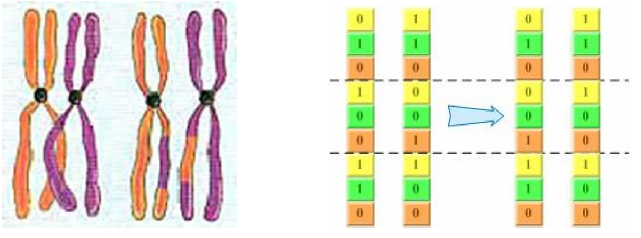

基因重组,就是两个个体基因发生交换。
基因突变是一种小概率事件。
使得一个基因变成他的等位基因,引起一定的表现型变化。

-
TSP问题求解
TSP问题,之前已经用费用流,和模拟退火做过了,现在用遗传来讨论。
程序过程和自然界中的遗传与进化是一样的。
-
生成50个个体,形成种群,并基因编码。
-
基因重组,突变,和父代,一起进行天择。
-
重复100次进化。得到较优的解。
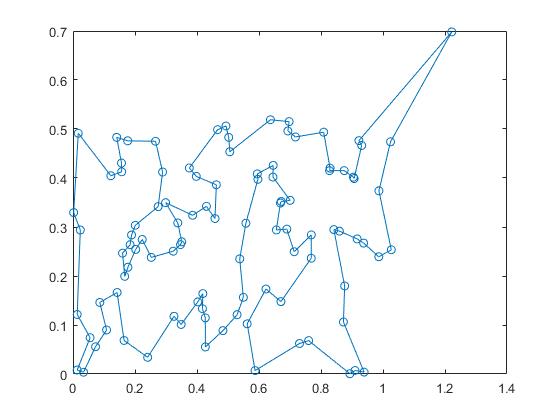

clc,clear; sj0 = load('sj.txt'); x = sj0(:,1:2:8); x = x(:); y = sj0(:,2:2:8); y = y(:); sj = [x,y];d1 = [70,40]; sj = [d1;sj;d1]; sj = sj*pi/180; d = zeros(102); for i = 1:101 for j = i+1:102 d(i,j) = 6370*acos(cos(sj(i,1)-sj(j,1))*cos(sj(i,2))*cos(sj(j,2))+sin(sj(i,2))*sin(sj(j,2))); end end d = d + d'; % w 为种群数,g为进化的代数 w = 50; g = 100; rand('state',sum(clock)); % 改良圈算法选取初始种群 for k=1:w %通过改良圈算法选取初始种群 c=randperm(100); %产生1,...,100的一个全排列 c1=[1,c+1,102]; %生成初始解 for t=1:102 %该层循环是修改圈 flag=0; %修改圈退出标志 for m=1:100 for n=m+2:101 if d(c1(m),c1(n))+d(c1(m+1),c1(n+1))<d(c1(m),c1(m+1))+d(c1(n),c1(n+1)) c1(m+1:n)=c1(n:-1:m+1); flag=1; %修改圈 end end end if flag==0 J(k,c1)=1:102; break %记录下较好的解并退出当前层循环 end end end % 染色体编码 J(:,1) = 0; J = J/102; % 100次进化 for k = 1:g A = J; c = randperm(w); for i = 1:2:w % 基因重组 F = 2 + floor(100*rand(1)); % 产生交叉操作的染色体对 tmp = A(c(i),[F:102]); A(c(i),[F:102]) = A(c(i+1),[F:102]); A(c(i+1),F:102) = tmp; end % 变异 by = []; while ~isempty(by) % 变异的个体数也是随机的 by =find(rand(1,w)<0.1); end B = A(by,:); % 变异染色体 for j = 1:length(by) bw = sort(2+floor(100*rand(1,3))); % 产生变异操作的3个地址 B(j,:) = B(j,[1:bw(1),bw(2)+1:bw(3),bw(1):bw(2),bw(3)+1:102]); end G=[J;A;B]; % 父代和子代 % 基因翻译为解空间,把染色体翻译成1,...,102的序列ind1 [SG,ind1] = sort(G,2); num = size(G,1); % 父子种群的总个体数 long = zeros(1,num); % 每一个体的优劣 for j = 1:num for i = 1:101 long(j) = long(j)+d(ind1(j,i),ind1(j,i+1)); end end [slong,ind2] = sort(long); J = G(ind2(1:w),:); end path = ind1(ind2(1),:); flong = slong(1); xx = sj(path,1); yy = sj(path,2); plot(xx,yy,'-o');
参考:
ACdreamer
司守奎