转自:https://blog.csdn.net/sinat_38163598/article/details/72851239
1.基本
GFF和GTF是两种最常用的数据库注释格式,基因注释文件。
GFF全称为general feature format,这种格式主要是用来注释基因组。
GTF全称为gene transfer format,主要是用来对基因进行注释,对染色体上的基因进行标注。
//我这里关注的主要是GTF文件。
2.格式
以tab键分割为9列:
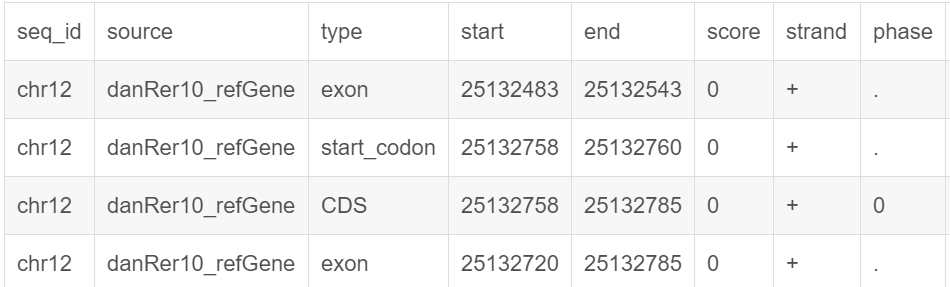
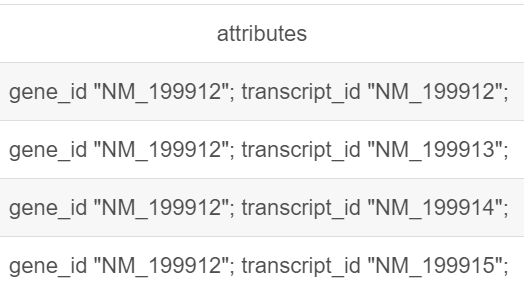
- seq_id:序列的编号;
- source: 注释的来源,一般为数据库或者注释的机构;
- type: 注释信息的类型,比如Gene、cDNA、mRNA、CDS等
- start:该基因或转录本在参考序列上的起始位置
- end: 该基因或转录本在参考序列上的终止位置
- score: 得分,数字,是注释信息可能性的说明
- strand: 该基因或转录本位于参考序列的正链(+)或负链(-)上;
3.读取gtf格式文件查看
https://www.jianshu.com/p/a5a23f926931,按照这个方法读取时遇到了问题:

尝试用read.csv打开也失败:
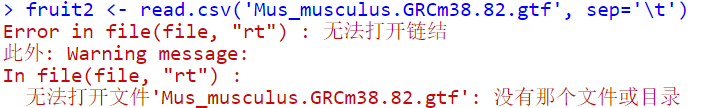
太不顺利了吧,这都能被卡住,
https://www.gitmemory.com/issue/kvittingseerup/IsoformSwitchAnalyzeR/83/719484994,这个链接中提出来的问题和我的类似,也是需要读取到gtf文件,它的R版本是4.0.3,我的也是4.0版本的R,所以是否有可能是R版本的问题导致对应包中的函数不可用了?
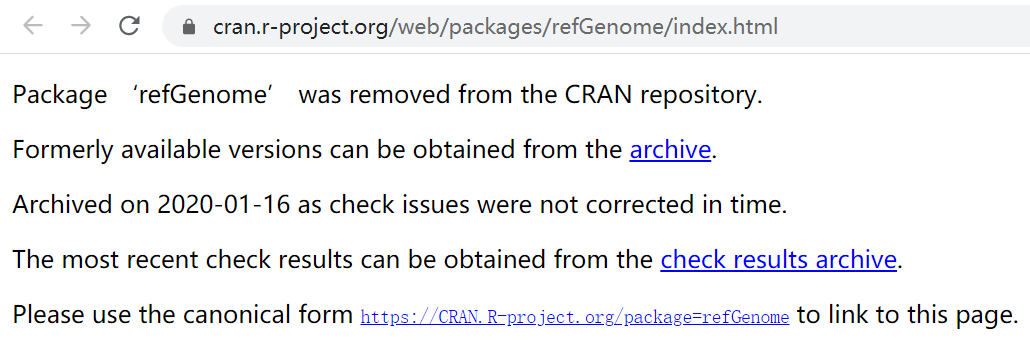
https://hwoihann.github.io/farnorth/analysis/2018/03/30/R-gtf-refGenome.html,这个链接中提到可以使用refGenome,但是
https://cran.r-project.org/web/packages/refGenome/index.html,发现已经被移除。
使用
install.packages("refGenome") BiocManager::install("refGenome")
安装,均不可。
可能性2:gtf文件存在问题,所以重新下载。
https://www.gencodegenes.org/mouse/,在这里下载了最新版的,并且解压:
并且我尝试导入
library(rtracklayer)
报出以下错误:

https://stackoverflow.com/questions/63958678/error-in-unloadnamespacepackage-namespace-rlang-is-imported-by-testthat,搜索相似问题,其中一个回复:

之后我就尝试在命令行而不是Rstudio上操作,先卸载了这个包,然后安装,虽然出现了这个问题:
Installation path not writeable, unable to update packages: codetools,但是我尝试不更新n所有包,之后library居然可以了,而且可以正常读取。
之后我重启Rstudio
.rs.restartR()
就可以读取gtf文件了。
看来以后安装包的操作都应该在命令行进行,而不是Rstudio。
4.查看GTF文件内容
https://www.jianshu.com/p/a5a23f926931,基序按照这个来读取
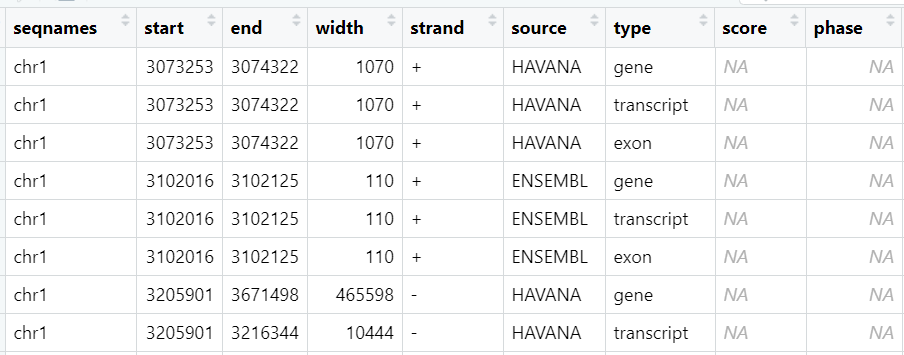
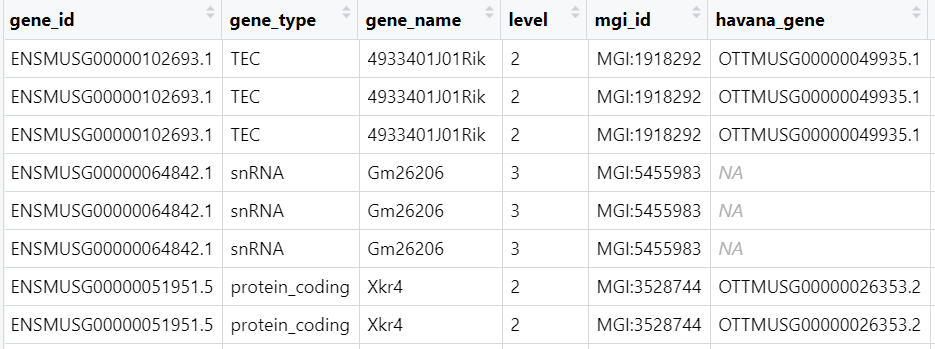


最新版的相较于之前有了很多新的描述信息,比如基因id,基因名称等等。共有26个特征。