如何计算每个基因的覆盖度与深度,有多种方法可以完成。如下演示使用samtools depth命令方法
1. 数据下载
1.1 Fastq文件下载
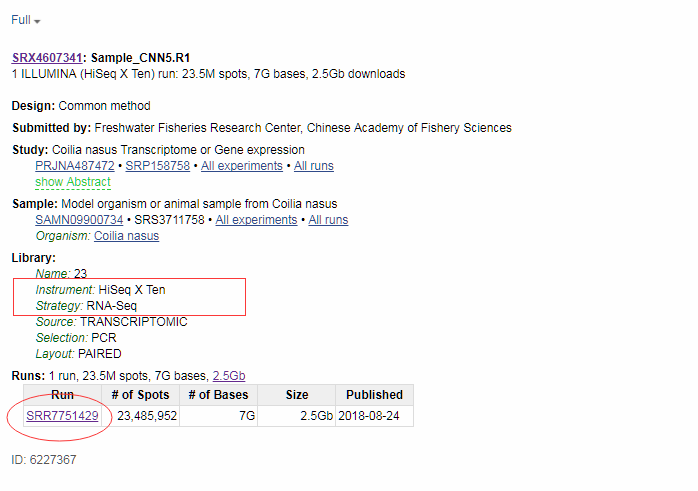
从NCBI下载Illumina Hiseq X Ten平台的RNA-Seq数据SRR7751429信息如上图所示。
1.1.1 使用wget命令(sra-toolkit工具下载太慢)下载
wget ftp://ftp.ncbi.nlm.nih.gov/sra/sra-instant/reads/ByRun/sra/SRR/SRR775/SRR7751429/SRR7751429.sra
1.1.2 在SRA Toolkit工具页面根据不同操作系统进行下载(例如,我的是编译好的Centos 64位)
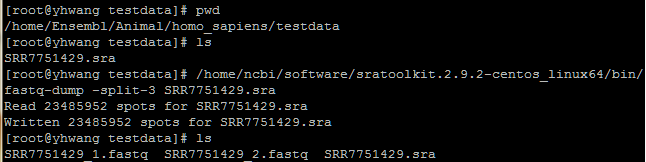
1.1.3 使用SRA toolkit工具将SRR7751429.sra数据转成fastq格式
fastq-dump -split-3 SRR7751429.sra
1.2 基因组及注释文件下载
人的参考基因组文件(版本GRCh38)下载
wget ftp://ftp.ensembl.org/pub/release-93/fasta/homo_sapiens/dna/Homo_sapiens.GRCh38.dna.toplevel.fa.gz
# 解压
gunzip Homo_sapiens.GRCh38.dna.toplevel.fa.gz
人的gtf注释文件下载
wget ftp://ftp.ensembl.org/pub/release-93/gtf/homo_sapiens/Homo_sapiens.GRCh38.93.gtf.gz
# 解压
gunzip Homo_sapiens.GRCh38.93.gtf.gz
2. 生成bam文件
因为是演示,只需要生成bam文件,我这里就用bwa比对了,节约时间。
# 创建索引 bwa index /home/Ensembl/Animal/homo_sapiens/Homo_sapiens.GRCh38.dna.all.fa
# 比对
bwa mem -t 32 -M /home/Ensembl/Animal/homo_sapiens/Homo_sapiens.GRCh38.dna.all.fa SRR7751429_1.fastq SRR7751429_2.fastq -o SRR7751429.sam
3. 基因CDS(编码区)获取
3.1 本地获取基因cds信息
下载的Homo_sapiens.GRCh38.93.gtf文件包含有基因exon、cds、3'utr、5'utr等相关的物理位置信息,获取基因CDS信息只需解析该文件就可以了。(有需要的话,后续跟新相关脚本)
3.2 使用ensembl获取cds信息
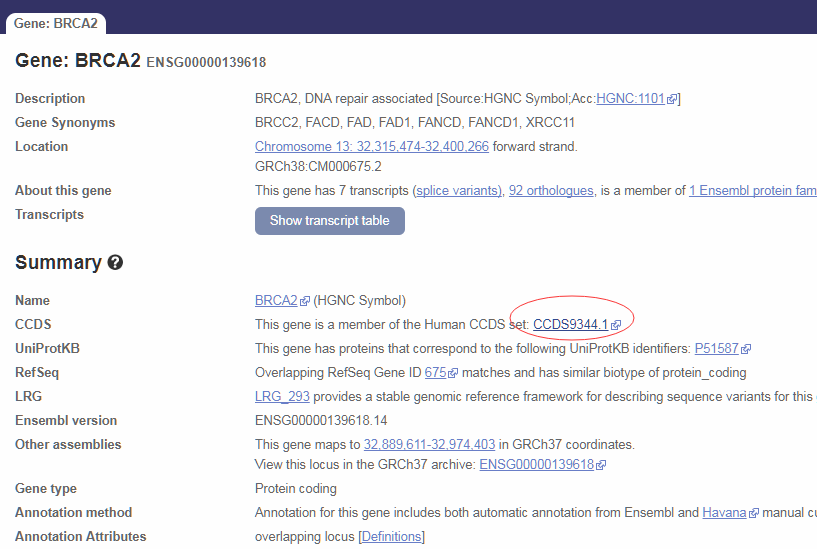
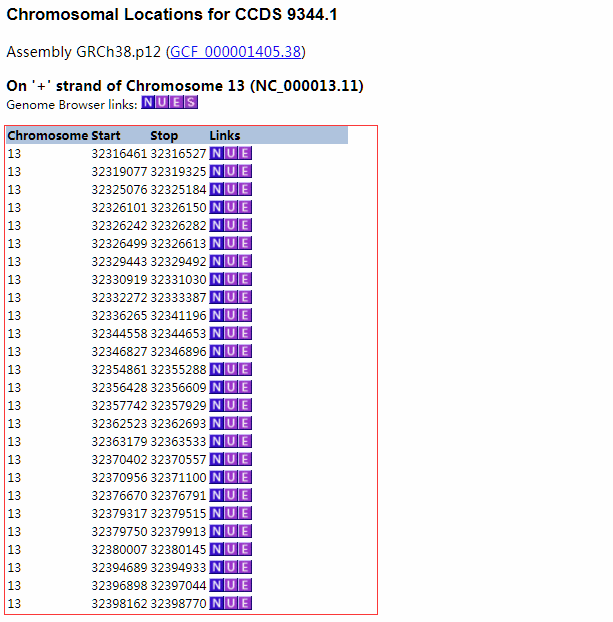
如上图所示,以人BRCA2基因为例,搜到后点击CCDS,出现该基因的物理位置信息。然后,将该信息复制粘贴,以如下图所示格式储存于文件BRCA2.bed中。
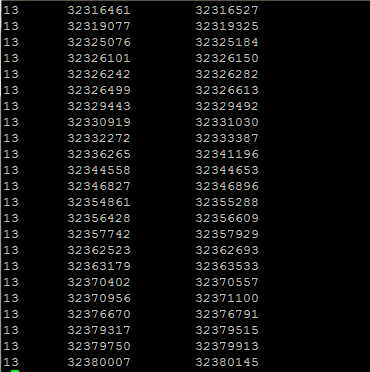
4. 使用samtools工具进行统计
samtools工具是对SAM/BAM文件进行操作的软件,其带有多种统计相关的命令及SAM↔BAM格式转换的命令。
4.1 SAM文件格式转换为BAM文件格式
samtools view -@ 16 -bS SRR7751429.sam -o SRR7751429.bam
4.2 sort BAM文件,然后建立BAM文件索引
# sort BAM文件
samtools sort -@ 16 -o SRR7751429_sorted.bam SRR7751429.bam
# 索引BAM文件
samtools index -@ 16 SRR7751429_sorted.bam
4.3 使用depth命令计算bed文件区域中每个位点的深度
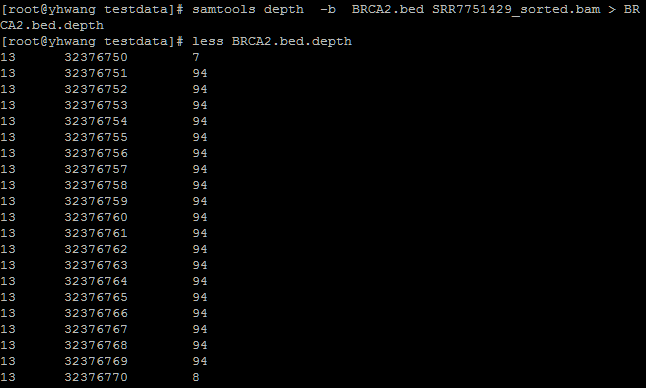
samtools depth -b BRCA2.bed SRR7751429_sorted.bam >BRCA2.bed.depth
一共得到3列以指标分隔符分隔的数据,第一列为染色体名称,第二列为位点,第三列为覆盖深度。
4.4 根据BED文件和深度文件来统计大于10×的区域占总CDS区域比例
# -*- coding: utf-8 -*-
from __future__ import division
import csv
# 定义cds文件名路径
cdsfh = 'BRCA2.bed'
# 区域长度
cdslen = 0
with open(cdsfh, 'r') as f:
cf = csv.reader(f, dialect='excel-tab')
for row in cf:
# 读取每一行区域
chrom, start, end = row
length = int(end) - int(start) + 1
# 迭代所有的cds区域长度,得到基因cds区域全长
cdslen += length
# 定义深度文件名路劲
depthfh = 'BRCA2.bed.depth'
# 大于10X区域长度
gt10len = 0
with open(depthfh, 'r') as f:
cf = csv.reader(f, dialect='excel-tab')
for row in cf:
# 读取每一行区域
chrom, pos, depth = row
# 判断覆盖度是否大于10X,是的gt10len就自增1
if int(depth) > 10: gt10len += 1
# 计算编码区大于10X的区域占总编码区的比例
percent = gt10len / cdslen * 100
# 输出
print("%.2f%%" % percent)
上述脚本只能针对单个基因,若是多个基因,可结合shell循环实现。