Python对Dicom图像进行处理,离不开pydicom,opencv-python,matplotlib,numpy四个代码库,安装完成这四个代码库后,
可以读取Dicom图像,并对图像进行处理,显式处理后的结果,下面就举例说明:
import cv2 import numpy import dicom from matplotlib import pyplot as plt # 读取单张Dicom图像 dcm = dicom.read_file("../Data/vhm.420.dcm") dcm.image = dcm.pixel_array * dcm.RescaleSlope + dcm.RescaleIntercept # 获取图像中的像素数据 slices = [] slices.append(dcm) # 复制Dicom图像中的像素数据 img = slices[ int(len(slices)/2) ].image.copy() # 对图像进行阈值分割 ret,img = cv2.threshold(img, 90,3071, cv2.THRESH_BINARY) img = numpy.uint8(img) # 提取分割结果中的轮廓,并填充孔洞 im2, contours, _ = cv2.findContours(img,cv2.RETR_LIST,cv2.CHAIN_APPROX_SIMPLE) mask = numpy.zeros(img.shape, numpy.uint8) for contour in contours: cv2.fillPoly(mask, [contour], 255) img[(mask > 0)] = 255 # 对分割结果进行形态学的开操作 kernel = cv2.getStructuringElement(cv2.MORPH_ELLIPSE,(2,2)) img = cv2.morphologyEx(img, cv2.MORPH_OPEN, kernel) # 根据分割mask获取分割结果的像素数据 img2 = slices[ int(len(slices)/2) ].image.copy() img2[(img == 0)] = -2000 # 显式原始数据,mask和分割结果 plt.figure(figsize=(12, 12)) plt.subplot(131) plt.imshow(slices[int(len(slices) / 2)].image, 'gray') plt.title('Original') plt.subplot(132) plt.imshow(img, 'gray') plt.title('Mask') plt.subplot(133) plt.imshow(img2, 'gray') plt.title('Result') plt.show()
运行后的结果:
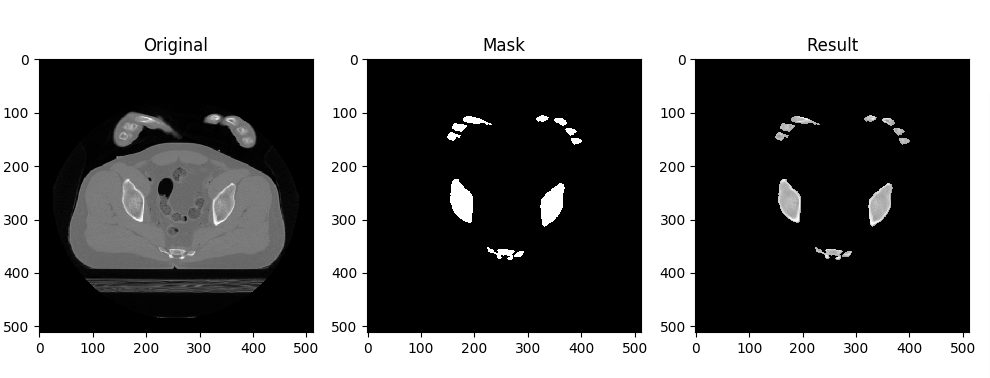
本例子以单张Dicom图像为例,分割图像中的骨骼信息,也可以同时读取多张Dicom图像,分割其他组织器官。