无论是ICF基于物品的协同过滤、UCF基于用户的协同过滤、基于内容的推荐,最基本的环节都是计算相似度。如果样本特征维度很高或者<user, item, score>的维度很大,都会导致无法直接计算。设想一下100w*100w的二维矩阵,计算相似度怎么算?
更多内容参考——我的大数据学习之路——xingoo
在spark中RowMatrix提供了一种并行计算相似度的思路,下面就来看看其中的奥妙吧!
相似度
相似度有很多种,每一种适合的场景都不太一样。比如:
- 欧氏距离,在几何中最简单的计算方法
- 夹角余弦,通过方向计算相似度,通常在用户对商品评分、NLP等场景使用
- 杰卡德距离,在不考虑每一样的具体值时使用
- 皮尔森系数,与夹角余弦类似,但是可以去中心化。比如评分时,有人倾向于打高分,有人倾向于打低分,他们的最后效果在皮尔森中是一样的
- 曼哈顿距离,一般在路径规划、地图类中常用,比如A*算法中使用曼哈顿来作为每一步代价值的一部分(F=G+H, G是从当前点移动到下一个点的距离,H是距离目标点的距离,这个H就可以用曼哈顿距离表示)
在Spark中使用的是夹角余弦,为什么选这个,道理就在下面!
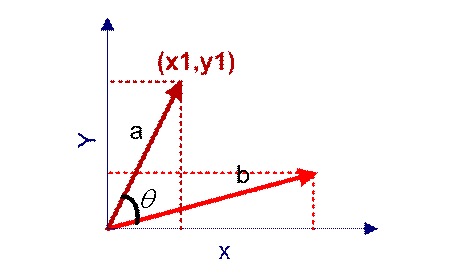
上面两个向量
和
计算其夹角的余弦值就是两个向量方向的相似度。
公式为:
其中,(||a||)表示a的模,即每一项的平方和再开方。
公式拆解
那么如果向量不只是两维,而是n维呢?比如有两个向量:
他们的相似度计算方法套用上面的公式为:
通过上面的公式就可以发现,夹角余弦可以拆解成每一项与另一项对应位置的乘积({ x }_{ 1 }ast { y }_{ 1 }),再除以每个向量自己的
就可以了。
矩阵并行
画个图看看,首先创建下面的矩阵:
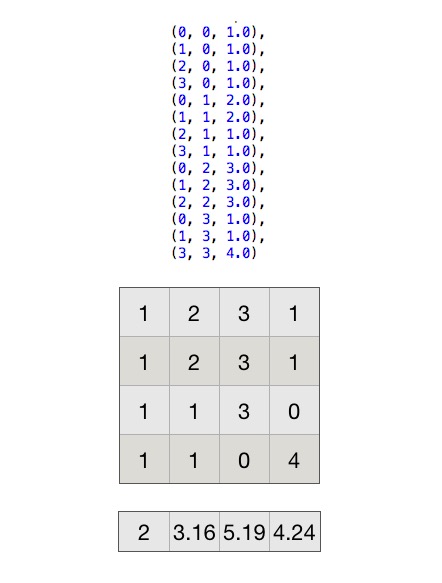
注意,矩阵里面都是一列代表一个向量....上面是创建矩阵时的三元组,如果在spark中想要创建matrix,可以这样:
val df = spark.createDataFrame(Seq(
(0, 0, 1.0),
(1, 0, 1.0),
(2, 0, 1.0),
(3, 0, 1.0),
(0, 1, 2.0),
(1, 1, 2.0),
(2, 1, 1.0),
(3, 1, 1.0),
(0, 2, 3.0),
(1, 2, 3.0),
(2, 2, 3.0),
(0, 3, 1.0),
(1, 3, 1.0),
(3, 3, 4.0)
))
val matrix = new CoordinateMatrix(df.map(row => MatrixEntry(row.getAs[Integer](0).toLong, row.getAs[Integer](1).toLong, row.getAs[Double](2))).toJavaRDD)
然后计算每一个向量的normL2,即平方和开根号。
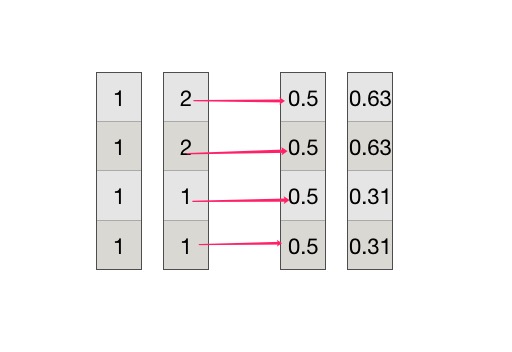
以第一个和第二个向量计算为例,第一个向量为(1,1,1,1),第二个向量为(2,2,1,1),每一项除以对应的normL2,得到后面的两个向量:
两个向量最终的相似度为0.94。
那么在Spark如何快速并行处理呢?通过上面的例子,可以看到两个向量的相似度,需要把每一维度乘积后相加,但是一个向量一般都是跨RDD保存的,所以可以先计算所有向量的第一维,得出结果
最后对做一次reduceByKey累加结果即可.....
阅读源码
首先创建dataframe形成matrix:
import org.apache.spark.mllib.linalg.distributed.{CoordinateMatrix, MatrixEntry}
import org.apache.spark.sql.SparkSession
object MatrixSimTest {
def main(args: Array[String]): Unit = {
// 创建dataframe,转换成matrix
val spark = SparkSession.builder().master("local[*]").appName("sim").getOrCreate()
spark.sparkContext.setLogLevel("WARN")
import spark.implicits._
val df = spark.createDataFrame(Seq(
(0, 0, 1.0),
(1, 0, 1.0),
(2, 0, 1.0),
(3, 0, 1.0),
(0, 1, 2.0),
(1, 1, 2.0),
(2, 1, 1.0),
(3, 1, 1.0),
(0, 2, 3.0),
(1, 2, 3.0),
(2, 2, 3.0),
(0, 3, 1.0),
(1, 3, 1.0),
(3, 3, 4.0)
))
val matrix = new CoordinateMatrix(df.map(row => MatrixEntry(row.getAs[Integer](0).toLong, row.getAs[Integer](1).toLong, row.getAs[Double](2))).toJavaRDD)
// 调用sim方法
val x = matrix.toRowMatrix().columnSimilarities()
// 得到相似度结果
x.entries.collect().foreach(println)
}
}
得到的结果为:
MatrixEntry(0,3,0.7071067811865476)
MatrixEntry(0,2,0.8660254037844386)
MatrixEntry(2,3,0.2721655269759087)
MatrixEntry(0,1,0.9486832980505139)
MatrixEntry(1,2,0.9128709291752768)
MatrixEntry(1,3,0.596284793999944)
直接进入columnSimilarities方法看看是怎么个流程吧!
def columnSimilarities(): CoordinateMatrix = {
columnSimilarities(0.0)
}
内部调用了带阈值的相似度方法,这里的阈值是指相似度小于该值时,输出结果时,会自动过滤掉。
def columnSimilarities(threshold: Double): CoordinateMatrix = {
//检查参数...
val gamma = if (threshold < 1e-6) {
Double.PositiveInfinity
} else {
10 * math.log(numCols()) / threshold
}
columnSimilaritiesDIMSUM(computeColumnSummaryStatistics().normL2.toArray, gamma)
}
这里的gamma用于采样,具体的做法咱们来继续看源码。然后看一下computeColumnSummaryStatistics().normL2.toArray这个方法:
def computeColumnSummaryStatistics(): MultivariateStatisticalSummary = {
val summary = rows.treeAggregate(new MultivariateOnlineSummarizer)(
(aggregator, data) => aggregator.add(data),
(aggregator1, aggregator2) => aggregator1.merge(aggregator2))
updateNumRows(summary.count)
summary
}
之前有介绍这个treeAggregate是一种带“预reduce”的map-reduce,返回的summary,里面帮我们统计了每一个向量的很多指标,比如
currMean 为 每一个向量的平均值
currM2 为 每个向量的每一维的平方和
currL1 为 每个向量的绝对值的和
currMax 为 每个向量的最大值
currMin 为 每个向量的最小值
nnz 为 每个向量的非0个数
这里我们只需要currM2,它是每个向量的平方和。summary调用的normL2方法:
override def normL2: Vector = {
require(totalWeightSum > 0, s"Nothing has been added to this summarizer.")
val realMagnitude = Array.ofDim[Double](n)
var i = 0
val len = currM2.length
while (i < len) {
realMagnitude(i) = math.sqrt(currM2(i))
i += 1
}
Vectors.dense(realMagnitude)
}
上面这步就是对平方和开个根号,这样就求出来了每个向量的分母部分。
下面就是最关键的地方了:
private[mllib] def columnSimilaritiesDIMSUM(
colMags: Array[Double],
gamma: Double): CoordinateMatrix = {
// 一些参数校验
// 对gamma进行开方
val sg = math.sqrt(gamma) // sqrt(gamma) used many times
// 这里把前面算的平方根的值设置一个默认值,因为如果为0,除0会报异常,所以设置为1
val colMagsCorrected = colMags.map(x => if (x == 0) 1.0 else x)
// 把抽样概率数组 和 平方根数组进行广播
val sc = rows.context
val pBV = sc.broadcast(colMagsCorrected.map(c => sg / c))
val qBV = sc.broadcast(colMagsCorrected.map(c => math.min(sg, c)))
// 遍历每一行,计算每个向量该维的乘积,形成三元组
val sims = rows.mapPartitionsWithIndex { (indx, iter) =>
val p = pBV.value
val q = qBV.value
// 获得随机值
val rand = new XORShiftRandom(indx)
val scaled = new Array[Double](p.size)
iter.flatMap { row =>
row match {
case SparseVector(size, indices, values) =>
// 如果是稀疏向量,遍历向量的每一维,除以平方根
val nnz = indices.size
var k = 0
while (k < nnz) {
scaled(k) = values(k) / q(indices(k))
k += 1
}
// 遍历向量数组,计算每一个数值与其他数值的乘机。
// 比如向量(1, 2, 0 ,1)
// 得到的结果为 (0,1,value)(0,3,value)(2,3,value)
Iterator.tabulate (nnz) { k =>
val buf = new ListBuffer[((Int, Int), Double)]()
val i = indices(k)
val iVal = scaled(k)
// 判断当前列是否符合采样范围,如果小于采样值,就忽略
if (iVal != 0 && rand.nextDouble() < p(i)) {
var l = k + 1
while (l < nnz) {
val j = indices(l)
val jVal = scaled(l)
if (jVal != 0 && rand.nextDouble() < p(j)) {
// 计算每一维与其他维的值
buf += (((i, j), iVal * jVal))
}
l += 1
}
}
buf
}.flatten
case DenseVector(values) =>
// 跟稀疏同理
val n = values.size
var i = 0
while (i < n) {
scaled(i) = values(i) / q(i)
i += 1
}
Iterator.tabulate (n) { i =>
val buf = new ListBuffer[((Int, Int), Double)]()
val iVal = scaled(i)
if (iVal != 0 && rand.nextDouble() < p(i)) {
var j = i + 1
while (j < n) {
val jVal = scaled(j)
if (jVal != 0 && rand.nextDouble() < p(j)) {
buf += (((i, j), iVal * jVal))
}
j += 1
}
}
buf
}.flatten
}
}
// 最后再执行一个reduceBykey,累加所有的值,就是i和j的相似度
}.reduceByKey(_ + _).map { case ((i, j), sim) =>
MatrixEntry(i.toLong, j.toLong, sim)
}
new CoordinateMatrix(sims, numCols(), numCols())
}
这样把所有向量的平方和广播后,每一行都可以在不同的节点并行处理了。
总结来说,Spark提供的这个计算相似度的方法有两点优势:
- 通过拆解公式,使得每一行独立计算,加快速度
- 提供采样方案,以采样方式抽样固定的特征维度计算相似度
不过杰卡德目前并不能使用这种方法来计算,因为杰卡德中间有一项需要对向量求dot,这种方式就不适合了;如果杰卡德想要快速计算,可以去参考LSH局部敏感哈希算法,这里就不详细说明了。