从线性回归到逻辑回归
最简单的回归是线性回归,在Andrew NG的讲义,有如图1.a所示,X为数据点——肿瘤的大小,Y为观测值——是否是恶性肿瘤。通过构建线性回归模型,如hθ(x)所示,构建线性回归模型后,即可以根据肿瘤大小,预测是否为恶性肿瘤hθ(x)≥.05为恶性,hθ(x)<0.5为良性。
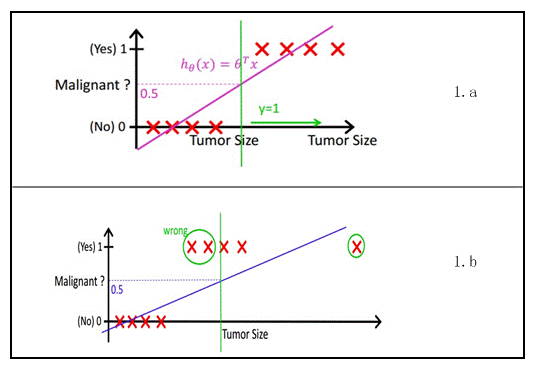
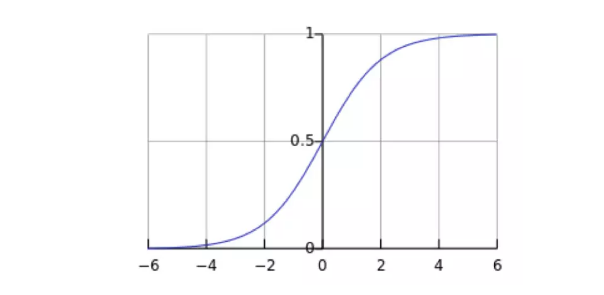
然而线性回归的鲁棒性很差,例如在图1.b的数据集上建立回归,因最右边噪点的存在,使回归模型在训练集上表现都很差。这主要是由于线性回归在整个实数域内敏感度一致,而分类范围,需要在[0,1]。逻辑回归就是一种减小预测范围,将预测值限定为[0,1]间的一种回归模型,其回归方程与回归曲线如图2所示。逻辑曲线在z=0时,十分敏感,在z>>0或z<<0处,都不敏感,将预测值限定为(0,1)。
分类问题
二分类问题就是给定的输入 ,判断它的标签是A类还是类。二分类问题是最简单的分类问题。
-
预测一个用户是否点击特定的商品
-
判断用户的性别
-
预测用户是否会购买给定的品类
-
判断一条评论是正面的还是负面的
要解决这些问题,通常会用到一些已有的分类算法,比如逻辑回归,或者支持向量机。它们都属于有监督的学习,因此在使用这些算法之前,必须要先收集一批标注好的数据作为训练集。有些标注可以从log中拿到(用户的点击,购买),有些可以从用户填写的信息中获得(性别),也有一些可能需要人工标注(评论情感极性)。另一方面,知道了一个用户或者一条评论的标签后,我们还需要知道用什么样的特征去描述我们的数据,对用户来说,可以从用户的浏览记录和购买记录中获取相应的统计特征,而对于评论来说,最直接的则是文本特征。
但是我们拿得到的数据往往是连续的变量,输出的结果也是连续的变量。所以就考虑到下面的问题。
用连续的数值去预测离散的标签值
线性回归的输出是一个数值,而不是一个标签,显然不能直接解决二分类问题。那我如何改进我们的回归模型来预测标签呢?
感知机(Perceptron)模型:
一个最直观的办法就是设定一个阈值,比如0,如果我们预测的数值 y > 0 ,那么属于标签A,反之属于标签B。
基于概率的辑回归模型 (Logistics Regression):
我们不去直接预测标签,而是去预测标签为A概率,我们知道概率是一个[0,1]区间的连续数值,那我们的输出的数值就是标签为A的概率。一般的如果标签为A的概率大于0.5,我们就认为它是A类,否则就是B类。
二分类模型评价
拿到数据的特征和标签后,就得到一组训练数据:
其中 xi 是一个 m 维的向量,Misplaced & ,y 在 {0, 1} 中取值。(本文用{1,0}表示正例和负例,后文沿用此定义。)
问题可以简化为,如何找到这样一个决策函数y∗=f(x),它在未知数据集上能有足够好的表现。
逻辑回归推导
从线性回归到逻辑回归
线性回归的表达式:,线性回归对于给定的输入
,输出的是一个数值 y ,因此它是一个解决回归问题的模型。
为了消除掉后面的常数项b,我们可以令 ,同时
,也就是说给x多加一项而且值恒为1,这样b就到了w里面去了,直线方程可以化简成为:
,后面使用的
代指
。
概率是属于[0,1]区间。但是线性模型 值域是
。所以不能直接基于线性模型建模。需要找到一个模型的值域刚好在[0,1]区间,同时要足够好用,sigmoid函数是不二之选。
它的表达式为: 。
结合sigmoid函数,线性回归函数,把线性回归模型的输出作为sigmoid函数的输入。于是最后就变成了逻辑回归模型:
假设我们已经训练好了一组权值 。只要把我们需要预测的
代入到上面的方程,输出的y值就是这个标签为A的概率,我们就能够判断输入数据是属于哪个类别。
逻辑回归的损失函数(Loss Function)
损失函数就是用来衡量模型的输出与真实输出的差别。
假设只有两个标签1和0, 。我们把采集到的任何一组样本看做一个事件的话,那么这个事件发生的概率假设为p。我们的模型y的值等于标签为1的概率也就是p。
。因为标签不是1就是0,因此标签为0的概率就是:
。
我们把单个样本看做一个事件,那么这个事件发生的概率就是:这个函数不方便计算,它等价于:
。
解释下这个函数的含义,我们采集到了一个样本 。对这个样本,它的标签是
的概率是
。 (当y=1,结果是p;当y=0,结果是1-p)。
如果我们采集到了一组数据一共N个, ,这个合成在一起的合事件发生的总概率怎么求呢?其实就是将每一个样本发生的概率相乘就可以了,即采集到这组样本的概率:
。注意
是一个函数,并且未知的量只有
(在p里面)。
通过两边取对数来把连乘变成连加的形式,即:
其中,
,这个函数
又叫做它的损失函数。
损失函数可以理解成衡量我们当前的模型的输出结果,跟实际的输出结果之间的差距的一种函数。这里的损失函数的值等于事件发生的总概率,我们希望它越大越好。但是跟损失的含义有点儿违背,因此也可以在前面取个负号。
使用最大似然估计MLE(Maximum Likelihood Estimation)估计参数 跟b的值呢,损失函数
是正比于总概率
的,而
又只有一个变量
。也就是说,通过改变
的值,就能得到不同的总概率值
。那么当我们选取的某个
刚好使得总概率
取得最大值的时候。我们就认为这个
就是我们要求得的
的值,这就是最大似然估计的思想。
现在问题变成了找到一个 ,使得我们的总事件发生的概率,即损失函数
取得最大值,数学表达就是:
求取 的梯度
?
p是一个关于变量 的函数,我们对p求导,通过链式求导法则,可得:
的值:
,我们也对1-p进行如上的求导。
最终求得: 和
。
正式开始对 求导,求导的时候请始终记住,我们的变量只有
,其他的什么
都是已知的,看做常数。
所以,梯度 的表达式最终为:
把p再展开,即:
梯度下降法(GD)与随机梯度下降法(SGD)
为什么可以用梯度下降法?
因为逻辑回归的损失函数L是一个连续的凸函数(conveniently convex)。这样的函数的特征是,它只会有一个全局最优的点,不存在局部最优。
在逻辑回归里面就不存在局部最优,因为它的损失函数的良好特性,导致它并不会有好几个局部最优。
当我们的GD跟SGD收敛以后,我们得到的极值点一定就是全局最优的点,因此我们可以放心地用GD跟SGD来求解。
已经解出了损失函数 在任意
处的梯度
,我们现在要求损失函数取最大值时候的
的值:
梯度下降法(Gradient Descent),可以用来解决这个问题。
核心思想就是先随便初始化一个 ,然后给定一个步长
,通过不断地修改
<-
,从而最后靠近到达取得最大值的点,即不断进行下面的迭代过程,直到达到指定次数,或者梯度等于0为止。
随机梯度下降法(Stochastic Gradient Descent),如果我们能够在每次更新过程中,加入一点点噪声扰动,可能会更加快速地逼近最优值。
在SGD中,我们不直接使用 ,而是采用另一个输出为随机变量的替代函数
:
当然,这个替代函数 需要满足它的期望值等于
,相当于这个函数围绕着
的输出值随机波动。
在GD求导每次都用到了所有的样本点,从1一直到N都参与梯度计算。
在SGD中,我们每次只要均匀地、随机选取其中一个样本 ,用它代表整体样本,即把它的值乘以N,就相当于获得了梯度的无偏估计值,即
,因此SGD的更新公式为:
这样我们前面的求和就没有了,同时 都是常数,
的值刚好可以并入
当中,因此SGD的迭代更新公式为:
,其中
是对所有样本随机抽样的一个结果。
加入正则项的损失函数
正则化是用来防止模型过拟合的过程,常用的有L1正则化和L2正则化两种选项,分别通过在损失函数后加上参数向
量 的L1范式和L2范式的倍数来实现。这个增加的范式,被称为“正则项”,也被称为"惩罚项"。损失函数改变,基
于损失函数的最优化来求解的参数取值必然改变,我们以此来调节模型拟合的程度。其中L1范式表现为参数向量中
的每个参数的绝对值之和,L2范数表现为参数向量中的每个参数的平方和的开方值。
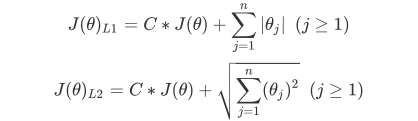
加入正则的损失函数,C是用来控制正则化程度的超参数,n是方程中特征的总数,也是方程中参
数的总数,j代表每个参数。j要大于等于1,是因为我们的参数向量 中,第一个参数是 ,是我们的截
距,它通常是不参与正则化的。
| penalty |
可以输入"l1"或"l2"来指定使用哪一种正则化方式,不填写默认"l2"。 |
| C |
C正则化强度的倒数,必须是一个大于0的浮点数,不填写默认1.0,即默认正则项与损失函数的 |
sklearn实战
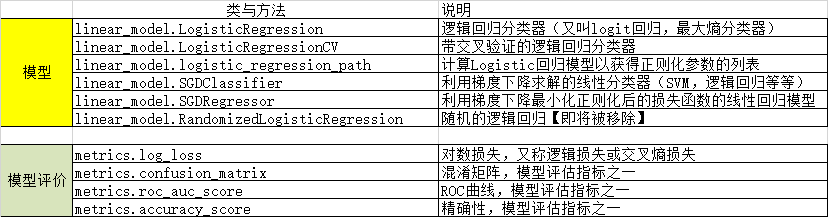
LogisticRegression详解
class sklearn.linear_model.LogisticRegression(penalty='l2', dual=False, tol=0.0001, C=1.0, fit_intercept=True, intercept_scaling=1, class_weight=None,
random_state=None, solver='lbfgs', max_iter=100, multi_class='auto', verbose=0, warm_start=False, n_jobs=None, l1_ratio=None)
参数介绍

求解器
梯度下降法,只是求解逻辑回归参数 的一种方法,sklearn提供了更多选择,让我们可以使用不同的求解器来计算逻辑回归。
求解器的选择,由参数"solver"控制,共有五种选择。其中“liblinear”是二分类专用,也是现在的默认求解器。

数据证明:
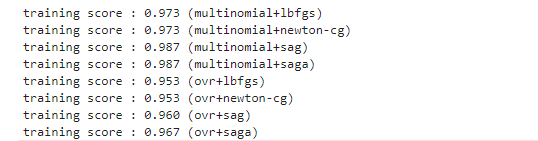
莺尾花数据集多分类问题

from sklearn.datasets import load_iris from sklearn.linear_model import LogisticRegression as LR iris = load_iris() for multi_class in ('multinomial', 'ovr'): for solver in ('lbfgs','newton-cg','sag','saga'): clf = LR(solver=solver, max_iter=100, random_state=42,multi_class=multi_class).fit(iris.data, iris.target) print("training score : %.3f (%s+%s)" % (clf.score(iris.data, iris.target),multi_class,solver))
乳腺癌数据实战
乳腺癌数据集
import numpy as np import matplotlib.pyplot as plt from sklearn.linear_model import LogisticRegression as LR from sklearn.datasets import load_breast_cancer from sklearn.model_selection import train_test_split from sklearn.metrics import accuracy_score#精确性分数 data = load_breast_cancer()#乳腺癌数据集 X = data.data y = data.target dir(data) data.feature_names

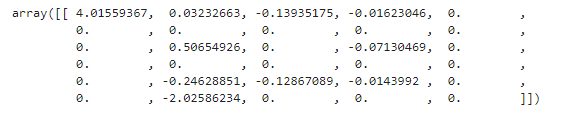
逻辑回归的重要属性coef_
X.data.shape#(569, 30) lrl1 = LR(penalty="l1",solver="liblinear",C=0.5,max_iter=1000) #逻辑回归的重要属性coef_,查看每个特征所对应的参数 lrl1 = lrl1.fit(X,y) lrl1.coef_ # (lrl1.coef_ != 0).sum(axis=1)#array([10]) 30个特征中有10个特征的系数不为0
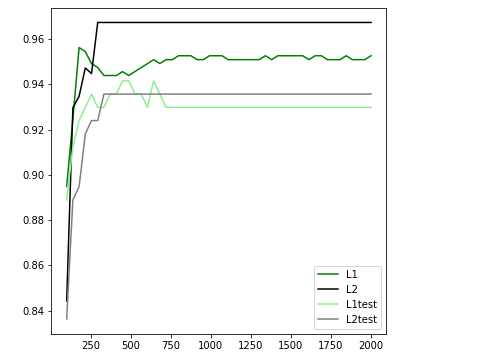
对参数C对的调优
l1 = [] l2 = [] l1test = [] l2test = [] Xtrain, Xtest, Ytrain, Ytest = train_test_split(X,y,test_size=0.3,random_state=420) for i in np.linspace(0.05,1.5,19): lrl1 = LR(penalty="l1",solver="liblinear",C=i,max_iter=1000) lrl2 = LR(penalty="l2",solver="liblinear",C=i,max_iter=1000) lrl1 = lrl1.fit(Xtrain,Ytrain) l1.append(accuracy_score(lrl1.predict(Xtrain),Ytrain)) l1test.append(accuracy_score(lrl1.predict(Xtest),Ytest)) lrl2 = lrl2.fit(Xtrain,Ytrain) l2.append(accuracy_score(lrl2.predict(Xtrain),Ytrain)) l2test.append(accuracy_score(lrl2.predict(Xtest),Ytest)) graph = [l1,l2,l1test,l2test] color = ["green","black","lightgreen","gray"] label = ["L1","L2","L1test","L2test"] plt.figure(figsize=(6,6)) for i in range(len(graph)): plt.plot(np.linspace(0.05,1.5,19),graph[i],color[i],label=label[i]) plt.legend(loc=4) #图例的位置在哪里?4表示,右下角 plt.show()

对参数max_iter的调
from sklearn.model_selection import cross_val_score l1 = [] l2 = [] l1test = [] l2test = [] Xtrain, Xtest, Ytrain, Ytest = train_test_split(X,y,test_size=0.3,random_state=420) for i in np.linspace(1,200,50): lrl1 = LR(penalty="l1",solver="liblinear",C=0.75,max_iter= i ) lrl2 = LR(penalty="l2",solver="liblinear",C=0.75,max_iter= i ) lrl1 = lrl1.fit(Xtrain,Ytrain) l1.append(cross_val_score(lrl1,data.data,data.target,cv=10).mean()) l1test.append(accuracy_score(lrl1.predict(Xtest),Ytest)) lrl2 = lrl2.fit(Xtrain,Ytrain) l2.append(accuracy_score(lrl2.predict(Xtrain),Ytrain)) l2test.append(accuracy_score(lrl2.predict(Xtest),Ytest)) graph = [l1,l2,l1test,l2test] color = ["green","black","lightgreen","gray"] label = ["L1","L2","L1test","L2test"] plt.figure(figsize=(6,6)) for i in range(len(graph)): plt.plot(np.linspace(100,2000,50),graph[i],color[i],label=label[i]) plt.legend(loc=4) #图例的位置在哪里?4表示,右下角 plt.show()
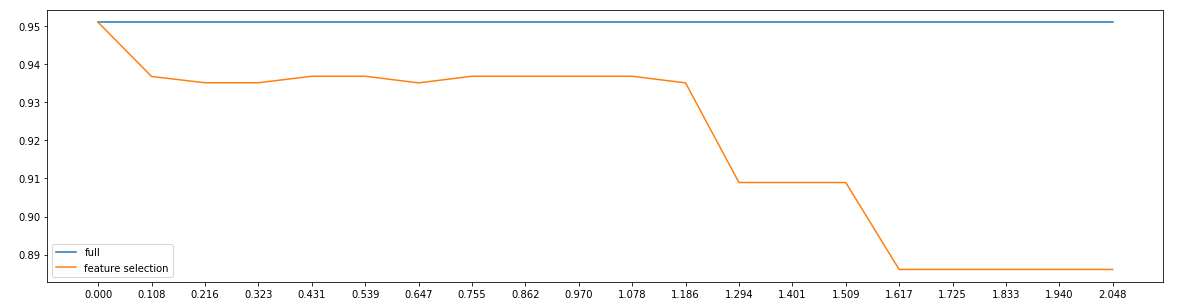
特征筛选
fullx = [] fsx = [] threshold = np.linspace(0,abs((LR_.fit(data.data,data.target).coef_)).max(),20) #全特征 cross_val_score_value=cross_val_score(LR_,data.data,data.target,cv=5).mean() #特征筛选 for i in threshold: X_embedded = SelectFromModel(LR_,threshold=i).fit_transform(data.data,data.target) fullx.append(cross_val_score_value) fsx.append(cross_val_score(LR_,X_embedded,data.target,cv=5).mean()) plt.figure(figsize=(20,5)) plt.plot(threshold,fullx,label="full") plt.plot(threshold,fsx,label="feature selection") plt.xticks(threshold) plt.legend() plt.show()