python机器学习生物信息学,博主录制,2k超清
http://dwz.date/b9vw
上一篇文章写了数据分析系列:归因分析原理、案例和python代码。但是现实中用户所归属的渠道可能很多,比如用户在网上商城的首页点击了一个产品,又在其他公众号的相关推荐点击了此产品,在所有转化用户中,每个渠道的贡献率是多大?这就是归因分析中的多渠道归因。
对于多渠道归因,有一些启发式的归因方法,比如“最终点击”(将订单归属于最后一个渠道)、“非最终点击”(归属于倒数第二个渠道)、“首次点击”(第一次点击)、“线性衰减”(时间越远权重越小)、“线性平均”(各经历渠道权重相同)、“根据位置”(自己定义每个位置权重),可参考Google Analysis
上面的启发式归因,需要人为定义渠道归属或每个位置的权重,在大多数情况,当合理根据业务定义归属时,得出的结果是合理的,也是使用最多的方法。但是却避免不了存在主观性的缺点。**还有一个是根据算法归因的方式,包括使用逻辑回归、生存分析中的cox回归,马尔科夫链等。
其中逻辑回归和生存分析的多渠道归因大致方法是,求出回归中对应每个渠道前的系数βiβ_iβi,再根据βiβ_iβi求出对应的odd ratios:exp(βi)exp(β_i)exp(βi)(cox回归可以直接求出RR),再根据前一篇数据分析系列:归因分析原理、案例和python代码相关的公式求出每个渠道的PAF。
马尔科夫链的大致计算方式是:计算每个渠道之间以及最初/终状态(start、转化、未转化)之间的状态转移概率矩阵,再每次去掉其中一个渠道,计算其移除效应,再对每个渠道的移除效应进行标准化,最终的得到就是这个渠道的贡献大小。
逻辑回归不用多说了,到处都在用。网上的生存分析大多是使用R或者SPSS。本文主要使用python进行生存分析。
生存分析的理论很多,本文不涉及,推荐写的很好的生存分析系列文章生存分析简介。写的非常清晰,本人也是在长时间不使用后,看着这个系列的文章才慢慢回想起来。
在python中,生存分析的包是lifelines,可以利用lifelines进行累计生存曲线的绘制、Log Rank test、Cox回归等。
一、绘制S(t)
先使用自带的数据集
from lifelines.datasets import load_waltons from lifelines import KaplanMeierFitter from lifelines.utils import median_survival_times df = load_waltons() print(df.head(),' ') print(df['T'].min(), df['T'].max(),' ') print(df['E'].value_counts(),' ') print(df['group'].value_counts(),' ')
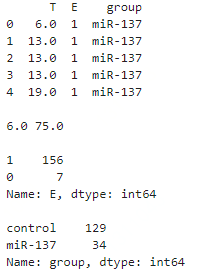
可以看到数据有三列,其中T代表min(T, C),其中T为死亡时间,C为观测截止时间。E代表是否观察到“死亡”,1代表观测到了,0代表未观测到,即生存分析中的删失数据,共7个。 group代表是否存在病毒, miR-137代表存在病毒,control代表为不存在即对照组,根据统计,存在miR-137病毒人数34人,不存在129人。
利用此数据取拟合拟生存分析中的Kaplan Meier模型(专用于估计生存函数的模型),并绘制全体人群的生存曲线。
kmf = KaplanMeierFitter() kmf.fit(df['T'], event_observed=df['E']) kmf.plot_survival_function() median_ = kmf.median_survival_time_ median_confidence_interval_ = median_survival_times(kmf.confidence_interval_) print(median_confidence_interval_)

图中蓝色实线为生存曲线,浅蓝色带代表了95%置信区间。随着时间增加,存活概率S(t)越来越小,这是一定的,同时S(t)=0.5时,t的95%置信区间为[53, 58]。这并不是我们关注的重点,我们真正要关注的实验组(存在病毒)和对照组(未存在病毒)的生存曲线差异。因此我们要按照group等于“miR-137”和“control”分组,分别观察对应的生存曲线:
groups = df['group']
ix = (groups == 'miR-137')
kmf.fit(df['T'][ix], df['E'][ix], label='miR-137')
ax = kmf.plot()
treatment_median_confidence_interval_ = median_survival_times(kmf.confidence_interval_)
print("带有miR-137病毒存活50%对应的存活时间95%置信区间:'
'", treatment_median_confidence_interval_, '
')
kmf.fit(df['T'][~ix], df['E'][~ix], label='control')
#共享一个画布
ax = kmf.plot(ax=ax)
control_median_confidence_interval_ = median_survival_times(kmf.confidence_interval_)
print("未带有miR-137病毒存活50%对应的存活时间95%置信区间:'
'", control_median_confidence_interval_)

可以看到,带有miR-137病毒的生存曲线在control组下方。说明其平均存活时间明显小于control组。同时带有miR-137病毒存活50%对应的存活时间95%置信区间为[19,29],对应的control组为[56,60]。差异较大,这个方法可以应用在分析用户流失等场景,比如我们对一组人群实行了一些防止流行活动,我们可以通过此种方式分析我们活动是否有效。
二、Cox回归
COX回归模型,又称“比例风险回归模型(proportional hazards model,简称Cox模型)”,是由英国统计学家D.R.Cox(1972)年提出的一种半参数回归模型。该模型以生存结局和生存时间为应变量,可同时分析众多因素对生存期的影响,能分析带有截尾生存时间的资料,且不要求估计资料的生存分布类型。由于上述优良性质,该模型自问世以来,在医学随访研究中得到广泛的应用,是迄今生存分析中应用最多的多因素分析方法。
对于回归模型的假设检验通常采用似然比检验、Wald检验和记分检验,其检验统计量均服从卡方分布。,其自由度为模型中待检验的自变量个数。一般说来,Cox回归系数的估计和模型的假设检验计算量较大,通常需利用计算机来完成相应的计算
通常存活时间与多种因素都存在关联,因此我们的面临的数据是多维的。下面使用一个更复杂的数据集。
from lifelines.datasets import load_regression_dataset from lifelines import CoxPHFitter regression_dataset = load_regression_dataset() print(regression_dataset.head()) print(regression_dataset['E'].value_counts())

其中T代表min(T, C),其中T为死亡时间,C为观测截止时间。E代表是否观察到“死亡”,1代表观测到了,0代表未观测到,即生存分析中的**“删失”**数据,删失数据共11个。var1,var2,var3代表了我们关系的变量,可以是是否为实验组的虚拟变量,可以是一个用户的渠道路径,也可以是用户自身的属性
我们利用此数据进行Cox回归
cph = CoxPHFitter() cph.fit(regression_dataset, 'T', event_col='E') cph.print_summary()

结果分析:从结果来看,我们认为var1和var3在5%的显著性水平下是显著的。认为var1水平越高,用户的风险函数值越大,即存活时间越短(cox回归是对风险函数建模,这与死亡加速模型刚好相反,死亡加速模型是对存活时间建模,两个模型的参数符号相反)。同理,var3水平越高,用户的风险函数值越大。
我们看到结果中第二列为exp(coef),我们还可以利用这个数字代替RR值,带入PAF的计算公式进行归因分析。请参考我的另一篇博客数据分析系列:归因分析原理、案例和python代码
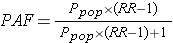
参考https://blog.csdn.net/u011517132/article/details/105528940
生存分析练习

从0到1Python数据科学之旅(博主录制)
http://dwz.date/cqpw