实验需要提取数据的空间信息,所以要对光谱进行降维,使用主成分分析算法,样例代码备份如下
# -*- coding: utf-8 -*-
"""
Created on Mon Feb 18 10:35:43 2019
@author: admin
"""
import numpy as np
from scipy.io import loadmat
#import spectral
from sklearn.decomposition import PCA
input_hsi = loadmat('dataset/huston/houston15.mat')['data']
input_lidar=loadmat('dataset/huston/houston_Lidar15.mat')['data']
train_label = loadmat('dataset/huston/houston15_mask_train.mat')['mask_train']
test_label=loadmat('dataset/huston/houston15_mask_test.mat')['mask_test']
#高光谱降维处理
array_x1=input_hsi.reshape(np.prod(input_hsi.shape[:2]),np.prod(input_hsi.shape[2:]))
pca=PCA(n_components=30)
array_x2=pca.fit_transform(array_x1)
print(array_x2.shape)
pca_hsi=array_x2.reshape(input_hsi.shape[0],input_hsi.shape[1],array_x2.shape[1])
print('hsi shape:',input_hsi.shape)
print('pca hsi shape:',pca_hsi.shape)
print('lidar shape:',input_lidar.shape)
实验数据是houston高光谱数据和lidar数据,数据的原始维度和降维后的维度如下图所示
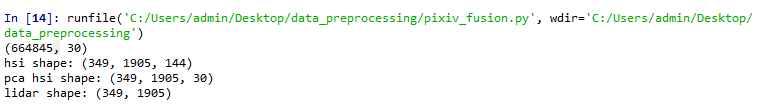
在进行pac降维之前需要先对实验reshape,因为pca只支持二维数据降维,将前两维(平面)reshape成列向量就变成了(664845,30),降维结束后再reshape回去,注意reshape时候第三个维度已经不是原来的144维了,所以在reshape回去时这样写是会报错的。input_hsi.shape[2]是原始数据的第三个维度为144,正确数值是30,即降维之后的维度,也就是array_x2.shape[1]
array_x3=array_x2.reshape(input_hsi.shape[0],input_hsi.shape[1],input_hsi.shape[2])
