四十而不惑——DNA测序技术的前世今生和未来
自上1953年DNA分子双螺旋结构被发表以来,生物学研究进入到了一个更精细化的分子时代,越来越多的科学家开始投入分子生物学研究,尤其是对DNA序列的研究,从而DNA测序技术应势而生。从1977年Sanger牛刀小试的双脱氧终止法到近十年如日中天的高通量测序技术,再到目前蓄势待发的三代单分子测序技术,这跨越近40年的风雨历程,
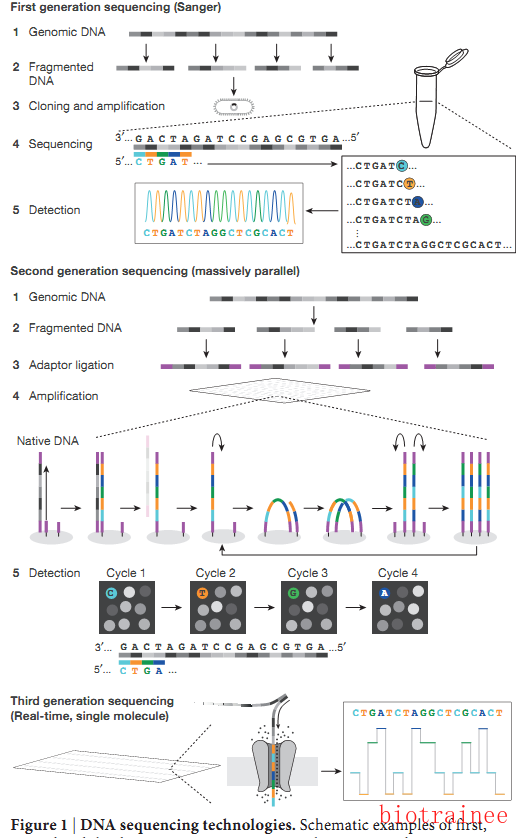
DNA测序是指测定和分析特定DNA片段中,碱基序列的排列情况。被广泛应用于生命科学和临床医学等领域的研究,如分子生物学,遗传学以及临床诊断,法医生物学等。而DNA测序技术自1977年sanger等人的双脱氧终止法到今天的单分子纳米孔测序法,已有40年的历史了。
这篇2017年10月发表在nature上的综述,回顾了DNA测序技术发明40年来的发展历程,并对DNA测序技术的现状及未来做了总结和展望。从最初几个碱基对到第一个人类基因组,这中间经历了多个技术革命和增长,如现在获得的百万人和无数其他多种的基因组等。我们预测,从长远来看,DNA测序创新将与显微镜的发明一样,影响深远。
DNA测序技术的历史
早期测序:
早在DNA测序技术被发明之前的1953年,就有胰岛素蛋白(insulin protein)被测序了;而后的1965年,tRNA(alanine tRNA:丙氨酸tRNA )也已经完成了测序,其中,第一个RNA测序用了140kg的酵母测出了76个核苷酸;在1968年,噬菌体λDNA的结合末端 (cohesive ends of phage lambda DNA)被测序完成。
在早期测序中,被测序的序列大多采用了类似的思路:将序列分离(色谱或电泳),分解成小片段(RNases或蛋白酶),然后破译每个小片段,并将片段重叠,最优由重叠部分推导出序列。
DNA测序技术的发明:
1973年,Gibert和Maxam使用引物延伸法测定了24个乳糖抑制因子结合位点的碱基(先将其复制成RNA,再测序),整个工作进行了两年时间,相当于每个核苷酸需要耗时一个月。
1976年,Sanger和Coulson开发的双脱氧链终止法;Gibert和Maxam发明的化学裂解法。这两种方法都是通过产生不同长度的片段(链终止合成或化学切割),然后都用聚丙烯酰胺凝胶电泳法测定了每个碱基反应中存在的片段的大小,并通过单碱基分辨率分离出DNA片段,将每个碱基一条标记的凝胶放置在X射线胶片上,产生一个梯形图像,从中即可读取该片段的序列,按照大小上升四条标记,推测碱基的顺序。
1979年,鸟枪法测序:是指将基因组按染色体分开后,将它全部打乱,切成碎片,然后进行随机测序,测序后再将其拼接起来。在HGP项目中,鸟枪法就被作为主要的测序技术起到作用,人类基因组的大片段被克隆成细菌人工染色体(BACs),每个BAC的DNA都是片段化的、被筛选的、被克隆的。单个克隆被选中并扩增生长,获得无性繁殖的基因文库,再筛选分离,从众多转化子菌株中选出含有目标序列的菌株,从中分离DNA,再将纯化的DNA作为sanger测序的模板,从凝胶扫描图像中提取信号,并调用碱基最终生成序列。
1987年,Smith、Hood和Applied Biosystems开发了桑格荧光测序仪,1000base/d
1982年,Genbank上存储了500 000bases
1986年,Genbank上存储了10 000 000bp
扩大到人类基因组:
1.技术进步:
1.从染料标记的引物到染料标记的末端;
2.突变的T7DNA聚合酶,更容易合并有染料标记的末端;
3.线性扩增反应,大大降低了模板要求;
4.基于磁珠的DNA纯化和提取,简化了测序前准备的自动化操作;
5.可以双链测序;可以使用利用质粒克隆,因此可以进行双末端测序;
6.毛细管电泳,消除了凝胶的浇注和负荷,也简化了荧光信号的提取和解释;
7.工业标准的加入,最大限度地提高了效率,减少了错误;
2.软件的开发:
phred,质量度量Q值,phrap,the TIGR assembler and the Celera assembler
3.测序成本的下降:
2001年,一个测序中心可测10 000 000bp/d
2004年 , ¥1/(600-700bP)
4.其他:
和HGP计划平行进行的私人基因组测序Craig Venter and Celera (2001)
大规模平行DNA测序:
1.核心是体外扩增产生每个模板拷贝被测序,而不是细菌克隆
2.几种扩增方式有polonies(polymerase colony ),桥式PCR或者滚环扩增(纳米球)
3.边合成边测序的3种方式:1)焦磷酸测序;2)使用DNA连接酶的特异性荧光寡核苷酸附在顺序的方式;3)聚合酶介导下逐步加入荧光标记的脱氧核苷酸
4.第一个NGS测序平台454(2005);Solexa(35bp 双端PE)
5.NGS测序平台:454(Roche公司),Solexa(illumina公司),Agencourt(Applied Biosystems),Helicos(founded by Quake),Complate Genomics(founded by Drmanac,华大收购 ),Ion Torrent(founded by Rothberg)
6.2012年illumina成为主流(454,Solid和Helicos停止开发);
准确度99.9%,Novaseq可以两天内产生1百亿reads,是HGP计划的40倍
单分子实时测序:
1.PacBio:
实时观察聚合酶介导的合成,是一种零模波导孔,即不到一半波长的光,将荧光激发到一个微小的体积内,其中一个聚合酶和它的模板存在,在不断增长的DNA链中加入了荧光标记的核苷酸,会发出足够持续时间的信号。
PacBio的通量比NGS平台(如illumina)要低很多,原始错误率也达到10%,但其读长长(大于10k)、GC偏好性低,并且PacBio的最小偏差、随机误差、长read和冗余覆盖率的组合可以致使从准确性和连续性方面进行重新组装的高质量。
2.纳米孔测序:
纳米孔测序(Oxford Nanopore Technologies,ONT)最早是在20世纪80年代提出的,它是基于一个单链DNA分子通过一个狭窄的通道时,离子流动的模式将揭示出链的主要序列,纳米孔测序 最长读长可达900k,便携式,电信号检测,但错误可能不是随机的,因为通过纳米级孔隙的电场驱动的DNA传输速度非常快,每个核苷酸的离子数量不足以产生足够的信号。
3.两者均可用于检测碱基修饰,如甲基化等
DNA测序的应用
DNA测序技术被广泛地应用于包括基因组从头组装、个体基因组重测序、临床测序和分子计数器等。
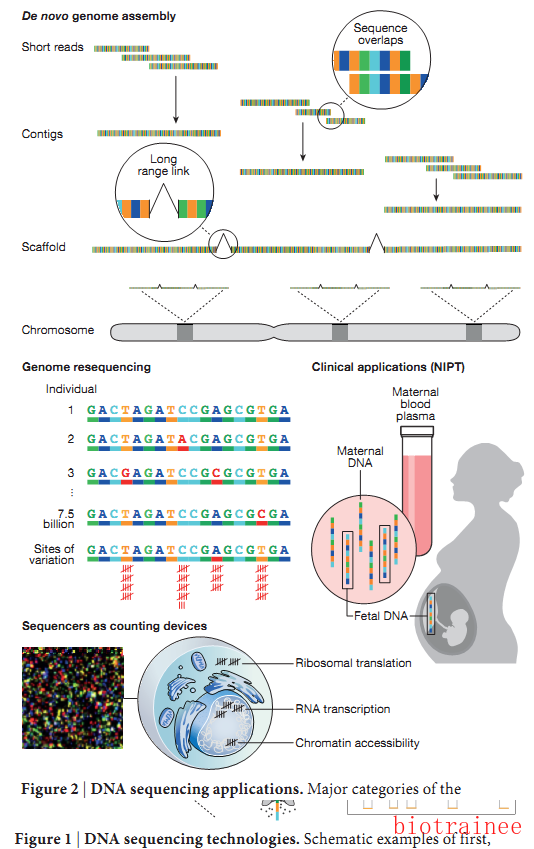
1.基因组的de novo(从头组装):
在早期的DNA测序工作中,对物种基因组进行部分或者完全测序是主要的内容,如1977年sanger测序的第一个基因组(phiX174;5kb),并且早期的这种DNA从头测序基本上是手工组装的。
随着NGS的到来,从头组装的数量急速增大,这时候基于de Bruijn图形的新组装算法(如EULER和Velvet)被开发出来了,这类算法克服了read和重复基因组的混乱的组合方式,但用于较大基因组时,尤其与HGP的基因组相比,其质量平均较差。较短的read可能是其中一个原因,但主要是因为缺乏补充NGS的邻接方法,NGS的文库方法在跨越距离方面很受限制,同时,也缺乏遗传和物理映射的“大规模平行”等价物。
另外,如HiC(全基因组的染色体构象捕获)和光学图谱,可以提供可伸缩的、可行性高的方法,在基因组框架下进行染色体规模的组装。
(1)遗传图谱;(2)物理图谱;(3)双端测序,8-10冗余,十万分之一的错误率;(4)Celera,从贪婪算法(phrap and the TIGR assembler,局部最优)到基于图的方法(重叠-布局-共识);(5)de Bruijn graphs(如 Euler and Velet);(6)高分子量high molecular weight(HMW);(7)HiC
2.基因组重测序:
基因组重测序正在被用于处理和探测遗传变异的筛查,如“1000美元人类基因组”等。与基因组组装不同,基因组重测序是指映射read到一个参考基因组来识别遗传变异,这其中就包括使用新的算法如bowtie and burrows-wheeler aligner(BWA)等,这类算法借鉴了数据压缩的思路,使得数百万的read能够有效地映射到参考基因组上,其中samtools和GATK是一个重要的应用。
由于测序成本的迅速降低,全基因组测序(WGS)的成本接近1000美元,而针对编码蛋白基因的选择性测序的全外显子测序(WES)的成本更是接近数百美元,从而会大大促进个人基因组重测序,如1000 genomes project,以及最近发布的基因组聚合数据库(http://gnomad.broadinstitute.org/)、Genomics England (https://www.genomicsengland.co.uk/)、NHLBI TOPMed (TransOmics for Precision Medicine, [https://www.nhlbiwgs.org/])等
测序的临床应用
1.无创产前诊断是一种非浸入性产前检测(non-invasion prenatal testing,NIPT),母体在怀孕期间,由胎儿释放到母体循环中的DNA片段的简单计数可以检测染色体非整倍体,基于该策略的筛查检测方法比之前任何一种分子检测速度都更快。
2.全外显子测序(WES),是快速发现新的基因,并用于诊断受孟德尔遗传疾病影响的患者,另外,由于很大比例的神经发育障碍都是由编码序列的从头突变所引起的,所以WES也被越来越多地用于诊断神经发育障碍的一个工具。
3.癌症诊断,癌症在根本上来说,也是一种基因突变引起的疾病,大规模的基因组重测序有助于揭示癌症的多重复杂的遗传异质性,因此,DNA测序在癌症的临床诊断中也起到了重要的作用,如:1)通过对肿瘤释放的循环细胞或无细胞DNA(ctDNA,cfDNA)的测序进行非侵入性诊断和监测;2)鉴定新的突变位点等
测序仪作为分子计数装置
在最早的时候,”表达序列标签(微阵列芯片分析)“是作为一种发现基因的方法被广泛应用,它通过基因表达的连续分析(SAGE),将基因表达变得可以“数字量化”。而随着NGS而来的RNA-Seq相对微阵列来说更具优势,比如2008年开发的RNA-Seq是通过新的软件包(tophat、cufflinks等)来对转录本进行计数的,这比原来基于杂交和模拟信号的统计方法来说,更加便捷高效。
在接下里的近十年时间里,又有数百种方案被开发出来,这极大地促进了DNA测序作为“分子计数器”而被广泛应用。DNA测序之于分子生物学,就如同显微镜之于细胞生物学一样,是基本的、必不可少的工具。
展望DNA测序
1.物种基因组的多样性
地球上有数百万的物种,至今已完成测序的物质只占有相当小的比例,所以,更全面、更广阔的基因组多样性正在等待被测序,被证明。
2.大规模人口的基因组重测序
随着DNA测序成本的降低,会有越来越多的人类选择将其基因组进行重测序,这可以发现突变位点(疾病);也可通过比较我们的基因组和祖先或者其他人类的基因组,可以更好地理解人类发展(进化)的历史。
3.DNA测序可作为新的显微镜
DNA测序发明至今只有40年,而显微镜已有近400年的时间,人们预测,DNA测序的寿命和影响,将与显微镜相提并论,甚至超过显微镜。
4.实时、便携的传感器
第三代纳米孔测序仪只用约70g重,并且其在30分钟内就可以获得样本的测序数据,所以,便携和快速可以使其成为一个传感器,实时地检测日常不同环境下的核酸数据。
参考:
https://mp.weixin.qq.com/s?src=11×tamp=1539134206&ver=1173&signature=Aj6zZoOjmo3DH7rxCrdCBEj2o4Z6zVQdnY3i8A0CAlJtg2qmMZS24uiI7GzIiJemhgHl6U2WkeVxZXzTWFmlzQEpA0hbeVjuQX0UBaQjbPoa6JKvuvMFpK-7m7NGkJRs&new=1