PCA可以将数据从原来的向量空间映射到新的空间中。由于每次选择的都是方差最大的方向,所以往往经过前几个维度的划分后,之后的数据排列都非常紧密了, 我们可以舍弃这些维度从而实现降维
原理
内积
两个向量的乘积满足:(ab= |a|cdot |b|cdot cos( heta)).如果(|b|=1)的话,(ab=|a| cdot cos( heta)). 而这个式子的含义就是a在b方向上的投影长度。pca用投影的长度的方差来衡量一个向量基的好坏。
基变换的矩阵表示
如果我想要把M个N维向量变换到M的R维向量。那么我需要:
- 把每条数据当成行向量X。即m行n列,共m条数据, (A=(x_1, x_2,...,x_m)^T)
- 每个基当成一个列向量,按列排成矩阵P, (P=(p_1, p_2, .., p_k))
- XP就是变换的结果
比如有两个基 ((1/sqrt2, 1/sqrt2)^T), ((-1/sqrt2, 1/sqrt2)^T).这是两个正交的基。一个向量((3, 2)^T).
- R个P表示R个基,即新空间有R维,因此 R<=m
- Pi* aj 表示 ai投影到pi上的投影的大小
这里也可以将新空间中的坐标转换回来,先看看他们的关系
所以要求X需要计算P的逆矩阵:
由于 (P cdot P^T =1), 故P是一个可逆矩阵,且模为1,(P^{-1} = P^T)。所以最后可以这样计算X:
最大化方差理论
每个向量投影到新的空间后,计算所有向量的投影的方差。方差越大,表示数据分布的越"散"。因为数据过于集中就不好处理,数据越散就越容易分开。实际上经过多次投影后,最后几个维度数据往往都集中在一起,这时这些维度就可以舍弃,这就是pca降维的思路。
pca降维的第一步是,让所有向量减去每个特征的平均值。这样会给后面的处理带来非常大的方便。比如计算一个向量的方差:
而减去均值后,每个向量的均值都为0,所以可以简化为:
注意!!!
这里的均值指的是同一个特征,不同的向量的均值。先通过每一个向量计算出各个特征的均值。再把每个向量的每个特征减去对应的均值。
PCA希望使用相关度最低的基来构建新的向量空间。也就是尽量寻找线性无关的向量(当然最好是正交),这样重合的信息会很少。在pca中,使用协方差来衡量各个特征之间的相关性。
- cov > 0 正相关
- cov == 0 不相关
- cov < 0 负相关
也就是说各个基向量之间满足cov==0就可以了。下面介绍协方差
协方差
协方差的计算公式如下(这里的(a_i, b_i)都是数字):
那么,要使协方差为0,也是ab=0,也就是向量正交!
一般情况下,a,b表示的是两个特征的列向量。并不是一条数据。而是各个数据的两个特征的一个列向量。
协方差矩阵
顾名思义,用矩阵来保存各个特征之前的协方差。如果数据有n个维度(n个特征),那么,他的协方差矩阵(sum)是一个n*n的矩阵。(sum_{ij}) 表示第i和第j个特征的相关度(协方差).
对于(m cdot n)的数据集X(m条数据集,每条数据n个特征),他的协方差可以通过下面的公式简单的计算出来:
也有人写成这种形式, (x^{(i)})表示第i个向量:
推理如下。(打公式太慢了)
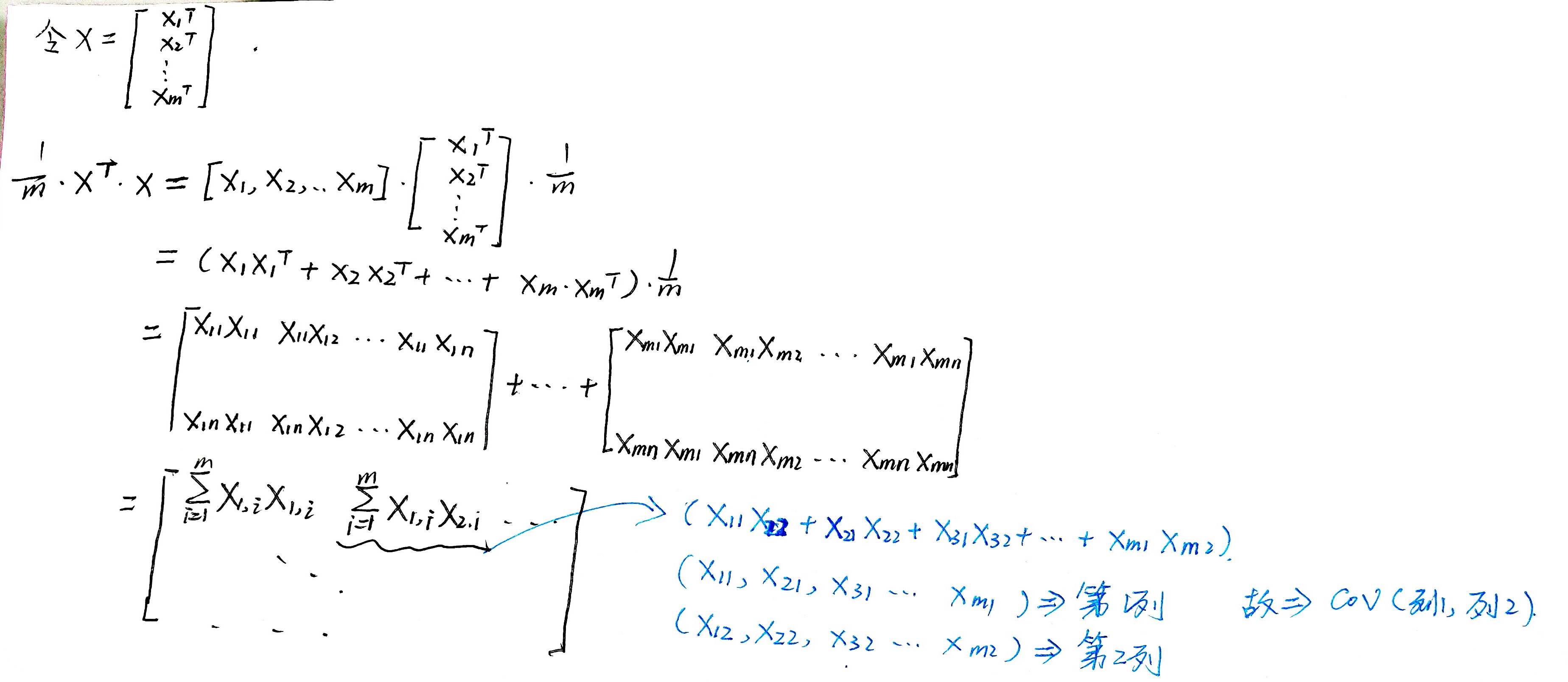
协方差矩阵和PCA
假设有m行n列的数据集 X(这里的X是1.2的X的转置),X 映射到新的向量空间后的坐标Y, Y的协方差矩阵为D。那么有以下关系:
这里的P是(n*k)的矩阵,由k的列向量组成,当k小于n时就是降维了。
所以,映射到新的坐标系后的协方差矩阵就是D这里有几点需要注意:
- 1、因为我们希望新的向量基是线性无关的,也就是不同的基之间的协方差应该为0。所以我们要让这个D变成对角矩阵(对角元其实就是方差)
- 2、n维实对称矩阵的性质:一定存在n的线性无关的特征向量。
- 3、通过(Lambda = P^TAP)的方式将实对称矩阵转换维对角矩阵。此时P是用n个线性无关的特征向量(列向量)组成的单位矩阵。(Lambda)是新的向量基对每个维度上的向量对应的方差。
- 4、选择最大的k个 (lambda) 对应的k个特征向量就是我们要求的向量基
实现
linalg.eig(covMat)是numpy的线性代数模块的函数。该函数的api的描述是,返回一个:
归一化(单位“长度”)特征向量,使得列v[:,i]是对应于特征值w[i]
pca的实现真的好简单啊,但是他的原理我花了好久才看明白。心累。
def pca(data, k):
# DeMean
dataMean = np.mean(data, axis=0)
data_demean = data - dataMean
# 计算协方差矩阵
covMat = data_demean.T.dot(data_demean) / len(data)
# 计算cov的特征值和特征向量
eigVals, eigVects = linalg.eig(covMat)
# 找到前k大的lambda对应的特征向量
eigVals_index = np.argsort(-eigVals) # 前k特征值的索引
eigVal_wanted = eigVals_index[:k] # 前k个特征值
eigVect_wanted = eigVects[:, eigVal_wanted] # 前k个特征向量
low_dim_data = data_demean.dot(eigVect_wanted) # 转换坐标
recon = low_dim_data.dot(eigVect_wanted.T) + dataMean # 还原坐标
return low_dim_data, recon
它可以将高维的数据转换到低维,也可以将转换过的坐标再还原。但是如果是从高维到低维,转换的过程已经损失了一部分数据了,再还原回去时,就和原来不一样了。这样有好也有坏,因为有时候,他还可以去除一些噪音数据。
随机使用一个数据:
X = np.empty((100,2))
X[:, 0] = np.random.uniform(0, 100, 100)
X[:, 1] = 0.75 * X[:, 0] + 3. + np.random.normal(0, 10, 100)
plt.scatter(X[:, 0], X[:, 1])
# 2维降1维
low, rec = pca(X, 1)
plt.scatter(rec[:, 0], rec[:,1])
如果,k的维度和原来一样,就可以无损的还原回来。但是如果降低了维度,再还原就会损失数据:

sklearn中的PCA
sklearn中的PCA在 sklearn.decomposition下的PCA中:
from sklearn.decomposition import PCA
pca = PCA(n_components=2)
pca.fit(X_train)
X_train_reduction = pca.transform(X_train)
X_test_reduction = pca.transform(X_test)
pca可以直接写浮点数,可以手动少选方差和大于阈值的维度
pca3 = PCA(0.9)
pca3.fit(X_train)
一些参数:
pca2.explained_variance_ # 在各个主成分上的方差
pca2.explained_variance_ratio_ # 各个主成分方差占总方差的比例