
传统的构建基因共表达网络的方法都是基于所有细胞的相似性,忽略了细胞之间的异质性,此方法提出了一种可以为每一个细胞构建此细胞对应的基因共表达网络的方法,并构建gene在不同细胞网络中的度矩阵,此度矩阵衡量了基因的重要程度,对度矩阵进行常规的聚类和分析操作为单细胞数据的特征分析提供了新的思路。【详见摘要】
方法:
方法用到了概率论中独立和用频数近似频率的概念,如果两个基因独立那么在当前的细胞状态下应该满足联合分布等于边缘分布的乘积形式,通过划定小的区域得到小的区域内点的个数近似概率。
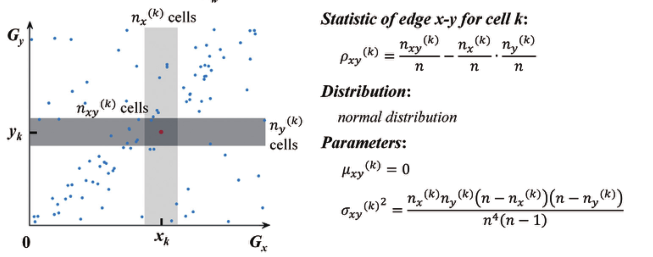
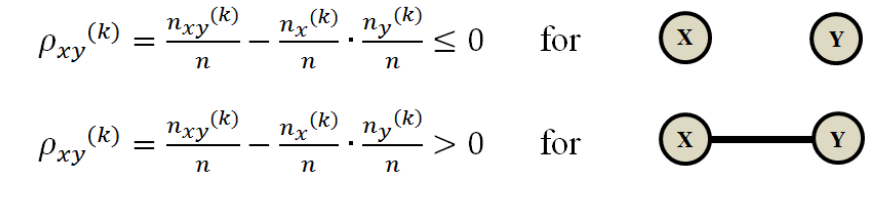
因为三个区域的大小会对结果影响比较大,所以推到了![]() 的分布,结果发现只有当nxy = nx*ny的时候,即两个基因独立的时候才满足分布是正态分布,并可以计算出正态分布的均值和方差,然后对
的分布,结果发现只有当nxy = nx*ny的时候,即两个基因独立的时候才满足分布是正态分布,并可以计算出正态分布的均值和方差,然后对![]() 进行正则化(减均值,去方差)然后定义原假设是两个gene独立,备择假设为两个gene不独立,则
进行正则化(减均值,去方差)然后定义原假设是两个gene独立,备择假设为两个gene不独立,则![]() 的拖尾严重不属于标准正态拖尾时拒绝原假设,即两个基因有一条边相连。
的拖尾严重不属于标准正态拖尾时拒绝原假设,即两个基因有一条边相连。
那么如何推到![]() 的分布就成为了问题的关键,首先我们直到当窗口nx和ny固定时,边缘分布是固定的,且出现在nxy区域的联合事件可以服从单次发生概率为nxy的二项分布,所以可以很容易的写出P(nxy = t | nx = i, ny = j)的概率形式具体推导:
的分布就成为了问题的关键,首先我们直到当窗口nx和ny固定时,边缘分布是固定的,且出现在nxy区域的联合事件可以服从单次发生概率为nxy的二项分布,所以可以很容易的写出P(nxy = t | nx = i, ny = j)的概率形式具体推导:

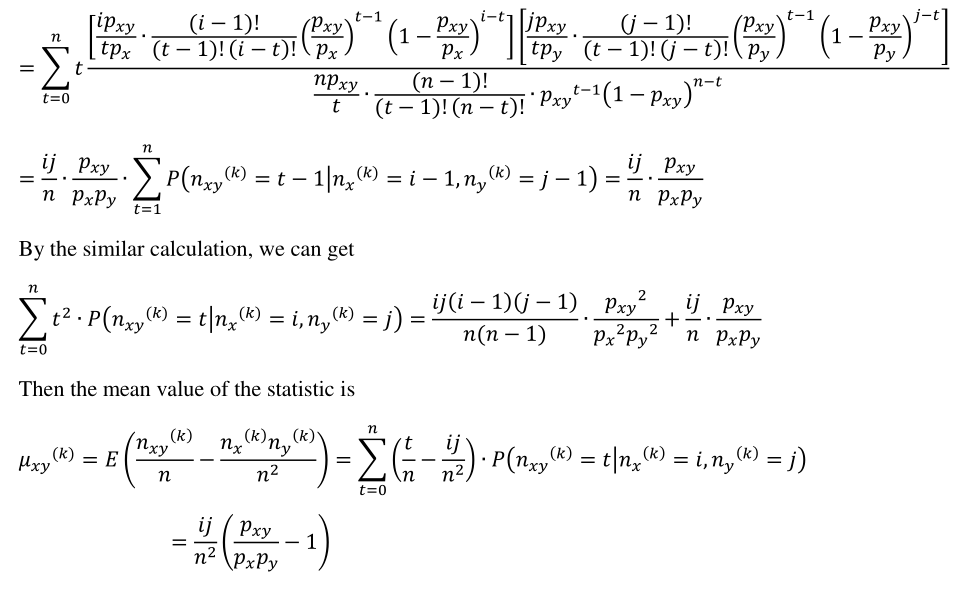

非常巧妙的就得到了每个细胞的基因网络,为了找到hard gene,设计了度矩阵,就是每个基因在不同的细胞网络中的度

公式推导的小tips:
