seaborn是基于matplotlib的数据可视化库.提供更高层的抽象接口.绘图效果也更好.

用seaborn探索数据分布
- 绘制单变量分布
- 绘制二变量分布
- 成对的数据关系可视化
绘制单变量分布
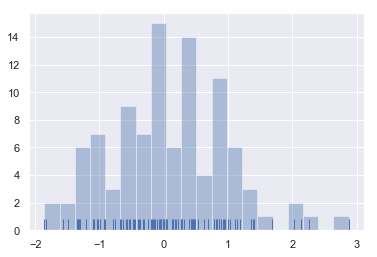
seaborn里最常用的观察单变量分布的函数是distplot()。默认地,这个函数会绘制一个直方图,并拟合一个核密度估计.如下所示:
x = np.random.normal(size=100)
sns.distplot(x);

首先解释一下啥叫核密度估计.wiki wiki里的一大堆数学证明看着就可怕....
简单地说:所谓核密度估计,就是采用平滑的峰值函数(“核”)来拟合观察到的数据点,从而对真实的概率分布曲线进行模拟.
说人话的话,就是:我们有很多样本,但再多的样本,也是离散的.所谓核密度估计,就是根据离散采样,估计连续密度分布. 所以样本量得大,假如你就3,5个样本,还有什么好估计的.
那核密度估计的原理是啥呢?
你上网搜索的话,大概率会搜到这个:
核密度函数的原理比较简单,在我们知道某一事物的概率分布的情况下,如果某一个数在观察中出现了,我们可以认为这个数的概率密度很大,和这个数比较近的数的概率密度也会比较大,而那些离这个数远的数的概率密度会比较小。
基于这种想法,针对观察中的第一个数,我们可以用K去拟合我们想象中的那个远小近大概率密度。对每一个观察数拟合出的多个概率密度分布函数,取平均。如果某些数是比较重要的,则可以取加权平均。
说实在的,我还是觉得很难理解.......
好在,一图胜千言!
x = np.random.normal(0, 1, size=30) bandwidth = 1.06 * x.std() * x.size ** (-1 / 5.) support = np.linspace(-4, 4, 200) kernels = [] for x_i in x: kernel = stats.norm(x_i, bandwidth).pdf(support) kernels.append(kernel) plt.plot(support, kernel, color="r") sns.rugplot(x, color=".2", linewidth=3);
.
即:假如有x个样本,如果我们用Gaussian Kernel Density(就是常说的正态分布啦),对每一个样本,我们都认为这个样本处在一个正态分布的峰值位置,(因为我们已经发现这个样本了).这样,就拟合出了x个正态分布曲线. 将这些曲线叠加取平均再正则化,就得到了最终的核密度估计分布曲线.
这里我们用的是kernel是gaussian.其实kernel有很多种,其中gau是最常用的.
kernel : {‘gau’ | ‘cos’ | ‘biw’ | ‘epa’ | ‘tri’ | ‘triw’ }, optional
displot参数
sns.distplot(x, kde=False, rug=True);
rug=True:绘制出离散样本

sns.distplot(x, bins=20, kde=False, rug=True);
bins=20:直方图的"柱子"的个数
更多参数,看文档去吧:displot api
绘制双变量分布
最常用的方法是散点图绘制 matplotlib.scatter seaborn中是jointplot
首先我们生成一个二元正态分布矩阵
mean, cov = [0, 1], [(1, .5), (.5, 1)]
data = np.random.multivariate_normal(mean, cov, 200)
df = pd.DataFrame(data, columns=["x", "y"])
散点图绘制sns.jointplot(x="x", y="y", data=df);
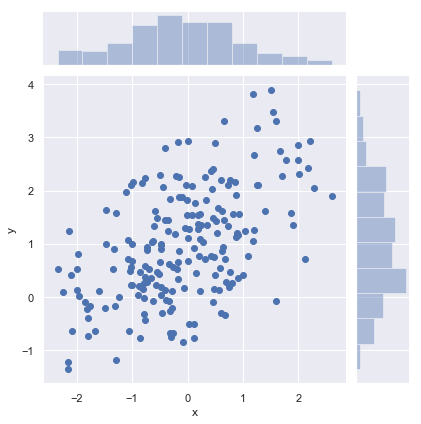
这种散点图有一个问题,相同的点会覆盖在一起.导致我们看不出来浓密和稀疏.
sns.jointplot(x=x, y=y, kind="hex", color="k");
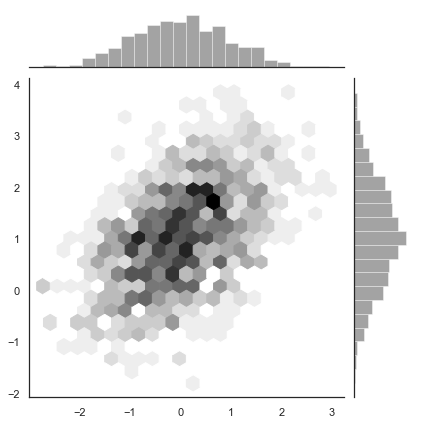
核密度分布估计

jointpoint函数使用JointGrid对象来绘图.为了提供更多的灵活性,jointpoint()返回一个JointGrid对象,你可以绘制出自己定制的图.
g = sns.jointplot(x="x", y="y", data=df, kind="kde", color="m")
g.plot_joint(plt.scatter, c="w", s=30, linewidth=1, marker="+")
g.ax_joint.collections[0].set_alpha(0)
g.set_axis_labels("$X$", "$Y$");

绘制成对的数据关系可视化
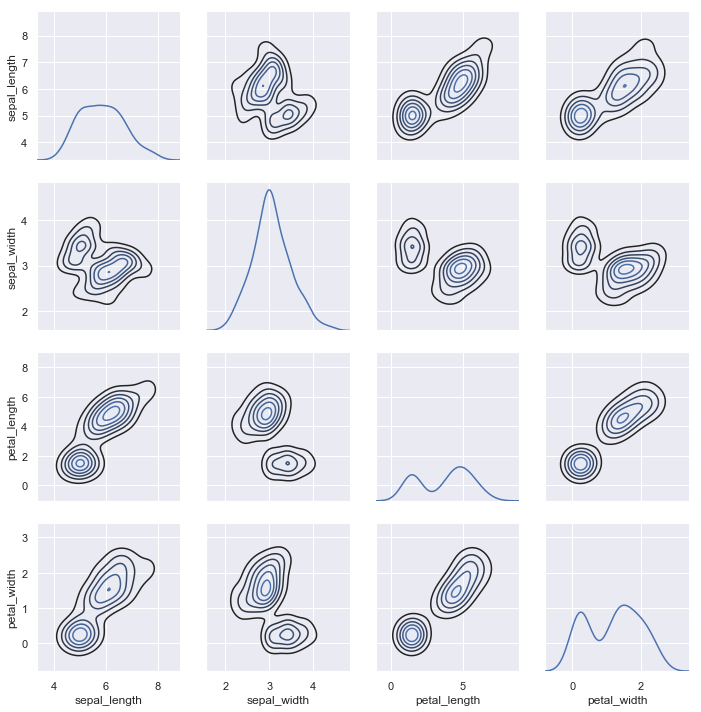
以著名的iris数据集为例. iris数据集有4个特征.那么两两组成一个pair的话,就有16种组合.用pairplot()绘制如下:
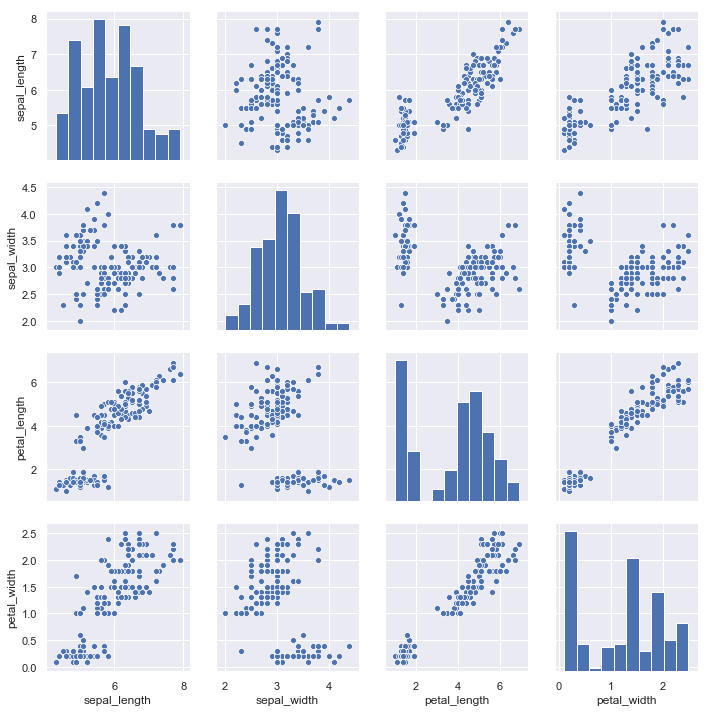
和jointplot类似,pairplot是基于PairPlot对象的.直接用PairPlot对象,可以更加灵活地绘制你想要的图
比如绘制iris数据集的核密度分布估计.
g = sns.PairGrid(iris)
g.map_diag(sns.kdeplot)
g.map_offdiag(sns.kdeplot, n_levels=6);