什么是snakemake?
snakemake 是一个流程搭建的工具,这里主要用来记录一些snakemake的使用方法
- 对于run或者shell部分的需要使用sample变量可以使用wildcards.sample来获取
- 对于写好的模块可以使用include来载入,然后使用rule all定义所有的输出,这样运行一个总的模块就可以直接开启整个流程
rule all
expand()
include:Filter.snakemake.py
- 指定一个config文件可以使用--configfile 参数来进行传入
- 如果只控制运行某一个rule,可以加上参数--forcerun rulex
- 做出流程图
snakemake --dag -s RNAseq.snakemake.py --configfile test.yaml |dot -Tpdf > dag.pdf
snakemake --dag -s RNAseq.snakemake.py --configfile test.yaml |dot -Tsvg > dag.svg
- rule 流程图
snakemake --rulegraph -s RNAseq.snakemake.py --configfile test.yaml |dot -Tpdf > rule.pdf
- 强制去重新运行一遍rule
snakemake -s RNAseq.snakemake.py --configfile test.yaml --forcerun filter_stat
- 指定运行的cpu数,使用-j参数
- 只打印不运行使用-n参数
- 在使用一个总的模块来将所有的模块串起来的过程中,rule all声明只能写在这个总的模块里面
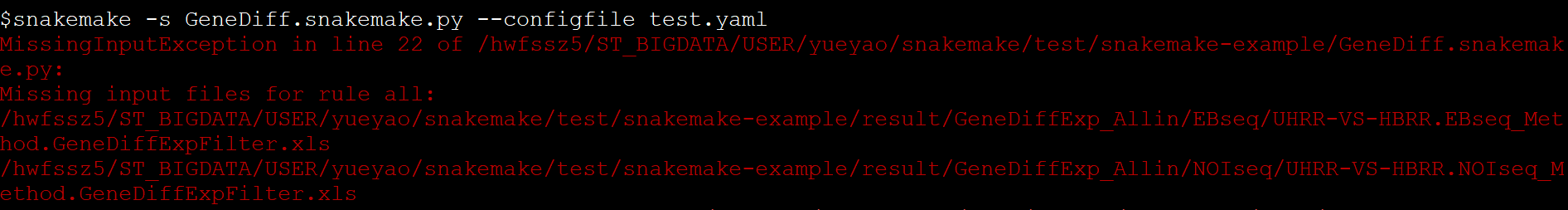
- 如下报错的原因是因为rule all里面的输出在其他的子rule里面没有做为输出
使用snakemake投递任务时
snakemake --cluster 'qsub -l vf=15G,num_proc=12 -cwd -q st.q -P P18Z10200N0170 -binding linear:12' --configfile test.yaml --snakefile Denovo.stLFR.snakemake.py --jobs 6
-
--jobs 能设置并行投递的任务
-
投递的命令需要加引号
-
当投递到集群,杀掉snakemake进程时,任务也会杀掉,而wdl是不会出现这种情况的
-
shell里面的命令{}需要用{{}}的方式来写,与python有点类似