转载自:http://blog.sciencenet.cn/blog-1509670-1000479.html
随着芯片和高通量测序技术的广泛应用,在肿瘤研究领域积累了越来越多的基因组学数据,特别是像The Cancer Genome Atlas(TCGA)、International Cancer Genome Consortium(ICGC)等大型肿瘤基因组测序计划的启动和完成,积累了大量的基因组学数据,这些数据至今为止已经占据了NCBI等公共数据库的半壁江山,同时也产生了各种各样的数据库以满足不同的研究需求。
对数据库的灵活运用已经成为科研工作者所必须掌握的基本技能,否则出去交流的时候都不好意思开口说不知道了。对于这些数据库,小编随口都能讲出十几个,例如***(此处省略一百字),然而,这么多数据库对于有选择困难症的人来说简直就是噩梦,很多人都在问有木有一个数据库能一次能满足我多个愿望呀?为此,小编对十几个数据库进行综合评估之后,给大家隆重推荐肿瘤基因组学数据库的终结者:cBioPortal网站(http://www.cbioportal.org/)。
cBioPortal网站整合了126个肿瘤基因组研究的数据,包括TCGA和ICGC等大型的肿瘤研究项目,涵盖了两万八千例标本的数据,此外部分样品还包括了临床预后等表型的信息。cBioPortal无需注册就能直接使用,而且提供一些小工具方便用户生成文章级别的图表,非常贴近用户的需求,下面小编就以EGFR基因在非小细胞肺腺癌的研究为例,带领各位小伙伴一步步玩坏cBioPortal数据库吧。
(一)EGFR在非小细胞肺腺癌的变异情况
首先,在cancer study选择要研究的肿瘤类型,如果想看候选基因在所有肿瘤的变异情况,就把所有肿瘤类型选上即可。在这里,我们可以看到对于肺癌有好几个研究项目的数据,一般我们选择样品量最多信息量最大的研究项目,所以我们选择了TCGA非小细胞肺癌项目(包括了522例样品)。然后,在genomic profiles选择要研究的组学数据类型,是研究突变还是拷贝数变化,还是多个组学的数据都考虑,在这类我们为了全面了解EGFR的变异情况,就把全部组学数据都选上了。最后,在gene set填上你所关注的基因,一个或者多个基因都可以,或者在user-defined list选择对特定肿瘤具有明确意义的基因。

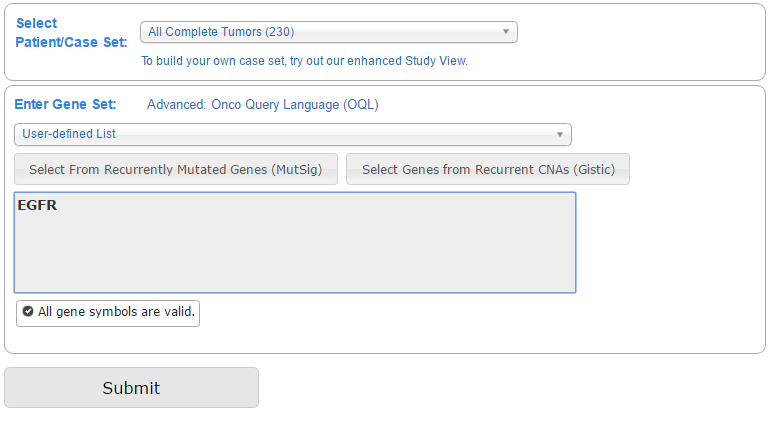
选择好上面的参数点击提交,之后只需静静等候即可得到详细的突变结果。在Oncoprint标签页里面,不同的图例代表不同的突变类型,此外,还能把变异比对到具体的蛋白结构域,并且配有蛋白三维结构的图(Mutations标签页)。关键是可以调整显示的方式和图形的大小,最终还能导出不同格式的图,满足各位发表文章的需要。

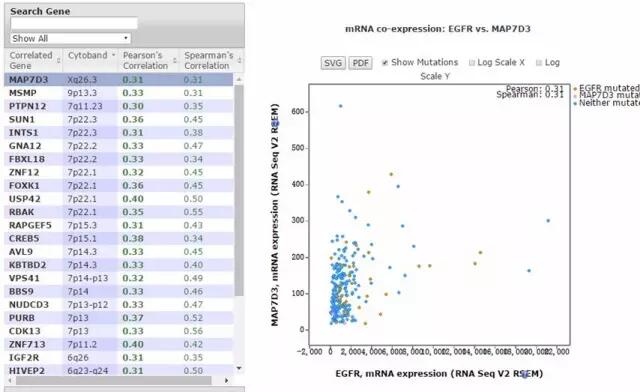
一般来说,要想研究特定基因作用的下游分子和信号通路,需要在体外做基因过表达或者敲除实验,然后通过qPCR或者高通量筛选的方法看哪些分子的表达量发生了变化,这些表达水平变化了的基因很可能就是对应的靶基因了。但是整个过程下来费时费力,而且实验还不一定顺利,并且我们有时候也仅仅想验证某类基因是否发生变化而已,这时候Co-Expression分析模块就非常适合了。它基于基因的表达量计算特定基因与其他每个基因的相关系数,相关系数越大证明两者之间关系越密切(正数代表正相关,负数代表负相关),越有可能是上下游作用关系,通过这个信息我们就能快速锁定下游的相关分子和通路了,指导我们下一步的验证工作。
临床上最关注的就是基因突变与预后之间的关系,一个好的标记物应该能很好的把病人进行分组,给临床治疗进行指导。为此cBioPortal也提供了临床预后的分析(仅适用于有临床预后信息的研究,例如TCGA项目),这样我们就可以不用SPSS等软件复杂的操作,也能做生存预后的分析,是不是很方便呢。

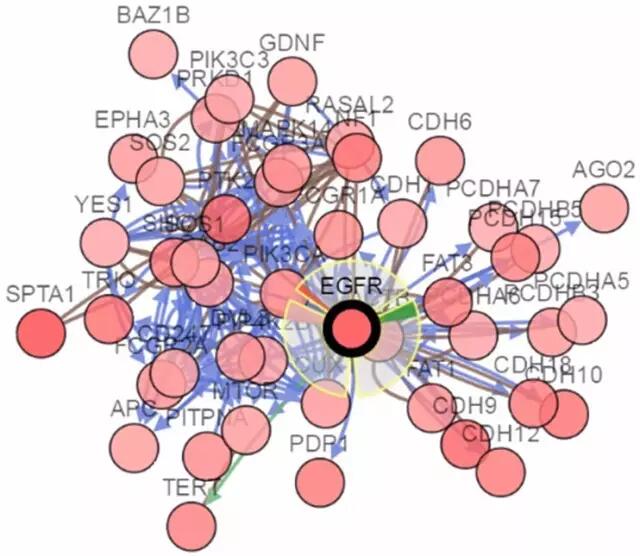
(四)EGFR调控网络和药物反应
对于调控网络cBioPortal通过基因互作数据库,例如PID、PANTHER、Reactome等数据库寻找与候选基因相关联的基因,然后通过互作强弱分析构建调控网络。此外,利用DrugBank、KEGG Drugs和Cancer Cell Line Encylopedia等药物反应数据库,针对相关的药物靶向的基因进行标注,从调控网络的角度指导分子互作和药物反应实验。
(转自:永诺生物)