Revealing disease-associated pathways by network integration of untargeted metabolomics--文献解读
Abstract
揭示失调的代谢物的分子环境是很重要的去理解疾病的通路.然而,由于全部代谢物鉴定的挑战,他们的系统分析一直受到限制.通过LC-MS法进行的非靶向代谢组学检测的大多数代谢物特征,如果没有额外的、耗时的实验,就不能唯一地确定。作者报告了一种基于网络的方法,用于非靶向代谢组学综合分析(PIUMet)的Steiner forest算法,该算法通过对代谢物特征的综合分析来推断分子路径和成分,而不需要对其进行识别。通过分析Huntington病模型中鞘脂、脂肪酸和类固醇代谢的变化,作者证明了PIUMet算法。此外,PIUMet使我们能够阐明病变细胞中代谢产物特征改变的假定身份,并通过实验推断出未检测到的与疾病相关的代谢物和失调蛋白。最后,作者建立了PIUMet将非靶向代谢组学数据与蛋白质组学数据进行综合分析的能力,证明该方法可产生无法通过单独分析这些数据推断的疾病相关代谢物和蛋白质。
Results
PIUMet overview
作者的PIUMet依靠 the prize-collecting Steiner forest algorithm算法.在正确的范围中,一个特征会匹配几种代谢物.PUIUMet使用可以区分出不同样本的原始峰作为输入,使用一个充分使用蛋白蛋白互作和蛋白代谢物互作的数据库(PPMI)的机器学习方法去推断一个失调代谢通路.并且在揭示疾病相关通路的背景下证明了我们的方法.作者认为改变的代谢物峰是'疾病特征'.我们将运行PIUMet之前身份未知的网络组件称为“隐藏”组件。与疾病特征直接相关的隐藏代谢物代表了它们的假定身份,而PIUMet鉴别出的其余隐藏代谢物和蛋白质是未通过实验直接测量的疾病相关蛋白质和代谢物。PIUMet可以进行多组分析,揭示代谢组失调和其他分子(如蛋白质)之间的联系.
PPMI数据库是作者通过整合生物化学反应的知识和从三个已建立的数据库中获得的蛋白质之间的相互作用来建立这个网络。算法的结果是一个加权图,其中节点代表代谢物或蛋白质,边缘显示蛋白质之间的相互作用以及酶和转运体反应.每条边都有一个权重来反映交互作用的可靠性.
为了解读疾病特征的背景,PIUMet不需要他们的事先识别;相反,它包含了特征的模糊身份.PIUMet将每个疾病特征表示为一个节点,与所有与疾病特征相似的物质连接。连接边具有任意且相等的权值.
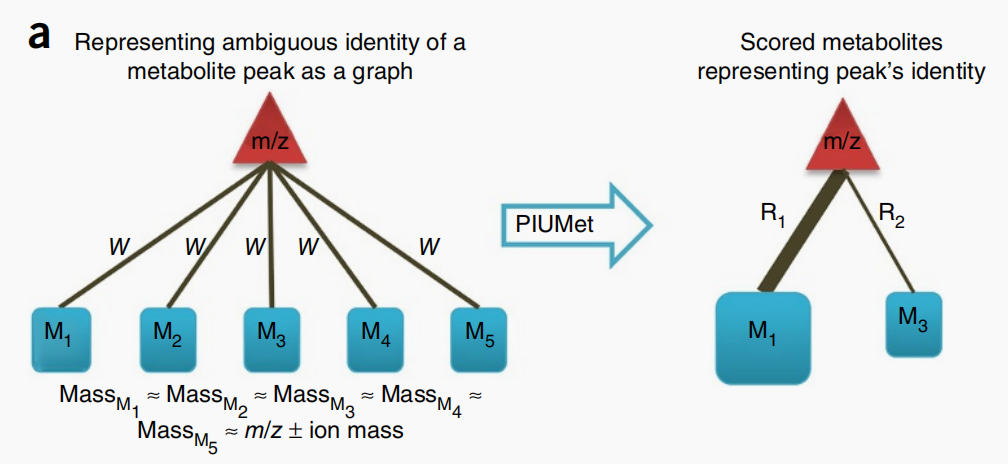
利用下面描述的网络优化技术,PIUMet确定了这些最可能对应疾病特征的代谢物的子集。它进一步计算出产生的代谢产物的分数(R),这表明它的识别对网络参数的鲁棒性增强.
PIUMet利用高概率的蛋白质-蛋白质和蛋白质-代谢物相互作用搜索PPMI相互作用体,寻找连接疾病特征的亚网络.
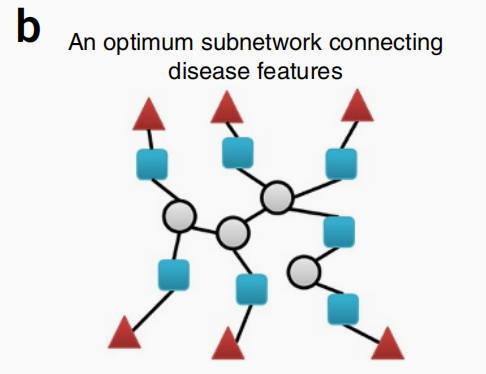
PIUMet使用 prize-collecting Steiner forest algorithm优化了网络,将奖励分配给疾病特征,并将成本分配给边缘权重.节点奖励反映了特征异常调节的重要性(由用户决定),而边缘成本与边缘置信度得分呈反相关.最佳的解决方案平衡了希望包含尽可能多的疾病特征和不愿使用低置信度边缘的愿望。具体地说,我们使关联疾病特征的奖励总和最大化,同时使最终网络包含的边缘成本最小化。