这一章主要讲的是逻辑回归,逻辑其实只是比线性回归多了一个逻辑函数。线性回归问题是f(x)=WTX+b用最优化方法求解W使得error=f(x)-y最小。线性回归是用f(x)取拟合的,但是逻辑回归的y值是{0,1},所以这里需要用一函数将所有输入映射到{0,1}。本来单位阶跃函数是最理想的,但是求最优时涉及求导什么的,单位阶跃在定义域内不可导。故一般用Sigmoid函数,即:
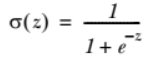 。Z=线性规划中的f(x)。这样一来问题还是用优化算法求最佳W。
。Z=线性规划中的f(x)。这样一来问题还是用优化算法求最佳W。
书中用的是梯度上升法与随机梯度上升法(训练样本大时用),梯度上升法与梯度下降基本差不多,一个求最大,一个求最小。书中不是损失函数,而是似然函数,故用梯度上升,下面复现书中的例子:
首先输入数据是.txt,是这样的:
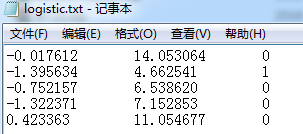
代码如下:
from numpy import * import matplotlib.pyplot as plt # 解析数据 def loadDataSet(file_name): ''' Desc: 加载并解析数据,应用范围是数据文件是.txt文件 Args: file_name -- 文件名称,要解析的文件所在磁盘位置 Returns: dataMat -- 原始数据的特征 labelMat -- 原始数据的标签,也就是每条样本对应的类别 ''' # dataMat为原始数据, labelMat为原始数据的标签 dataMat = [] labelMat = [] fr = open(file_name) for line in fr.readlines(): lineArr = line.strip().split() if len(lineArr) == 1: continue # 这里如果就一个空的元素,则跳过本次循环 # 为了方便计算,我们将 X0 的值设为 1.0 ,也就是在每一行的开头添加一个 1.0 作为 X0 dataMat.append([1.0, float(lineArr[0]), float(lineArr[1])]) labelMat.append(int(lineArr[2])) return dataMat, labelMat # sigmoid跳跃函数(有所改进) def sigmoid(inX): # return 1.0 / (1 + exp(-inX)) # Tanh是Sigmoid的变形,与 sigmoid 不同的是,tanh 是0均值的。因此,实际应用中,tanh 会比 sigmoid 更好。 return 2 * 1.0/(1+exp(-2*inX)) - 1 # 随机梯度下降算法(随机化) def stocGradAscent1(dataMatrix, classLabels, numIter=100): ''' Desc: 改进版的随机梯度下降,使用随机的一个样本来更新回归系数。步长a在这里是自适应的(可变的) Args: dataMatrix -- 输入数据的数据特征(除去最后一列数据) classLabels -- 输入数据的类别标签(最后一列数据) numIter=150 -- 迭代次数 Returns: weights -- 得到的最佳回归系数 ''' m, n = shape(dataMatrix) weights = ones(n) # 创建与列数相同的矩阵的系数矩阵,所有的元素都是1,即回归系数的初始值 # 随机梯度, 循环150,观察是否收敛 for j in range(numIter): # [0, 1, 2 .. m-1] dataIndex = list(range(m)) for i in range(m): # i和j的不断增大,导致alpha的值不断减少,但是不为0 alpha = 4 / ( 1.0 + j + i ) + 0.0001 # alpha 会随着迭代不断减小,但永远不会减小到0,因为后边还有一个常数项0.0001 # 随机产生一个 0~len()之间的一个值 # random.uniform(x, y) 方法将随机生成下一个实数,它在[x,y]范围内,x是这个范围内的最小值,y是这个范围内的最大值。 randIndex = int(random.uniform(0, len(dataIndex))) #设置randIndex随机选取样本 # sum(dataMatrix[i]*weights)为了求 f(x)的值, f(x)=a1*x1+b2*x2+..+nn*xn h = sigmoid(sum(dataMatrix[dataIndex[randIndex]] * weights)) error = classLabels[dataIndex[randIndex]] - h # print weights, '__h=%s' % h, '__'*20, alpha, '__'*20, error, '__'*20, dataMatrix[randIndex] weights = weights + alpha * error * dataMatrix[dataIndex[randIndex]] del (dataIndex[randIndex]) return weights # 可视化展示 def plotBestFit(dataArr, labelMat, weights): ''' Desc: 将我们得到的数据可视化展示出来 Args: dataArr:样本数据的特征 labelMat:样本数据的类别标签,即目标变量 weights:回归系数 Returns: None ''' n = shape(dataArr)[0] xcord1 = [] ycord1 = [] xcord2 = [] ycord2 = [] for i in range(n): if int(labelMat[i]) == 1: xcord1.append(dataArr[i, 1]) ycord1.append(dataArr[i, 2]) else: xcord2.append(dataArr[i, 1]) ycord2.append(dataArr[i, 2]) fig = plt.figure() ax = fig.add_subplot(111) ax.scatter(xcord1, ycord1, s=30, c='red', marker='s') ax.scatter(xcord2, ycord2, s=30, c='green') x = arange(-3.0, 3.0, 0.1) """ y的由来,卧槽,是不是没看懂? 首先理论上是这个样子的。 dataMat.append([1.0, float(lineArr[0]), float(lineArr[1])]) w0*x0+w1*x1+w2*x2=f(x) x0最开始就设置为1叻, x2就是我们画图的y值,而f(x)被我们磨合误差给算到w0,w1,w2身上去了 所以: w0+w1*x+w2*y=0 => y = (-w0-w1*x)/w2 """ y = (-weights[0] - weights[1] * x) / weights[2] ax.plot(x, y) plt.xlabel('X') plt.ylabel('Y') plt.show() #测试主函数 def simpleTest(): # 1.收集并准备数据 path = "E:ML_datalogistic.txt" dataMat, labelMat = loadDataSet(path) # print dataMat, '--- ', labelMat # 2.训练模型, f(x)=a1*x1+b2*x2+..+nn*xn中 (a1,b2, .., nn).T的矩阵值 # 因为数组没有是复制n份, array的乘法就是乘法 dataArr = array(dataMat) # print dataArr # weights = gradAscent(dataArr, labelMat) # weights = stocGradAscent0(dataArr, labelMat) weights = stocGradAscent1(dataArr, labelMat) # print '*'*30, weights # 数据可视化 plotBestFit(dataArr, labelMat, weights) if __name__ == '__main__': simpleTest()
书中的示例是从疝气病预测病马的死亡率:
只要在上面的基础加数据预处理代码即可,完整如下:

#!/usr/bin/env python #-*-coding:utf-8 -*- ''' 基于随机梯度上升优化算法的逻辑回归。 里面包含两个测试,一个是简单的,一个是病马预测 ''' from numpy import * import matplotlib.pyplot as plt # 解析数据 def loadDataSet(file_name): ''' Desc: 加载并解析数据,应用范围是数据文件是.txt文件 Args: file_name -- 文件名称,要解析的文件所在磁盘位置 Returns: dataMat -- 原始数据的特征 labelMat -- 原始数据的标签,也就是每条样本对应的类别 ''' # dataMat为原始数据, labelMat为原始数据的标签 dataMat = [] labelMat = [] fr = open(file_name) for line in fr.readlines(): lineArr = line.strip().split() if len(lineArr) == 1: continue # 这里如果就一个空的元素,则跳过本次循环 # 为了方便计算,我们将 X0 的值设为 1.0 ,也就是在每一行的开头添加一个 1.0 作为 X0 dataMat.append([1.0, float(lineArr[0]), float(lineArr[1])]) labelMat.append(int(lineArr[2])) return dataMat, labelMat # sigmoid跳跃函数(有所改进) def sigmoid(inX): # return 1.0 / (1 + exp(-inX)) # Tanh是Sigmoid的变形,与 sigmoid 不同的是,tanh 是0均值的。因此,实际应用中,tanh 会比 sigmoid 更好。 return 2 * 1.0/(1+exp(-2*inX)) - 1 # 随机梯度下降算法(随机化) def stocGradAscent1(dataMatrix, classLabels, numIter=100): ''' Desc: 改进版的随机梯度下降,使用随机的一个样本来更新回归系数。步长a在这里是自适应的(可变的) Args: dataMatrix -- 输入数据的数据特征(除去最后一列数据) classLabels -- 输入数据的类别标签(最后一列数据) numIter=150 -- 迭代次数 Returns: weights -- 得到的最佳回归系数 ''' m, n = shape(dataMatrix) weights = ones(n) # 创建与列数相同的矩阵的系数矩阵,所有的元素都是1,即回归系数的初始值 # 随机梯度, 循环150,观察是否收敛 for j in range(numIter): # [0, 1, 2 .. m-1] dataIndex = list(range(m)) for i in range(m): # i和j的不断增大,导致alpha的值不断减少,但是不为0 alpha = 4 / ( 1.0 + j + i ) + 0.0001 # alpha 会随着迭代不断减小,但永远不会减小到0,因为后边还有一个常数项0.0001 # 随机产生一个 0~len()之间的一个值 # random.uniform(x, y) 方法将随机生成下一个实数,它在[x,y]范围内,x是这个范围内的最小值,y是这个范围内的最大值。 randIndex = int(random.uniform(0, len(dataIndex))) #设置randIndex随机选取样本 # sum(dataMatrix[i]*weights)为了求 f(x)的值, f(x)=a1*x1+b2*x2+..+nn*xn h = sigmoid(sum(dataMatrix[dataIndex[randIndex]] * weights)) error = classLabels[dataIndex[randIndex]] - h # print weights, '__h=%s' % h, '__'*20, alpha, '__'*20, error, '__'*20, dataMatrix[randIndex] weights = weights + alpha * error * dataMatrix[dataIndex[randIndex]] del (dataIndex[randIndex]) return weights # 可视化展示 def plotBestFit(dataArr, labelMat, weights): ''' Desc: 将我们得到的数据可视化展示出来 Args: dataArr:样本数据的特征 labelMat:样本数据的类别标签,即目标变量 weights:回归系数 Returns: None ''' n = shape(dataArr)[0] xcord1 = [] ycord1 = [] xcord2 = [] ycord2 = [] for i in range(n): if int(labelMat[i]) == 1: xcord1.append(dataArr[i, 1]) ycord1.append(dataArr[i, 2]) else: xcord2.append(dataArr[i, 1]) ycord2.append(dataArr[i, 2]) fig = plt.figure() ax = fig.add_subplot(111) ax.scatter(xcord1, ycord1, s=30, c='red', marker='s') ax.scatter(xcord2, ycord2, s=30, c='green') x = arange(-3.0, 3.0, 0.1) """ y的由来,卧槽,是不是没看懂? 首先理论上是这个样子的。 dataMat.append([1.0, float(lineArr[0]), float(lineArr[1])]) w0*x0+w1*x1+w2*x2=f(x) x0最开始就设置为1叻, x2就是我们画图的y值,而f(x)被我们磨合误差给算到w0,w1,w2身上去了 所以: w0+w1*x+w2*y=0 => y = (-w0-w1*x)/w2 """ y = (-weights[0] - weights[1] * x) / weights[2] ax.plot(x, y) plt.xlabel('X') plt.ylabel('Y') plt.show() # ----------------------------------------------------------------------------------------------------- # 测试主函数 def simpleTest(): # 1.收集并准备数据 path = "E:ML_datalogistic.txt" dataMat, labelMat = loadDataSet(path) # print dataMat, '--- ', labelMat # 2.训练模型, f(x)=a1*x1+b2*x2+..+nn*xn中 (a1,b2, .., nn).T的矩阵值 # 因为数组没有是复制n份, array的乘法就是乘法 dataArr = array(dataMat) # print dataArr # weights = gradAscent(dataArr, labelMat) # weights = stocGradAscent0(dataArr, labelMat) weights = stocGradAscent1(dataArr, labelMat) # print '*'*30, weights # 数据可视化 plotBestFit(dataArr, labelMat, weights) # -------------------------------------------------------------------------------- # 从疝气病症预测病马的死亡率 # 分类函数,根据回归系数和特征向量来计算 Sigmoid的值 def classifyVector(inX, weights): ''' Desc: 最终的分类函数,根据回归系数和特征向量来计算 Sigmoid 的值,大于0.5函数返回1,否则返回0 Args: inX -- 特征向量,features weights -- 根据梯度下降/随机梯度下降 计算得到的回归系数 Returns: 如果 prob 计算大于 0.5 函数返回 1 否则返回 0 ''' prob = sigmoid(sum(inX * weights)) if prob > 0.5: return 1.0 else: return 0.0 # 打开测试集和训练集,并对数据进行格式化处理 def colicTest(): ''' Desc: 打开测试集和训练集,并对数据进行格式化处理 Args: None Returns: errorRate -- 分类错误率 ''' path_train='E:ML_dataMLiA_SourceCodemachinelearninginactionCh05horseColicTraining.txt' path_test='E:ML_dataMLiA_SourceCodemachinelearninginactionCh05horseColicTest.txt' frTrain = open(path_train) frTest = open(path_test) trainingSet = [] trainingLabels = [] # 解析训练数据集中的数据特征和Labels # trainingSet 中存储训练数据集的特征,trainingLabels 存储训练数据集的样本对应的分类标签 for line in frTrain.readlines(): currLine = line.strip().split(' ') lineArr = [] for i in range(21): lineArr.append(float(currLine[i])) trainingSet.append(lineArr) trainingLabels.append(float(currLine[21])) # 使用 改进后的 随机梯度下降算法 求得在此数据集上的最佳回归系数 trainWeights trainWeights = stocGradAscent1(array(trainingSet), trainingLabels, 500) # trainWeights = stocGradAscent0(array(trainingSet), trainingLabels) errorCount = 0 numTestVec = 0.0 # 读取 测试数据集 进行测试,计算分类错误的样本条数和最终的错误率 for line in frTest.readlines(): numTestVec += 1.0 currLine = line.strip().split(' ') lineArr = [] for i in range(21): lineArr.append(float(currLine[i])) if int(classifyVector(array(lineArr), trainWeights)) != int( currLine[21]): errorCount += 1 errorRate = (float(errorCount) / numTestVec) print("the error rate of this test is: %f" % errorRate) return errorRate # 调用 colicTest() 10次并求结果的平均值 def multiTest(): numTests = 10 errorSum = 0.0 for k in range(numTests): errorSum += colicTest() print("after %d iterations the average error rate is: %f" % (numTests, errorSum / float(numTests))) if __name__ == '__main__': # simpleTest() multiTest()
其结果大概是错误率32%左右
上面是书中的自写模块,更详细的可以参考这里。下面是sklearn中的模块进行逻辑回归预测,还是针对病马数据集,注意这里的病马数据集是已经处理过的:
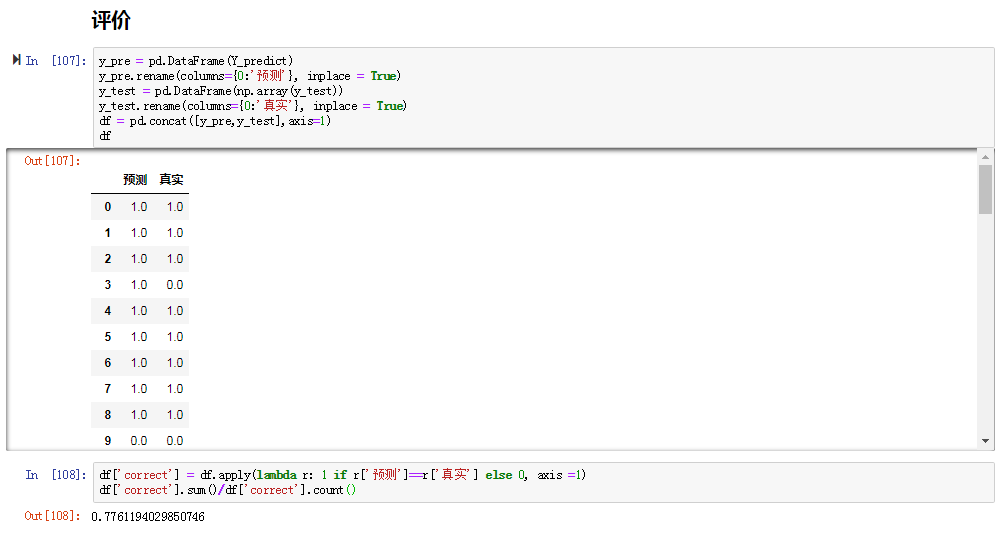
这里隐去了数据标准化,因为自己写的模块中也没有数据标准化,方便对比,其实数据标准化后的结果反而还差一点。至于建模函数LogisticRegressionCV的选择(有三个逻辑回归建模函数)以及里面的参数解释可以看这里。
