在真实的世界中,缺失数据是经常出现的,并可能对分析的结果造成影响。在R中,经常使用VIM(Visualization and Imputation of Missing values)包来对缺失值进行可视化和插补。在使用VIM绘图时,有些绘图函数会对缺失值会自动进行插补。
缺失数据的分类:
- MCAR(完全随机缺失):若变量的缺失数据与其他任何观测或未观测的变量都不相关,则数据为MCAR.。
- MAR(随机缺失):若变量的缺失数据与其他观测变量相关,与未观测变量无关,则数据缺失是随机缺失。
- NMAR(非随机缺失):若缺失数据不属于MCAR和MAR,则数据是非随机缺失。
大部分处理缺失数据的方法都是假定数据是MCAR或MAR。
一,识别缺失值
R使用NA代表缺失值,NaN(不是一个数)代表不可能的值,符号Inf和-Inf分别代表正无穷和负无穷。
函数is.na()、is.nan()和is.infinite()分别用来识别缺失值,不可能值和无穷值。
函数complete.cases() 可以用来识别矩阵或数据框中的没有缺失值的行,若每行有一个或多个缺失值,则返回FALSE。注意,complete.cases()仅把NA和Nan识别为缺失值,无穷值(Inf和-Inf)被当作有效值。
二,探索缺失值的模式
在决定如何处理缺失数据前,了解哪些变量有缺失值、数目有多少、是什么组合等信息,是非常有用的。
1,列表显示缺失值
mice包中的md.pattern()函数可以生成一个以矩阵或数据框形式展示缺失值模式,0表示变量的列中存在缺失值,1则表示没有缺失值。注意,md.pattern()函数仅把NA识别为缺失值。
md.pattern(x, plot = TRUE)
例如,以VIM包提供的哺乳动物的睡眠数据(sleep,基础安装包中还有一个描述药效的sleep数据集)。
library(VIM) library(mice) data(sleep,package='VIM') md.pattern(sleep, FALSE) BodyWgt BrainWgt Pred Exp Danger Sleep Span Gest Dream NonD 42 1 1 1 1 1 1 1 1 1 1 0 9 1 1 1 1 1 1 1 1 0 0 2 3 1 1 1 1 1 1 1 0 1 1 1 2 1 1 1 1 1 1 0 1 1 1 1 1 1 1 1 1 1 1 0 1 0 0 3 1 1 1 1 1 1 1 0 0 1 1 2 2 1 1 1 1 1 0 1 1 1 0 2 2 1 1 1 1 1 0 1 1 0 0 3 0 0 0 0 0 4 4 4 12 14 38
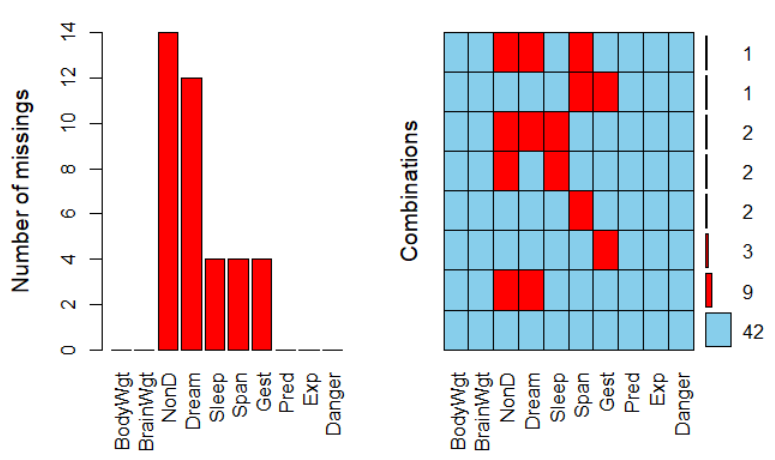
显示的结果中,模式是列的组合,第一列表示模式的数量,最后一列表示行中包含缺失值的列的数量,最后一行表示列中缺失值的数量。
比如,对于第一行,缺失值的数量是0,这样的数据行在数据集中有42个。对于第二行,包含缺失值的列(Dream和NonD 同时缺失)数量是2,这样的行在数据集中有9个。
2,用图形探究缺失数据
VIM包提供了大量能可视化缺失值模式的函数,aggr()、marginplot()和scattMiss()。
library(VIM) data(sleep,package='VIM') aggr(sleep,prop=FALSE,numbers=TRUE)
aggr重要参数注释:
- prob:当为TRUE时,显示为缺失值的占比;当为FALSE时,显示为缺失值的数量;
- numbers:是否显示数值,默认为FALSE,不显示缺失值的占比或数量。
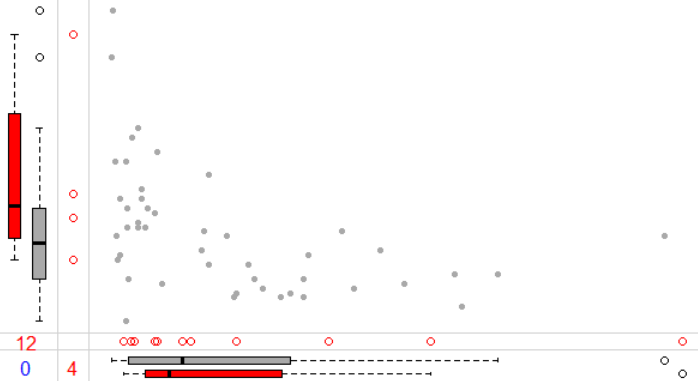
使用散点图来可视化两个变量的异常值:
library(VIM) data(sleep,package='VIM') marginplot(sleep[,c('Gest','Dream')],pch = c(20,1),col=c('darkgray','red','blue'))
对于包含两个变量的数据点,两个变量表示一个点(x,y),如果x和y都不包含缺失值,称作有效点;如果x或x中有一个包含缺失值,叫做单变量缺失,对于单变量缺失,该函数会自动插补;如果x和y都包含缺失值,叫做不可用点。
marginplot重要参数注释:
- x:包含两列的数据框,第一列是x轴,第二列是y轴,
- pch:点的形状,是个双值的向量,分别用于设置用于显示包含缺失值/插补值的点的形状,和有效点的形状。
- col:向量,用于设置绘图中使用的颜色。第一个颜色用于绘制有效点的散点图和箱图,第二个颜色用于绘制单变量缺失的散点图和箱图;第三个图颜色用于绘制不可用点。
红色箱图表示包含插补值之后的分布,灰色箱图表示有效点的分布。红色的空心点是插补之后的点。
3,用相关性探索缺失值
用指示变量代替数据集中的数据(1代表缺失,0代表存在),这样生成的矩阵有时被称作影子矩阵,求这些指示变量之间的相关性,有助于观察哪些变量经常一起缺失。
library(VIM) data(sleep,package='VIM') x <- as.data.frame(abs(is.na(sleep))) y <- x[which(apply(x,2,sum)>0)] cor(y) NonD Dream Sleep Span Gest NonD 1.00000000 0.90711474 0.48626454 0.01519577 -0.14182716 Dream 0.90711474 1.00000000 0.20370138 0.03752394 -0.12865350 Sleep 0.48626454 0.20370138 1.00000000 -0.06896552 -0.06896552 Span 0.01519577 0.03752394 -0.06896552 1.00000000 0.19827586 Gest -0.14182716 -0.12865350 -0.06896552 0.19827586 1.00000000
可以看到,Dream和NonD常常一起缺失(r=0.91)。
使用影子矩阵和原始数据之间的相关性,可以探索数据缺失的类型。
cor(sleep,y,use='pairwise.complete.obs') NonD Dream Sleep Span Gest BodyWgt 0.22682614 0.22259108 0.001684992 -0.05831706 -0.05396818 BrainWgt 0.17945923 0.16321105 0.007859438 -0.07921370 -0.07332961 NonD NA NA NA -0.04314514 -0.04553485 Dream -0.18895206 NA -0.188952059 0.11699247 0.22774685 Sleep -0.08023157 -0.08023157 NA 0.09638044 0.03976464 Span 0.08336361 0.05981377 0.005238852 NA -0.06527277 Gest 0.20239201 0.05140232 0.159701523 -0.17495305 NA Pred 0.04758438 -0.06834378 0.202462711 0.02313860 -0.20101655 Exp 0.24546836 0.12740768 0.260772984 -0.19291879 -0.19291879 Danger 0.06528387 -0.06724755 0.208883617 -0.06666498 -0.20443928 Warning message: In cor(sleep, y, use = "pairwise.complete.obs") : 标准差为零
在这个相关系数矩阵中,行是可观测的变量,列为表示缺失的指示变量。注意,表中的相关系数都不大,表明数据是MCAR的可能性比较小,更可能是MAR。
三,缺失数据的处理
识别缺失数据的数目、分布和模式,有两个目的:分析生成缺失数据的潜在机制,评价缺失数据对回答实质性问题的影响。具体来讲,需要弄清楚以下几个问题:
- 缺失数据的比例有多大?
- 缺失数据是否集中在少数几个变量上,抑或广泛存在?
- 缺失是随机产生的吗?
- 缺失数据间的相关性或与可观测数据之间的相关性,是否可以表明产生缺失值的机制。
回答这些问题将有助于采用合适的方法来处理缺失数据。
- 如果缺失数据集中在几个相对不重要的变量上,那么可以删除这些变量。
- 如果有一小部分数据(如小于10%)随机分布在整个数据集中(MCAR),那么你可以删除存在缺失数据的行,而只分析数据完整的实例,这样扔可以得到可靠且有效的结果。
- 如果可以假定数据是MCAR或MAR,那么可以应用多重插补法来获得有效的结论。
1,推理恢复
根据变量之间的关系来填补或恢复缺失值,通过推理,数据的恢复可能是准确的或近似的。
2,行删除
把包含一个或多个缺失值的行删除,称作行删除法,或个案删除(case-wise deletion),大部分统计软件包默认采用的是行删除法。
newdata <- mydata[complete.cases(mydata),]
newdata <- na.omit(mydata)
当数据是MCAR(完整的观测只是全数据集的一个随机样本)时,采用行删除之后,也会得到可靠和有效的结论。
3,简单插补
简单插补,也叫均值插补,是用均值,中位数或众数来替换变量中缺失的值。将初始数据集中的属性分为数值属性和非数值属性来分别进行处理。
- 如果空值是数值型的,就根据该属性在其他所有对象的取值的平均值来填充该缺失的属性值;
- 如果空值是非数值型的,就根据统计学中的众数原理,用该属性在其他所有对象的取值次数最多的值(即出现频率最高的值)来补齐该缺失的属性值。
简单插补对于非MCAR的数据会产生有偏向的结果,适用于缺失数据的数量较小的数据集。均值插补是在低缺失率下首选的插补方法,缺点是不能反映缺失值的变异性;
四,插补法(MI)
插补法可以在一定程度上减少偏差,常用的插补法是回归插补、多重插补和热卡插补。
- 回归插补,要求变量间存在强的相关性;
- 多重插补(MCMC法),是在高缺失率下的首选插补方法,优点是考虑了缺失值的不确定性;
1,回归插补
基于完整的数据集,建立回归方程。对于包含空值的对象,将已知属性值代入方程来估计未知属性值,以此估计值来进行填充。当变量不是线性相关时会导致有偏差的估计。
2,多重插补
多重插补(MI)是一种基于重复模拟的处理缺失值的方法,它从一个包含缺失值的数据集中生成一组数据完整的数据集(即不包含缺失值的数据集,通常是3-10个)。每个完整数据集都是通过对原始数据中的缺失数据进行插补而生成的。在每个完整的数据集上引用标准的统计方法,最后,把这些单独的分析结果整合为一组结果。
多重插补法大致分为三步:
- 为每个空值产生一套可能的插补值,这些值反映了无响应模型的不确定性;每个值都被用来插补数据集中的缺失值,产生若干个完整数据集合。
- 每个插补数据集合都用针对完整数据集的统计方法进行统计分析。
- 对来自各个插补数据集的结果进行整合,产生最终的统计推断,这一推断考虑到了由于数据插补而产生的不确定性。该方法将空缺值视为随机样本,这样计算出来的统计推断可能受到空缺值的不确定性的影响。
R中可以利用Amelia、mice和mi包来执行这些操作。下图可以帮助理解mice包的操作过程。
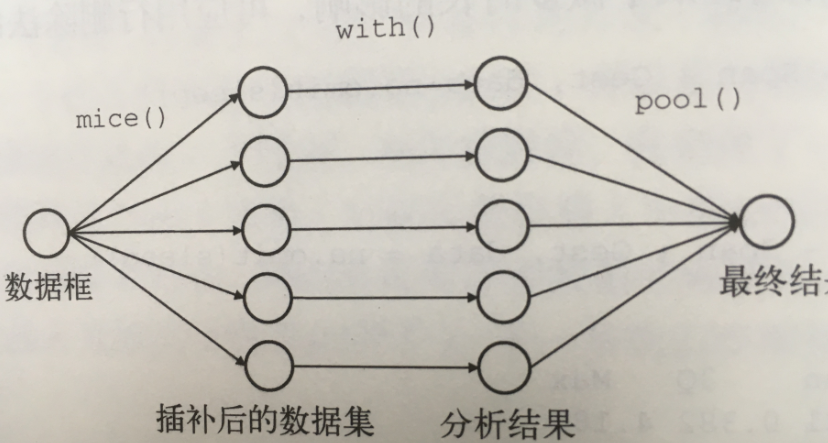
函数mice()首先从一个包含缺失数据的数据框开始,然后返回一个包含多个(默认为5个)完整数据集的对象。每个完整数据集都是通过对原始数据框中的缺失数据进行插补而生成的。由于插补有随机的成分,因此每个完整数据集都略有不同。然后,with()函数可依次对每个完整的数据集应用统计模型(如线性模型或广义线性模型),最后,pool()函数把这些单独的分析结果整合为一组结果。最终模型的标准差和p值都准确地反映出由于缺失值和多重插补而产生的不确定性。
library(mice) imp <- mice(data seed=m) fit <- with( imp, lm()) po<- pool(fit) summary(po)
mice()函数如何插补缺失值?
缺失值的插补法通过Gibbs抽样完成,每个包含缺失值的变量都默认可通过数据集中的其他变量预测的来,于是这些预测方程便可用于预测数据的有效值。该方程不断迭代直到所有的缺失值都收敛为止。
默认情况下,预测的均值用于替换连续性变量中的缺失数据,而Logistic或多元Logisitcs回归分别用于替换二值目标变量(两水平因子)或多值变量(多水平因子)。
mice包中的mice函数:
mice(data, m = 5, method = NULL, defaultMethod = c("pmm", "logreg", "polyreg", "polr"), seed = NA, ...)
重要参数注释:
- m:多重插补的次数,默认值是5
- method:用于每列插补的算法
- defaultMethod:默认的插补算法
- seed:随机数种子
3,热卡填充(Hot deck imputation,或就近补齐)
对于一个包含空值的对象,热卡填充法在完整数据中找到一个与它最相似的对象,然后用这个相似对象的值来进行填充。不同的问题可能会选用不同的标准来对相似进行判定。该方法概念上很简单,且利用了数据间的关系来进行空值估计。这个方法的缺点在于难以定义相似标准,主观因素较多。
参考文档: