Multi-scale Cell Instance Segmentation with Keypoint Graph Based Bounding Boxes
细胞分割的难点
-
细胞边界的低分辨率
-
背景杂质
-
细胞粘附
-
细胞聚集
相关工作
DCAN 提出将细胞轮廓叠在分割结果上,由于touching cell间的模糊轮廓,这会产生over-segmentation
STARDIST 提出使用凸多边形来分隔细胞,但是它假设细胞形状应该是凸的
CosineEmbedding 提出通过对pixel-embeddings进行聚类来分离细胞,但是这会导致单独聚类的细胞产生大量误报false-positive
box-free的实例分割缺乏全局目标特征,box-based的方法先用边界框定位,再在该框内进行单独的细胞分割任务,不仅能学到局部像素信息,还能学到全局目标特征,但是box-based的maskrcnn有正负anchor boxes不平衡的问题
方法
边界框生成
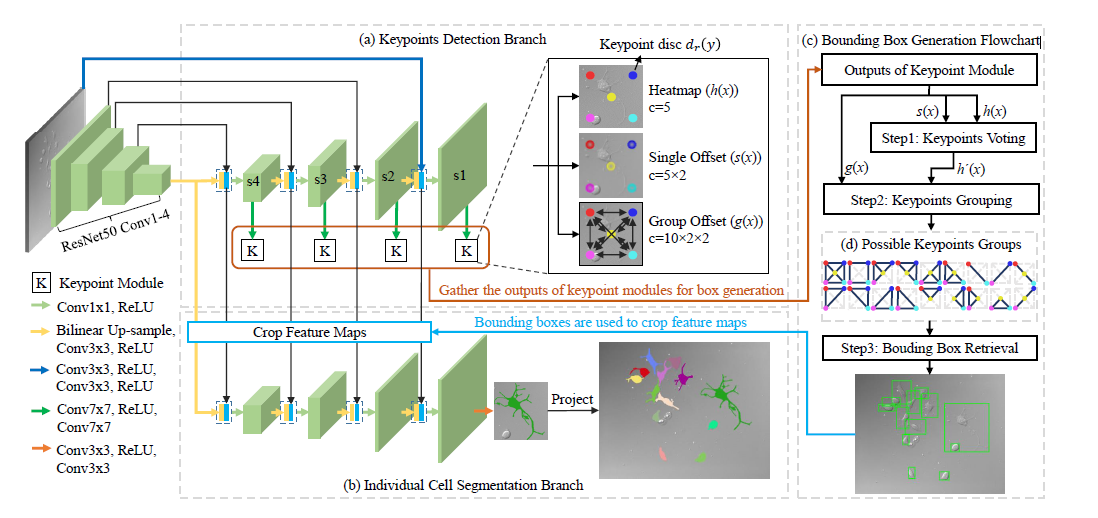
细胞分割
在获得输入图像中所有细胞实例的边界框之后,我们为每个细胞实例执行单个细胞分割。 像u-net一样,我们将浅层的特征图与深层的特征相结合以利用高级语义和低级图像细节。 具体来说是从backbone中裁剪多尺度特征图,然后对裁剪的细胞块进行自下而上的分割
用于裁剪的细胞补丁。 请注意,我们特意采用对单个细胞分割的分支用于细胞分割,而不是直接重复使用s1处的特征图。 我们的动机是利用分支机构来指导模型消除相邻细胞的干扰,特别是对于形状不规则的细胞。
结论分析
MASK R-CNN 的RoI Align操作,会导致其无法预测细长的目标。
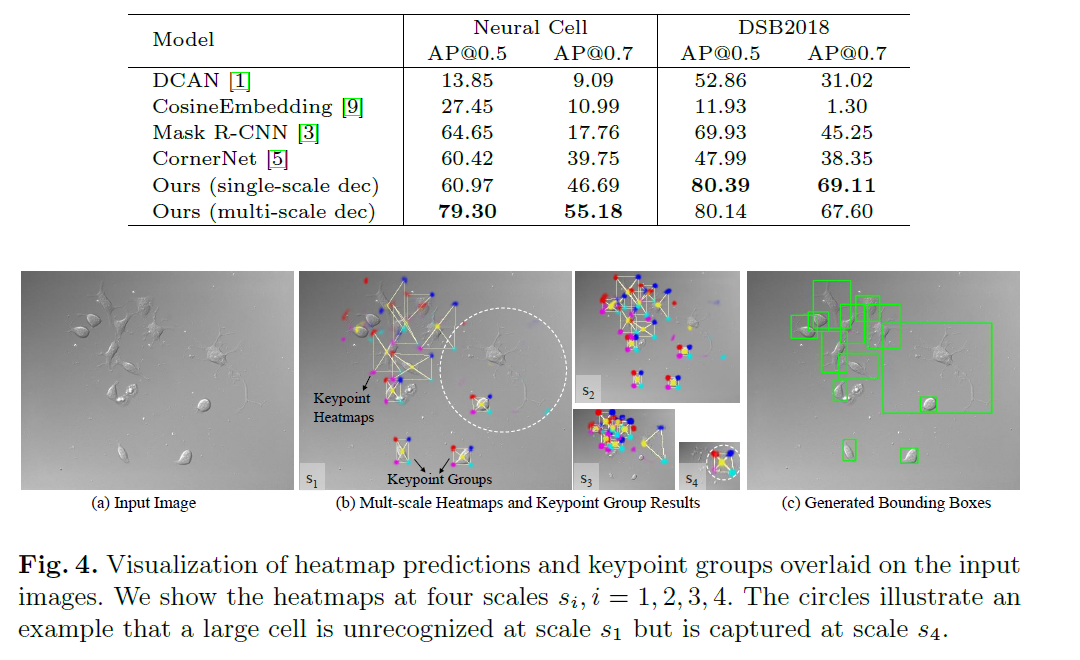
Multi-scale比single-scale具有更多不同尺度的感受野,兼顾大细胞和小细胞。