单细胞流程跑了不少,但依旧看不懂结果,是该好好补补了。
有些人可能会误会,觉得单细胞的RNA-seq数据很好分析,跟分析常规的RNA-seq应该没什么区别。今天的这篇文章2015年3月发表在Nature Genetics Review上,专门说明了一下单细胞RNA测序数据在数据分析和计算上的挑战(虽然已经过去1年多了,这里指出的问题和挑战仍然是不过时的,至于这些问题和挑战现在是不是完美解决了,这里就暂且先不讨论了。)。
主要说了以下问题:
1. 单细胞RNA测序 (single cell RNA sequencing,以下简称scRNA-seq)数据质控和归一化(Normalization),其实主要是归一化。
次要还涉及了以下问题:
2. 单细胞测序应该测多少深度合适,即测几个G的数据量。
3. 批次效应(batch effect)的问题。
另外,我在另一篇文章中看到的,也很有意思:
很多基因表达值为0的问题,当然这个也可以归类到归一化的问题中去。
1. spike-in. 在说明问题之前,首先要明确一下实验设计。有一个方法,which is strongly recommended for all scRNA-seq实验,那就是使用spike-in,而spike-in最广泛的就是ERCC。有些实验的protocol,使用3‘或5’端的特征序列(unique melocular identifier, UMI)来当barcode,但还是同时加上spike-in的好,加上spike-in之后,这种实验方法可以帮助后期分析绕开扩增中产生的biases这一问题,而扩增biases是技术不稳定的最主要的一个来源。所以,强调一点,单细胞RNA-seq要做spike-in.
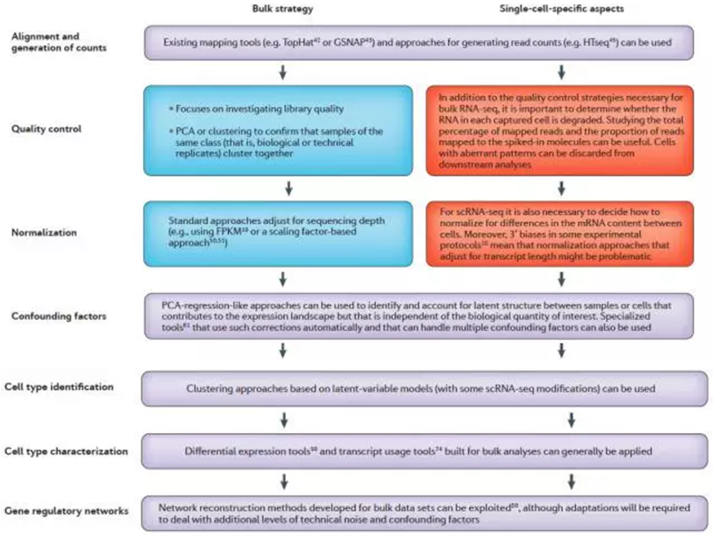
2. 分析pipeline. 之前针对常规RNA-seq的分析pipeline大部分还是可以公用的,比如:原始数据的回贴就可以沿用TopHat或者GSNAP等,数read counts还是可以用HTseq,样本的聚类,差异表达分析等都可以沿用常规RNA-seq的pipeline。
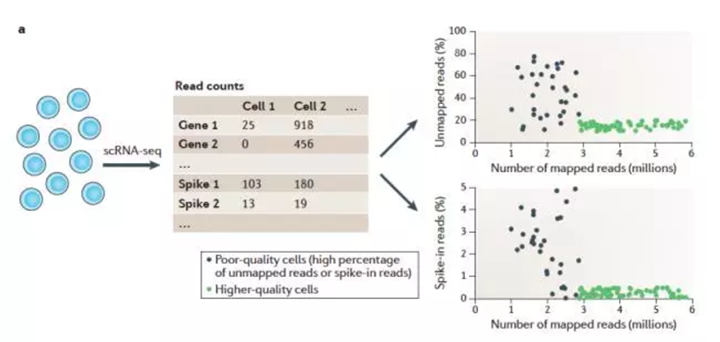
3. 但是QC和Normalization这两步,单细胞测序要格外小心。QC的时候,除了要注意常规的RNA-seq的QC条目,单细胞中非常重要的一点是还要确认RNA是否有降解。这点可以通过看总的回贴片段及回贴到spike-in上的片段的比例。(这里其实有个问题:如果RNA降解很严重,还能够反转扩增成功吗?我个人猜测可能会比较难。)
总结下,单细胞的QC可以分成以下三步:Fastqc,HTseq(数reads后,看reads回贴在哪里,下图展示的是统计整理之后的样子)及PCA。
以下插播一段题外话:PCA的图可以长成下面这样。PCA挺有用的,不管是单细胞测序还是常规测序,特别是样本量多的时候。
(图片来源:Petropoulos et al., 2016, Cell 165, 1012–1026)
4. Normalization. 我们对常规的RNA-seq做归一化有RPKM,FPKM或者read counts,且这种归一化基于一个假设,即这些细胞中的RNA的量是一样多的。但是,如果没有spike-in的话,我们没有办法知道一个细胞里面到底有多少RNA,也就没有办法做归一化。然而加入spike-in之后,细胞大小测序深度的不一致也会使得常用的归一化方法不适用。这篇综述提到的方法是Philip Brennecke 2013年发表在nature method上的,首先根据测序深度和细胞中的RNA的量对read counts进行归一化,然后再针对spike-in和自己本身的RNA计算样本间的变异系数。不过这个方法,后来又被另外一个方法(也是发表在nature method上的
,Dominic Grün,2014年)嫌弃了。尚无定论。
5. 测序深度。这个每个人也有每个人的做法。基本原则是:
sequenced the library to a sufficient depth to ensure that each cDNA molecule is observed at least once. 看上去有点玄乎(个人感觉:一般6-8G),细胞量越少,测序深一点,这两者有个balance。
6. batch effect. Batch effect的问题在scRNA-seq中更为显著、严重。
One way to overcome this problem is to increase the number of biological replicates. 一种办法是增加重复样。有没有别的办法,文章没有提。