10X Genomics已经广泛应用于单细胞测序、组装领域,现在也是火的不行。
10X Genomics原理
通过将来自相同DNA片段(10-100kb)的reads加上相同的barcode,然后在illumina平台上进行测序,从而实现长片段的测序。其基本原理是同一长片段的reads会具有同样的标签,称为linked-reads,利用这些barcode的信息,可将短reads拼接为长reads。这样的linked-reads可进行结构变异检测及单倍型定相的分析。
总结:10X Genomics发明了一种独特的barcode建库技术,能将同一种barcode的reads连成linked reads从而提高了reads的长度。但10X Genomics的测序之本仍然是illumina,GC偏向性是硬伤。同时barcode技术有一定的环境限制。
附:未来基因组组装的标配为“PacBio纯三代组装contig“+”光学图谱进行纠错与super scaffold组装“+”遗传图谱或HiC进行染色体组装”。
DNA分析流程
RNA分析流程
转自:菲沙基因
2015年测序行业杀出一匹黑马,10X Genomics公司,他们开发了一个巧妙的方法,能使Illumina测序仪产生长片段数据。What?!100-300bp变成10-100kb,巨大的片段长度提升一时间吸引了无数人的眼球。提到长片段测序,永远都绕不开 PacBio。下面我们来一起看看,10X Genomics是如何挑战PacBio,而PacBio又是如何应对10X Genomics的“来势汹汹”。
1巧妙的平台互补
GEM反应体系中的DNA模板打断后与Barcode结合形成适合建库的小片段,配合Illumina测序得到原始数据。GemCode平台配套的数据分析软件将Barcode标记能力与短读取数据相结合,产生了独特的一类数据类型:Linked-Reads,利用Barcode标记信息将同一模版DNA来源的序列信息进行拼接,从而获得大片段遗传信息(10-100kb)。10X Genomics官方宣传这种Linked-Reads数据让研究人员能够以前所未有的水平检测单体型和结构变异。同时,缺失、重复和重排等具有挑战性的结构变异也能够自信鉴定,从而更准确地了解基因组复杂性。Fig.1 GEMcode平台原理 10X Genomics 公司开发的GemCode平台是一套分子条形码和分析系统,由仪器、试剂盒和信息学软件组成。目前,它只与Illumina测序系统兼容。GemCode技术的核心是对1 ng的DNA进行精确分区a,形成含有1条DNA模板链和特定相同的Barcode序列的微小反应体系(GEM,Gel Bead in Emulsion),不同GEM反应体系中的Barcode不同。
2惊人的组装效果
去年PacBio公司在《Nature Methods》上发表了SMRT测序组装人类基因组的成果,选用的样本是HapMap样品NA12878[1]。而在今年5月,10X Genomics很“偶然地”选择了同样的样本,结合short-read测序和10X的linked-read测序对人类基因组进行了高质量de novo组装,并且该成果也很“偶然地”发表于《Nature Methods》上。相同的样本,相似的长片段,给人带来无限联想,看来10X Genomics已经卯足了劲准备“开大”了。
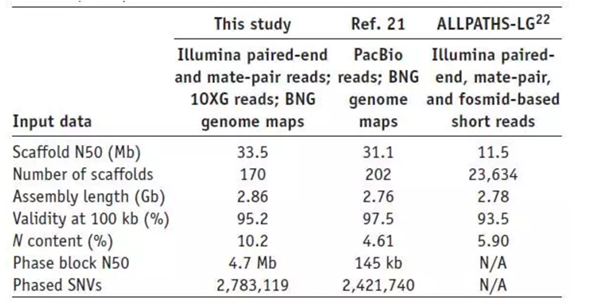
F ig.2 NA12878组装效果对比
在10X Genomics研究中,研究人员利用这种方法对人类HapMap样品NA12878进行组装和分相。最初的Illumina组装产生了超过14,000个scaffold,而N50为0.59 Mb。在混合组装后,scaffold数量降为170个,而N50大小达到33.5 Mb,相对之前提高了57倍[2]。从组装结果来看,不管是N50大小、组装长度还是scaffold数,10X Genomics的组装效果都要强于PacBio。这一局,看起来10X Genomics赢了,事实真的是这样吗,我们继续往下看。
3长片段优劣之争
一种新技术的出现,往往会伴随着争议。10X Genomics到底有没有它宣传的那么好,我们结合技术原理和文章来仔细分析一下。
1) 长片段的产生
简单来说,10X Genomics获得长片段的方法基于DNA精确分区和Barcode技术。将100kb长片段模板和Barcode混合组成一个GEM,打断后该体系中每个短片段都带有相同Barcode。使用Illumina平台对短片段进行测序,然后使用Barcode即可将短片段拼接成长片段。这种方法对DNA要求高,长期保存的样品往往无法使用。另外,一个文库中linked-read的长片段模板不一定会全部被扩增,所以该方法也需要构建不同大小的多个文库。相对而言,PacBio的长片段直接由测序读取,无需拼接,一致准确性和覆盖度都较高。
2) GC异常区域的检测
去年10月《Nature Review Genetics》的一篇文章中总结了Illumina、10X Genomics和PacBio技术在不同GC含量区域的覆盖度分布情况[3]。结果显示,虽然10X Genomic比Illumina测序覆盖效果有所提升,但是两者整体的一致覆盖度趋势相似,都呈拱形,即对高GC或低GC区域的覆盖效果都比较差。而PacBio在这方面效果良好,基本无偏倚。从本质上看,10X Genomics只是将Illumina短片段变长,并没有彻底消除Illumina平台GC偏好的问题。归根结底,是否进行PCR扩增是影响10X Genomics和PacBio长片段覆盖度的主要因素。
Fig.3 各测序平台不同GC含量区域覆盖度比较
3) 结构变异检测
既然都能得到长片段,那么各平台在SV检测方面的能力又怎样呢,我们通过两张图来对比一下。
Fig.4 SV检测
10X Genomics今年2月在《Nature Biotechnology》发表文章,主打结构变异检测和单体型分相。左图是10X Genomics关于缺失变异的检测结果,通过数据覆盖度的趋势拟合结构变异特征,从图中可以看出Chr8 39.3Mb处出现high-confidence deletion[4]。右图是PacBio去年《Nature Methods》文章中的sv检测结果[1],粉红部分代表deletion,灰色部分代表Inversion。我只想说,PacBio太凶残了有木有,10X Genomics只给出个趋势,你偏偏要画出准确结构!
最后说两句,Illumina测序是10X Genomics的根,Illumina做不到的,10X Genomics也无能为力。PacBio虽然贵,但是贵得值,毕竟花多少钱做多大事!
4参考文献
1. Pendleton M, Sebra R, Pang A W, et al. Assembly and diploid architecture of an individual human genome via single-molecule technologies.[J]. Nature Methods, 2015, 12(8).
2. Zheng G X, Lau B T, Schnall-Levin M, et al. Haplotyping germline and cancer genomes with high-throughput linked-read sequencing.[J]. Nature Biotechnology, 2016.
3. Chaisson M J P, Wilson R K, Eichler E E. Genetic variation and the de novo assembly of human genomes[J]. Nature Reviews Genetics, 2015, 16(11):627-40.
4. Mostovoy Y, Levy-Sakin M, Lam J, et al. A hybrid approach for de novo human genome sequence assembly and phasing.[J]. Nature Methods, 2016.