近期利用遥感影像进行路网提取,利用Unet网络进行图像分割
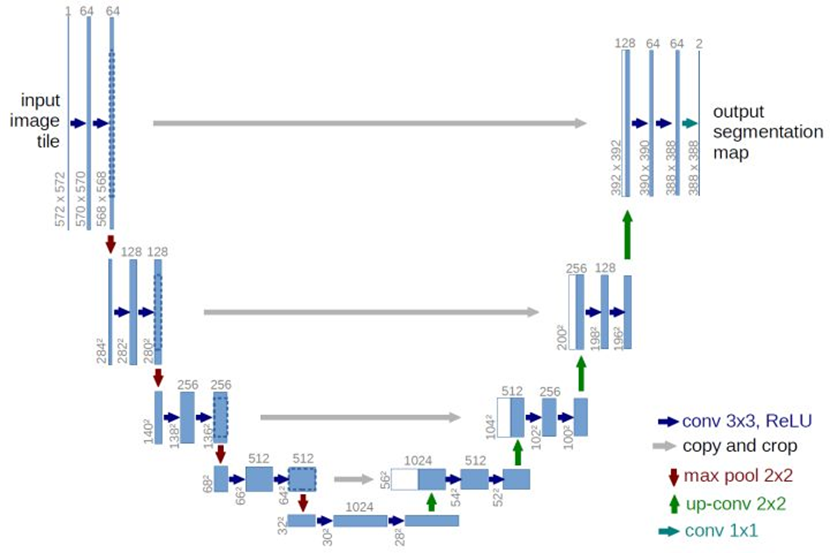
介绍如下:
U-net网络非常简单,前半部分作用是特征提取,后半部分是上采样。在一些文献中也把这样的结构叫做编码器-解码器结构。由于此网络整体结构类似于大写的英文字母U,故得名U-net。
U-net与其他常见的分割网络有一点非常不同的地方:U-net采用了完全不同的特征融合方式:拼接,U-net采用将特征在channel维度拼接在一起,形成更厚的特征。而FCN融合时使用的对应点相加,并不形成更厚的特征。
所以语义分割网络在特征融合时有两种办法:
1. FCN式的对应点相加,对应于TensorFlow中的tf.add()函数;
2. U-net式的channel维度拼接融合,对应于TensorFlow的tf.concat()函数,比较占显存。
2、上采样部分会融合特征提取部分的输出,这样做实际上是将多尺度特征融合在了一起,以最后一个上采样为例,它的特征既来自第一个卷积block的输出(同尺度特征),也来自上采样的输出(大尺度特征),这样的连接是贯穿整个网络的,你可以看到上图的网络中有四次融合过程,相对应的FCN网络只在最后一层进行融合。
链接:https://www.zhihu.com/question/269914775/answer/586501606
来源:知乎
著作权归作者所有。商业转载请联系作者获得授权,非商业转载请注明出处。
这个问题在面试医疗影像算法岗位的时候,偶尔会提到,我这里提供一些个人的思考。问题中有两个关键词,【UNet】和【医疗影像】,接下来我们一一分析这两个关键词。
首先我们说说【UNet】。
UNet最早发表在2015的MICCAI上,短短3年,引用量目前已经达到了4070,足以见得其影响力。而后成为大多做医疗影像语义分割任务的baseline,也启发了大量研究者去思考U型语义分割网络。而如今在自然影像理解方面,也有越来越多的语义分割和目标检测SOTA模型开始关注和使用U型结构,比如语义分割Discriminative Feature Network(DFN)(CVPR2018),目标检测Feature Pyramid Networks for Object Detection(FPN)(CVPR 2017)等。
我们言归正传,UNet只是一个网络结构的代号而已,我们究其细节,到底UNet是由哪些组件构成的呢?
UNet的结构,我认为有两个最大的特点,U型结构和skip-connection(如下图)。

UNet的encoder下采样4次,一共下采样16倍,对称地,其decoder也相应上采样4次,将encoder得到的高级语义特征图恢复到原图片的分辨率。
相比于FCN和Deeplab等,UNet共进行了4次上采样,并在同一个stage使用了skip connection,而不是直接在高级语义特征上进行监督和loss反传,这样就保证了最后恢复出来的特征图融合了更多的low-level的feature,也使得不同scale的feature得到了的融合,从而可以进行多尺度预测和DeepSupervision。4次上采样也使得分割图恢复边缘等信息更加精细。
其次我们聊聊【医疗影像】,医疗影像有什么样的特点呢(尤其是相对于自然影像而言)?
1.图像语义较为简单、结构较为固定。我们做脑的,就用脑CT和脑MRI,做胸片的只用胸片CT,做眼底的只用眼底OCT,都是一个固定的器官的成像,而不是全身的。由于器官本身结构固定和语义信息没有特别丰富,所以高级语义信息和低级特征都显得很重要(UNet的skip connection和U型结构就派上了用场)。举两个例子直观感受下。
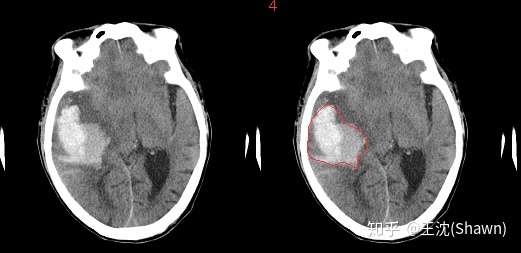
A.脑出血. 在CT影像上,高密度的区域就大概率是一块出血,如下图红色框区域。
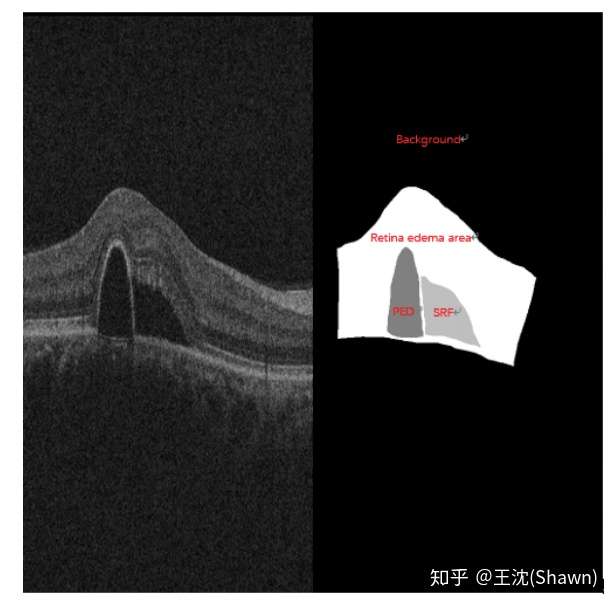
B.眼底水肿。左图原图,右图标注(不同灰度值代表不同的水肿病变区域)。在OCT上,凸起或者凹陷的区域就大概率是一个水肿病变的区域。
2.数据量少。医学影像的数据获取相对难一些,很多比赛只提供不到100例数据。所以我们设计的模型不宜多大,参数过多,很容易导致过拟合。
原始UNet的参数量在28M左右(上采样带转置卷积的UNet参数量在31M左右),而如果把channel数成倍缩小,模型可以更小。缩小两倍后,UNet参数量在7.75M。缩小四倍,可以把模型参数量缩小至2M以内,非常轻量。个人尝试过使用Deeplab v3+和DRN等自然图像语义分割的SOTA网络在自己的项目上,发现效果和UNet差不多,但是参数量会大很多。
3.多模态。相比自然影像,医疗影像比较有趣和不同的一点是,医疗影像是具有多种模态的。以ISLES脑梗竞赛为例,其官方提供了CBF,MTT,CBV,TMAX,CTP等多种模态的数据。
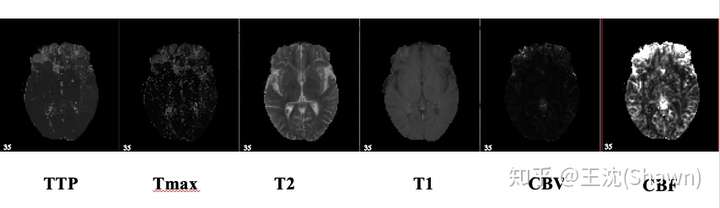
这就需要我们更好的设计网络去提取不同模态的特征feature。这里提供两篇论文供大家参考。
Joint Sequence Learning and Cross-Modality Convolution for 3D Biomedical Segmentation(CVPR 2017) ,
Dense Multi-path U-Net for Ischemic Stroke Lesion Segmentation in Multiple Image Modalities.
4.可解释性重要。由于医疗影像最终是辅助医生的临床诊断,所以网络告诉医生一个3D的CT有没有病是远远不够的,医生还要进一步的想知道,病灶在哪一层,在哪一层的哪个位置,分割出来了吗,能求体积嘛?同时对于网络给出的分类和分割等结果,医生还想知道为什么,所以一些神经网络可解释性的trick就有用处了,比较常用的就是画activation map。看网络的哪些区域被激活了,如下图。
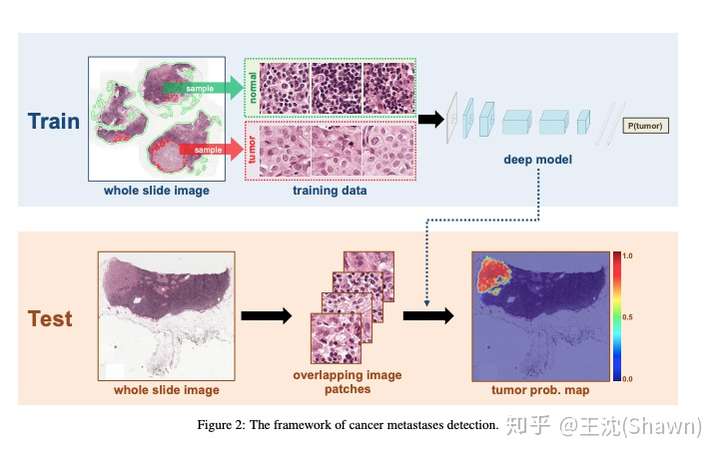
这里推荐两篇工作:
老师的Learning Deep Features for Discriminative Localization(CVPR2016)和其实验室同学的 Deep Learning for Identifying Metastatic Breast Cancer(上图的出处)BTW:没有偏题的意思,只是觉得医疗影像的特点和本问题息息相关,就一起总结了。
最后提一个问题,引发关注医疗影像的同学们思考和讨论?
前面有提到,UNet成为大多做医疗影像语义分割任务的baseline,也启发了大量研究者去思考U型语义分割网络。 那UNet还有什么不足呢?欢迎大家关注我的项目UNet-family,寻找答案。
ShawnBIT/UNet-family同时也推荐,UNet++作者
的分享:研习Unet。 周纵苇:研习U-Net
好的,在高铁上完成了这个一直想回答的问题,开心~
链接:https://www.zhihu.com/question/269914775/answer/586501606
来源:知乎
著作权归作者所有。商业转载请联系作者获得授权,非商业转载请注明出处。
这个问题在面试医疗影像算法岗位的时候,偶尔会提到,我这里提供一些个人的思考。问题中有两个关键词,【UNet】和【医疗影像】,接下来我们一一分析这两个关键词。
首先我们说说【UNet】。
UNet最早发表在2015的MICCAI上,短短3年,引用量目前已经达到了4070,足以见得其影响力。而后成为大多做医疗影像语义分割任务的baseline,也启发了大量研究者去思考U型语义分割网络。而如今在自然影像理解方面,也有越来越多的语义分割和目标检测SOTA模型开始关注和使用U型结构,比如语义分割Discriminative Feature Network(DFN)(CVPR2018),目标检测Feature Pyramid Networks for Object Detection(FPN)(CVPR 2017)等。
我们言归正传,UNet只是一个网络结构的代号而已,我们究其细节,到底UNet是由哪些组件构成的呢?
UNet的结构,我认为有两个最大的特点,U型结构和skip-connection(如下图)。

UNet的encoder下采样4次,一共下采样16倍,对称地,其decoder也相应上采样4次,将encoder得到的高级语义特征图恢复到原图片的分辨率。
相比于FCN和Deeplab等,UNet共进行了4次上采样,并在同一个stage使用了skip connection,而不是直接在高级语义特征上进行监督和loss反传,这样就保证了最后恢复出来的特征图融合了更多的low-level的feature,也使得不同scale的feature得到了的融合,从而可以进行多尺度预测和DeepSupervision。4次上采样也使得分割图恢复边缘等信息更加精细。
其次我们聊聊【医疗影像】,医疗影像有什么样的特点呢(尤其是相对于自然影像而言)?
1.图像语义较为简单、结构较为固定。我们做脑的,就用脑CT和脑MRI,做胸片的只用胸片CT,做眼底的只用眼底OCT,都是一个固定的器官的成像,而不是全身的。由于器官本身结构固定和语义信息没有特别丰富,所以高级语义信息和低级特征都显得很重要(UNet的skip connection和U型结构就派上了用场)。举两个例子直观感受下。
A.脑出血. 在CT影像上,高密度的区域就大概率是一块出血,如下图红色框区域。

B.眼底水肿。左图原图,右图标注(不同灰度值代表不同的水肿病变区域)。在OCT上,凸起或者凹陷的区域就大概率是一个水肿病变的区域。

2.数据量少。医学影像的数据获取相对难一些,很多比赛只提供不到100例数据。所以我们设计的模型不宜多大,参数过多,很容易导致过拟合。
原始UNet的参数量在28M左右(上采样带转置卷积的UNet参数量在31M左右),而如果把channel数成倍缩小,模型可以更小。缩小两倍后,UNet参数量在7.75M。缩小四倍,可以把模型参数量缩小至2M以内,非常轻量。个人尝试过使用Deeplab v3+和DRN等自然图像语义分割的SOTA网络在自己的项目上,发现效果和UNet差不多,但是参数量会大很多。
3.多模态。相比自然影像,医疗影像比较有趣和不同的一点是,医疗影像是具有多种模态的。以ISLES脑梗竞赛为例,其官方提供了CBF,MTT,CBV,TMAX,CTP等多种模态的数据。

这就需要我们更好的设计网络去提取不同模态的特征feature。这里提供两篇论文供大家参考。
Joint Sequence Learning and Cross-Modality Convolution for 3D Biomedical Segmentation(CVPR 2017) ,
Dense Multi-path U-Net for Ischemic Stroke Lesion Segmentation in Multiple Image Modalities.
4.可解释性重要。由于医疗影像最终是辅助医生的临床诊断,所以网络告诉医生一个3D的CT有没有病是远远不够的,医生还要进一步的想知道,病灶在哪一层,在哪一层的哪个位置,分割出来了吗,能求体积嘛?同时对于网络给出的分类和分割等结果,医生还想知道为什么,所以一些神经网络可解释性的trick就有用处了,比较常用的就是画activation map。看网络的哪些区域被激活了,如下图。

这里推荐两篇工作:
老师的Learning Deep Features for Discriminative Localization(CVPR2016)和其实验室同学的 Deep Learning for Identifying Metastatic Breast Cancer(上图的出处)BTW:没有偏题的意思,只是觉得医疗影像的特点和本问题息息相关,就一起总结了。
最后提一个问题,引发关注医疗影像的同学们思考和讨论?
前面有提到,UNet成为大多做医疗影像语义分割任务的baseline,也启发了大量研究者去思考U型语义分割网络。 那UNet还有什么不足呢?欢迎大家关注我的项目UNet-family,寻找答案。
ShawnBIT/UNet-family同时也推荐,UNet++作者
的分享:研习Unet。 周纵苇:研习U-Net
好的,在高铁上完成了这个一直想回答的问题,开心~

