本文将介绍如何使用party包,rpart包及randomForest包来建立预测模型。
首先,我们将使用party包来建立决策树,并用决策树用于分类。其次,利用randomForest包来训练随机森林模型。
最后学习使用rpart包来建立决策树。
所使用的数据集为R中自带的iris数据集

使用party包来建立决策树
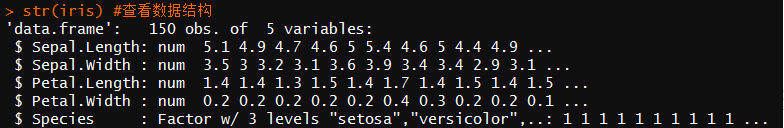
- 首先查看数据集结构
- 划分训练集和测试集
1 #划分训练集和测试集 2 set.seed(1234) 3 nrow(iris) #共150行 4 ind <- sample(x = 1:2,size = nrow(iris),replace = T,prob = c(0.7,0.3)) #设随机数1或者2 5 train_data <- iris[ind==1,] 6 test_data <- iris[ind==2,]
- 建立决策树
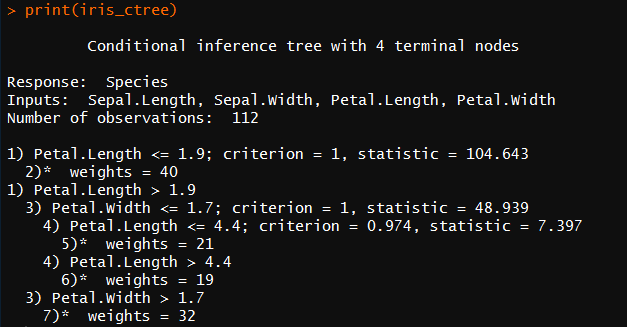
1 #建立决策树 2 library(party) #加载library包 3 iris_ctree <- ctree(formula = Species~.,data = train_data) #建立决策树,Species(种类为目标变量),其他为输入变量 4 5 table(predict(iris_ctree),train_data$Species) #建立频数表检查预测结果

可以看到,预测结果中,versicolor有一个预测错误,virginica有三个预测错误,预测结果可以接受。
- 输出规则和绘制图表
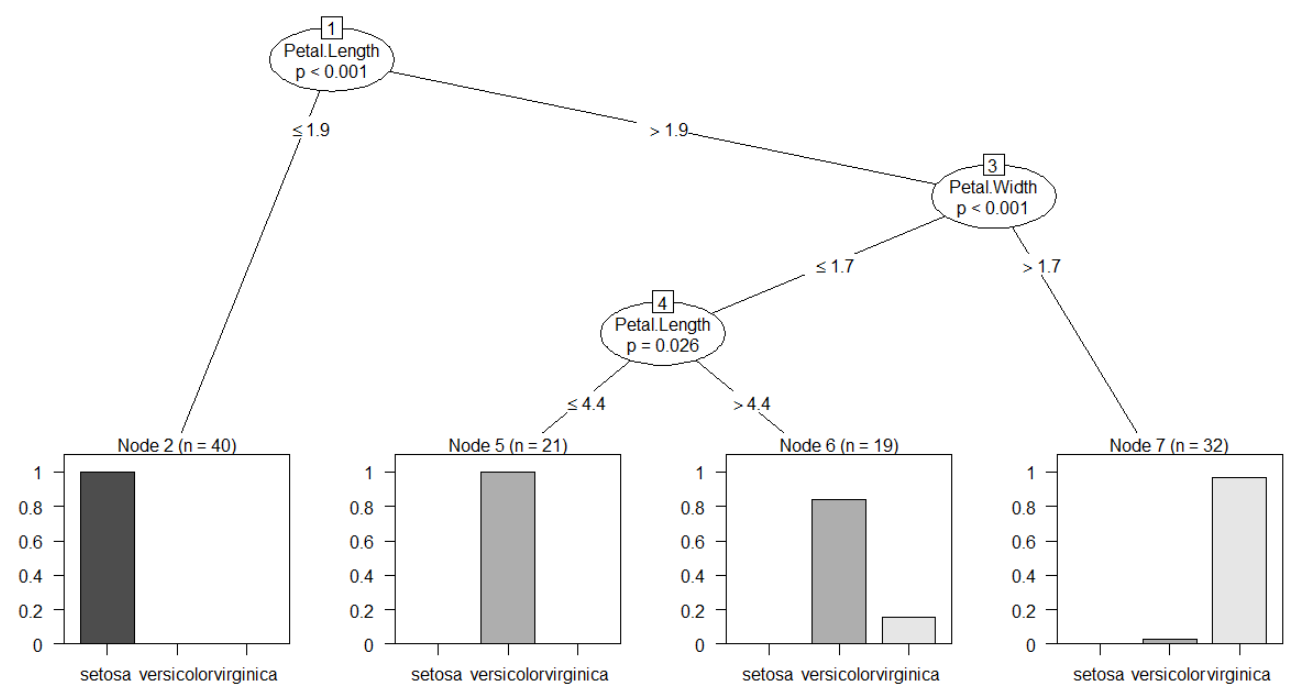
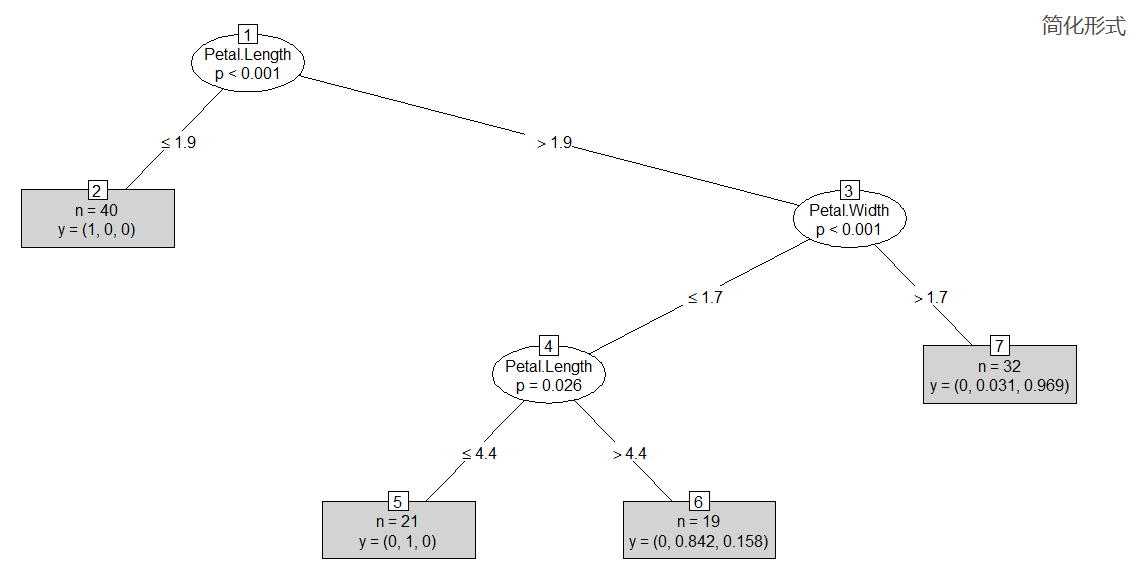
print(iris_ctree) plot(iris_ctree) #绘制决策树 plot(iris_ctree,type='simple') #简化形式
- 使用测试集对用训练集构建的决策树进行测试
iris_test <- predict(iris_ctree,newdata=test_data) #以iris_ctree为模型,代入需要预测的数据test_data,即学习集,进行预测 table(iris_test,test_data$Species) #输出列联表查看预测结果

仅有virginica中有两个错误归类为versicolor,预测模型准确率达到94.7%。
-
使用randomForest包训练随机森林
1 #使用randomForest包训练随机森林 2 library(randomForest) 3 rf <- randomForest(Species~. ,data = train_data,ntree=10,proximity=T)#产生随机森林,ntree:决策树数量 4 table(predict(rf),train_data$Species) #查看频数表
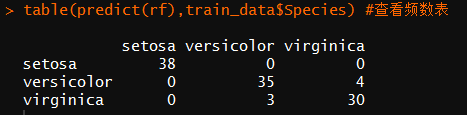
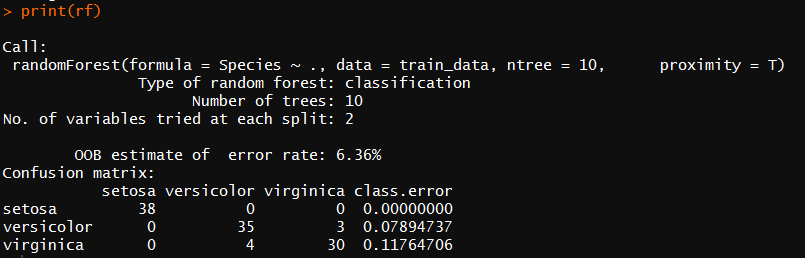
print(rf) #查看规则
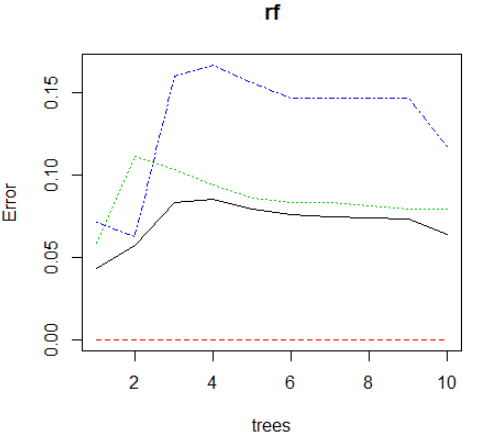
plot(rf)#根据随机森林生成的不同的树绘制误差率
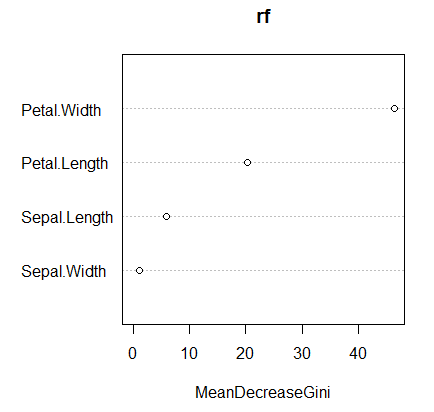
importance(rf) #查看每个变量的重要性

varImpPlot(rf) #通过图形查看每个变量的重要性
-
使用测试集测试模型
#使用测试集测试训练模型 iris_ran_PRED <-predict(object = rf,newdata = test_data) table(iris_ran_PRED,test_data$Species)

-
使用rpart包构建数据集
-
载入数据集
调用TH.data包中的Bodyfat数据集

-
划分数据集
1 #划分数据集 2 set.seed(1234) 3 ind <- sample(x = 1:2,size=nrow(bodyfat),replace = T,prob = c(0.7,0.3)) 4 train_data <- bodyfat[ind==1,] 5 test_data <- bodyfat[ind==2,]
-
构建决策树
1 #构建决策树 2 library(rpart) #加载包使用rpart,prune等函数 3 #查看体脂和年龄、腰围、臀围、肘宽、膝宽的关系 4 bodyfat_rpart <- rpart(formula = DEXfat~age+waistcirc+hipcirc+elbowbreadth+kneebreadth, 5 data = train_data, 6 control=rpart.control(minsplit = 10)) 7 print(bodyfat_rpart) #输出规则 8 plot(bodyfat_rpart) #绘制决策树 9 text(bodyfat_rpart,use.n = T) #添加标签
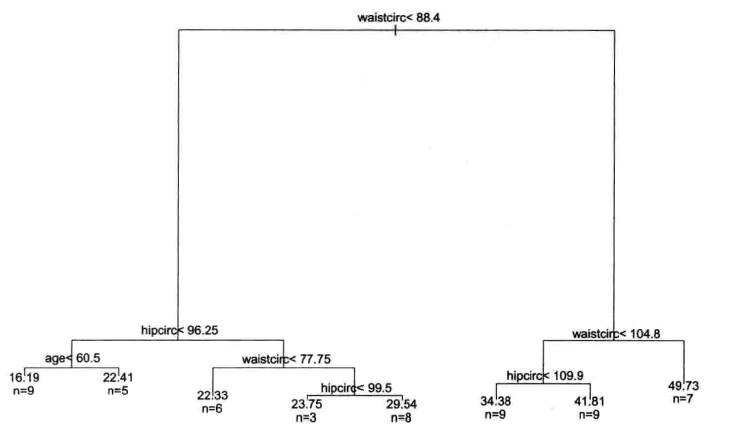
-
修剪决策树
1 #修剪决策树 2 opt <- which.min(bodyfat_rpart$cptable[,'xerror']) #选择具有最小误差的决策树,第七行的xerror为最小 3 cp <- bodyfat_rpart$cptablep[opt,'CP'] #选择第七行的'CP'值 4 bodyfat_prune <- prune(bodyfat_rpart,cp=cp) #修剪树 5 print(bodyfat_prune) 6 plot(bodyfat_prune) #绘制修剪后的决策树 7 text(bodyfat_prune,use.n = T) #添加标签 8 DEXfat_pred <- predict(bodyfat_prune,newdata = test_data) #预测 9 xlim <- range(bodyfat$DEXfat) #X坐标的区间为体脂的区间 10 plot(DEXfat_pred~DEXfat,data=test_data,xlab='Observed',ylab='Predicted',ylim=xlim,xlim=xlim) #绘制图形查看观测值和预测值的差异 11 abline(a=0,b=1) #加入对角线进行对比,y=a+bx

上图具有最小预测误差的决策树。
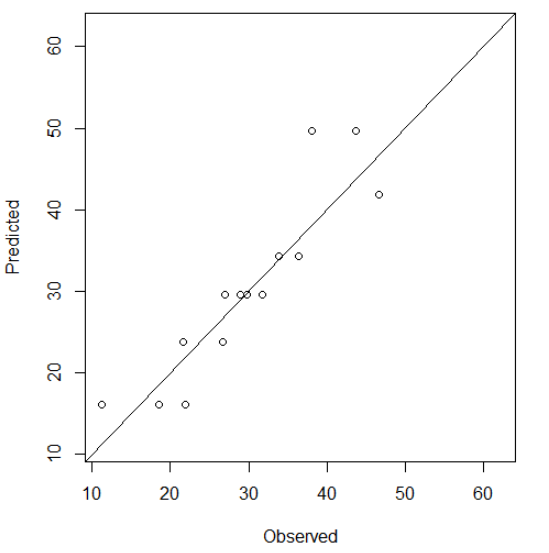
可观察到测试集中,观测值和预测值的差异,从而判断是否为一个好的预测模型。可看到大部分点都在对角线上或附近,
约5个点差异较大,该决策树模型的准确率约为1-(5/15)=66.7%
: 通过这个接口,可以在R中使用Weka的所有算法