传统化疗是对抗癌症的常见方法,但它会攻击全身,造成不必要的副作用,如脱发,恶心和疲劳。
靶向治疗选择性地杀死癌细胞而不影响健康组织。 靶向药物开发将成为治疗癌症的重要手段。
肿瘤的生物信息学数据库对肿瘤基础研究的发展、临床治疗水平提供具有重要作用。
1. 综合性肿瘤数据库
TCGA
The Cancer Genome Atlas (TCGA)TCGA是由美国国立癌症研究所(NCI)和国家人类基因组研究所资助,关注与癌症的发生和发展相关的分子突变图谱。该数据库主要对样本进行外显子组和基因组测序分析,所提供的数据包括:基因组拷贝数变化、表观遗传、基因表达谱、miRNA等。
ICGC
InternationalCancer Genome Consortium (ICGC)ICGC的目标是获取包括胆道癌、膀胱癌、血癌等多达50种肿瘤及其亚型的基因组、转录组和表观遗传的全部信息。这些数据可促进癌症的机理和治疗研究。
COSMIC
Catalogue of Somatic Mutations inCancer (COSMIC)COSMIC是世界上最大最全面的有关肿瘤的体细胞突变以及其影响的资源库。它主要提供多种肿瘤细胞基因组中的CNA、甲基化、基因融合、SNP及基因表达等信息,这些突变信息是从科学文献中手工整理的。主页面分为项目、数据管理、工具、帮助、搜索框等几大块,简洁清晰。
cBioPortal
cBioPortal for Cancer Genomics(cBioPortal)cBioPortal是一个癌症基因组数据探索、可视化及分析平台,可用于多个癌症基因组学数据集的交互式探索。该数据库可提供CNA、基因突变信息。针对每个基因,它可给出多个信息,主要包括:基因的CNA信息、基因突变在样本中的分布、突变位点和频率、共表达基因以及生存曲线等。对于用户提供的基因列表,还可生成互作网络并提供已知的相互作用的药物。在发现肿瘤相关突变、分析基因的生物学功能以及药物选择等方面的研究中具有重要推进作用。
UCSC Cancer Genomics Browser
UCSC Cancer Genomics Browser UCSC Cancer Genomics Browser是一个可以对癌症基因组学和临床数据进行整合、可视化、分析的网络分析工具。它保存癌症基因组及临床数据并收集了样本的多种信息,包括基因表达水平、CNA、通路信息等。在UCSC的癌症基因组浏览器中,可实现不同样本以及癌症类型之间的比较,分析基因组变异与表型之间的相关性。
用户可以通过它浏览基因组的任何一部分,并且同时可以得到与该部分有关的基因组注释信息,如已知基因、预测基因、表达序列标签、mRNA、CpG岛,克隆组装间隙和重叠、染色体带型、小鼠同源性等。
canEvolve
canEvolve 存储的信息包括:基因、microRNA (miRNA)和蛋白质表达谱、多种癌症类型的拷贝数变化(CNAs)以及蛋白质-蛋白质相互作用信息。
CGAP
Cancer Genome Anatomy Project(CGAP) CGAP主要提供了cDNA克隆、文库、基因表达、SNP以及基因组变异等信息。CGAP收集的数据包括正常组织、前癌组织以及癌细胞的基因表达水平。
CGHub
Cancer Genomics Hub (CGHub) CGHub是美国国家癌症研究所(NCI)测序项目的在线存储库,其数据来源包括癌症基因组图谱(TCGA)、癌症细胞系百科全书(CCLE)和产生有效治疗(目标)项目的治疗应用研究(TARGET)3个国家癌症协会项目,数据来自25种不同类型的癌症。
CGWB
Cancer Genome Work Bench (CGWB) CGWB提供了一系列工具来挖掘、整合以及可视化TCGA等数据库中的基因组和临床数据,它是第一个将临床肿瘤突变谱与参考人类基因组整合在一起的计算平台。用户可快速地比较患者临床信息与基因组的变异及甲基化等。
2.肿瘤基因组数据库
肿瘤细胞的基因组中都存在着大量的变异,主要包括染色体结构的变异、CNA、基因融合以及SNP等。拷贝数改变(CNAs)在很大程度上有助于癌症发病机制和进展。
ArrayMap
ArrayMap ArrayMap提供预处理过的肿瘤基因组芯片数据以及CNA图谱。在ArrayMap数据库中,用户可搜索自己感兴趣的样本,并在此基础上分析感兴趣的基因或基因组片段上的CNA;用户还可以比较两个样本之间的CNA的差异arrayMap数据库为高分辨率致癌基因组CNA数据的meta分析和系统级数据集成提供了切入点。
BioMuta
BioMuta BioMuta数据库存储了癌症细胞中基因的非同义单核苷酸变异,这些突变会影响基因的正常功能。BioMuta中的数据来源于COSMIC、ClinVar、UniProtKB以及一些文献中。用户可搜索感兴趣的基因,获得该基因在癌细胞中的突变位点及其分布频率。
Cancer Hotspots
Cancer Hotspots数据库由Memorial Sloan Kettering癌症中心的Kravis分子肿瘤学中心维护,提供大规模癌症基因组学数据中发现的在统计学上有显著复发突变的信息。
目前,Cancer Hotspots里面包含有24592个肿瘤样品中鉴定的单残基和框内indel突变热点。用户还可按照gene、residue、type、variants等对其内容进行排列查看。
OncoKB
OncoKB是由Memorial Sloan Kettering癌症中心(MSK)维护的全面的精准肿瘤学知识库,包含来自FDA,NCCN或ASCO,ClinicalTrials.gov和科学文献的专业指导方针和建议,治疗策略,肿瘤专家或肿瘤协会共识,参考文献等信息。
OncoKB目前包含有关554种癌症基因特定改变的详细信息,还有1级(FDA批准)、2级(标准护理)的治疗信息,3级临床证据和生物学证据。
CanGEM
Cancer GEnome Mine (CanGEM) CanGEM是一个公共的数据库,用于存储定量微阵列数据和临床肿瘤样本数据。它主要利用ArrayCGH芯片来发掘基因的拷贝数变异。
CGP
Cancer Genome Project (CGP) CGP提供了肿瘤中的CNA及基因型信息,该数据库的主要目标是利用人类基因组序列和高通量的突变检测技术识别体细胞突变,进而发现人类肿瘤发生过程中重要的基因。该数据库还提供了一些识别突变、CNA的软件,如BioView、GRAFT等。
3. 肿瘤DNA甲基化数据库
DNA甲基化修饰是表观遗传学的一种重要形式,它调节基因的转录水平,对维持细胞的正常功能起着重要作用。DNA甲基化模式的改变可能导致癌症。
DiseaseMeth
DiseaseMeth DiseaseMeth是一个人类疾病甲基化数据库,其重点是对各种疾病的DNA甲基化数据集进行有效的存储和统计分析。它涉及的疾病包括癌症、神经发育和退行性疾病、自身免疫疾病等。在DiseaseMeth中可以比较疾病与疾病之间、基因与基因之间以及疾病与基因之间的甲基化关系。
MENT
MENT MENT数据库收集和整合了来自GeneExpression Omnibus(GEO)和TCGA的DNA甲基化、基因表达水平数据,同时将DNA甲基化和基因表达水平关联起来。
MethHC
MethHC MethHC是一个集成数据库,包含大量DNA甲基化数据和mRNA/microRNA在人类癌症中的表达谱。这些数据可以帮助研究人员确定表观遗传模式。
MethyCancer
MethyCancer 该数据库拥有来自公共资源的高度整合的DNA甲基化数据、癌症相关基因、突变和癌症信息,以及我们大规模测序得到的CpGIsland (CGI)克隆。MethyCancer可用于研究DNA甲基化、基因表达与癌症的相互作用。
MethDB
MethDB 是较早的DNA甲基化数据库,主要集中于环境因子对甲基化的影响;
NGSmethDB
NGSmethDB 基于高通量测序数据,最近更新中还包含了SNP信息,以便后续分析。
4. 肿瘤转录组数据库
肿瘤细胞具有较强的生长和繁殖能力,生命活动旺盛,因此与正常细胞相比,基因的转录水平和模式也存在较大的差异。
Oncomine
Oncomine Oncomine是大型的肿瘤基因芯片数据库,致力于收集、标准化并分析肿瘤样本的基因表达谱芯片数据。
它可提供基因在肿瘤样本和正常样本间、肿瘤样本和肿瘤样本间、正常样本和正常样本间的差异表达、基因表达谱、预测共表达基因等信息,并可根据肿瘤分期、分级、组织类型等临床信息进行分类。
GEO
Gene Expression Omnibus (GEO) GEO是由美国国家生物技术信息中心(NCBI)建立的,其最初的目标是作为一个公共存储库,存储主要由微阵列技术生成的高通量基因表达数据。此外,该数据库还包括比较基因组分析、描述基因组蛋白相互作用的染色质免疫沉淀分析、非编码RNA分析、SNP基因分型和基因组甲基化状态分析。
ArrayExpress
ArrayExpress ArrayExpress是欧洲生物信息协会(EMBL-EBI)下属的功能基因组数据库,收集整理基于芯片和测序的基因组学实验的数据,以支持可重复的研究。ArrayExpress是基于微阵列和高通量测序的功能基因组实验的主要知识库之一,所有数据都以MAGE-TAB格式提供。
在帮助页面有详细的在线教程,供用户学习如何搜索、提交数据。
ChiTaRS
ChiTaRS ChiTaRS数据库包含嵌合转录本和RNA-Seq数据。ChiTaRS嵌合转录本和RNA-Seq数据数据库是由GenBank、ChimerDB、dbCRID、TICdb和其他用于人类、小鼠和苍蝇的数据库的表达序列标记(ESTs)和mRNA识别的嵌合转录本集合。
miRCancer
miRCancer miRCancer基于从文献中提取的结果,提供了较为全面的miRNA集合以及它们在多种肿瘤中的表达情况。所有miRNA的癌变关联都是在自动提取后手动确认的。
OncomiRDB
OncomiRDB OncomiRDB主要收集和注释通过实验验证的对癌症具有促进或抑制作用的miRNA信息。该数据库的所有数据是通过人工收集和整理。
SomamiR
SomamiR SomamiR数据库集成了多种类型的数据,用于研究体细胞和种系突变对癌症中miRNA功能的影响。该数据库主要收集miRNA及其靶序列上的突变。另外,数据库还提供了存在miRNA靶序列体细胞突变与肿瘤相关的基因及其参与的通路。
5. 肿瘤蛋白组数据库
蛋白是生命活动的主要承担者,蛋白结构变异、蛋白修饰的改变以及蛋白含量的变化等导致细胞的生长和代谢变化是肿瘤发生的重要因素。
CPTAC
ClinicalProteomic Tumor Analysis Consortium (CPTAC) CPTAC整合了基因组和蛋白组的数据,旨在识别和描述肿瘤组织和正常组织中的全部蛋白,发掘可作为肿瘤生物标记的候选蛋白。
Cancer3D
Cancer3D Cancer3D数据库整合了来自TCGA和CCLE的体细胞错义突变信息,在蛋白结构水平上分析其对蛋白功能的影响。该数据库通过e-Driver和e-Drug两种算法,帮助用户分析突变的分布模式及其与药物活性变化的关系。
CancerPPD
CancerPPD CancerPPD是一个抗癌肽(ACPs)和抗癌蛋白的储存库,在设计基于肽的抗癌疗法中非常有用。在CancerPPD中,针对每个条目,都有其详细的注释信息,如肽的来源、肽的性质、抗癌活性、N-和C-末端修饰、构象等。除了天然肽,CancerPPD还含有非天然的、经过化学修饰的残基肽和D-氨基酸。CancerPPD还整合了一些基于web的工具,包括关键字搜索、数据浏览、序列和结构相似性搜索。
CanProVar
Cancer Proteome Variation Database(CanProVar) CanProVar数据库整合了来自各种公共资源的蛋白质序列变异信息,重点是癌症相关的变异,CanProVar中的数据主要来源于TCGA、COSMIC、OMIM、HPI等数据库以及一些文献研究。在该数据库中,用户可在网站中搜索特定蛋白或者某种肿瘤,获取蛋白的突变情况,在结果页面会给出蛋白的基本信息、GO注释以及相关的研究文献。
dbDEPC
DbDEPC DbDEPC是一个专门收集肿瘤样本中出现的差异表达蛋白的数据库。在该数据库中,你可以了解你所感兴趣的蛋白质是否在某些癌症中发生了变化。
6.肿瘤相关基因数据库
DriverDB
DriverDB DriverDB收集了来自TCGA、ICGC、TARGET等数据库的大量exome-seq数据,并根据不同方面提供突变信息的可视化。这些可视化结果将有助于用户快速了解驱动基因之间的关系。
NCG
Network of Cancer Genes (NCG) 癌症基因网络(NCG)致力于收集关于人工筛选的已知和候选癌症基因的信息。针对每个基因,用户可获得与该基因相关的功能和疾病注释信息、突变信息、表达谱、miRNA及蛋白互作关系等,还可以可视化miRNA调控关系和蛋白互作网络。
TP53MUL TLoad
TP53MULTLoad TP53MULTLoad是一个人工收集的有关TP53突变和突变体资源中心,包含了UMDTP53数据库以及与TP53有关的信息。它既可以作为一个容易操作的平面文件,也可以作为一个新的多平台分析软件,用于分析TP53突变的各个方面。
7. 肿瘤与药物数据库
耐药性是肿瘤治疗的一大障碍,药物靶点突变是产生获得性耐药的主要原因之一。对这些药物靶点突变的充分了解将有助于设计有效的个性化治疗。
CancerDR
CancerDR CancerDR是一种针对癌症治疗的个性化药物的尝试。CancerDR收集了148种抗癌药物以及它们在952种细胞系中的药理状况。
CancerResource
CancerResource CancerResource通过文献挖掘以及整合多种数据源的方式收集并发现了大量化合物及其靶点的信息。通过CancerResource数据库,你可以得到包含化合物与靶标的详细信息、表达图谱及相关数据来源链接等。
canSAR
canSAR canSAR整合ArrayExpress、UniProt、COSMIC等11种数据源的数据。它是一个支持癌症转化研究和药物发现的公共癌症综合知识库。该数据库包含了包括生物学、药理学、化学、结构生物学和蛋白质相互作用网络等多种类型的数据。
GDSC
Genomics of Drug Sensitivity inCancer (GDSC) GDSC是关于癌症细胞药物敏感性和药物反应分子标记的数据库,GDSC提供了一个独特的资源,结合了大的药物敏感性和基因组数据集,以促进发现新的治疗生物标志物的癌症治疗。该数据库中的癌基因组突变信息包括癌基因点突变、基因扩增与丢失、组织类型以及表达谱等
Platinum
Platinum Platinum是一个广泛收集耐药性信息的数据库,是为了研究和理解错义突变对配体与蛋白质组相互作用的影响而开发的。该数据库包含超过1000种蛋白配体复合物的三维结构突变,以及这些突变对其亲和力的影响。Platinum数据库将蛋白质结构突变与配体的亲和力关联起来,有助于研究由突变引起的疾病耐药性。
8. 其他相关数据库
1000 Genomes
1000 Genomes Project(缩写为1KGP)在2008年到2015年间进行,1000Genome Project 的目标是在群体中找到频率至少为1%的遗传变异,为人类遗传变异的研究提供了一个综合的资源。最终数据集包含来自26个群体的2504个个体的数据。所有样本都有外显子测序数据。比其他同类数据库优越的是,1000 Genomes Project里有24个个体进行了全基因组测序。现在1000 Genomes Project的数据可以通过IGSR(The International Genome Sample Resource)的数据门户网站访问,现已更新匹配GRCh38参考基因组的数据。在首页可以进行检索和分析,进入FTP站点可以进行数据下载。
ESP
ESP全称是NHLBIExome Sequencing Project(国家心肺血液研究所外显子组测序项目), 是由多个大学和研究结构合作开展的一个大型的外显子测序项目,主要目的是通过对不同人群进行SNP分型,来辅助心脏,肺,血液相关疾病的研究。
ExAC
ExAC全名ExomeAggregation Consortium(外显子组整合数据库),由哈佛-麻省理工Broad研究所的科学家完成,整合了17个人类基因组项目,60706个个体的外显子测序数据。整合数据的过程中去除了严重儿科疾病的个体和TCGA中的肿瘤样本,因此适合作为研究严重疾病的等位基因频率的参考组。
ClinVar
ClinVar(Clinical Significance for Variants Relative to Phenotypes, NCBI)是NCBI主办的与疾病相关的人类基因组变异数据库。它的强大在于整合了dbSNP、dbVar、Pubmed、OMIM等多个数据库在遗传变异和临床表型方面的数据信息,形成一个标准的、可信的遗传变异-临床相关的数据库
HGMD
HGMD The Human Gene Mutation Database存储了人类疾病相关的突变信息,由于其全面性,在突变研究领域,有广泛应用。有免费和收费版本。
OMIM
omim全称为“Online Mendelian Inheritance in Man”,它通过对新的病症分类并命名、收录表型和相关病因基因的关系来收录人类孟德尔疾病信息。
omim主要关注在疾病与基因层面上,而clinvar核心是和临床相关的基因突变。
CIViC
CIViC即Clinical Interpretations of Variants in Cancer,主要是解读用,里面有癌症相关的变异位点variant,基因gene还有临床信息(ClinicalEvidence)、用药信息等。
9. 总结
下表是一篇综述统计的肿瘤生物信息数据库:
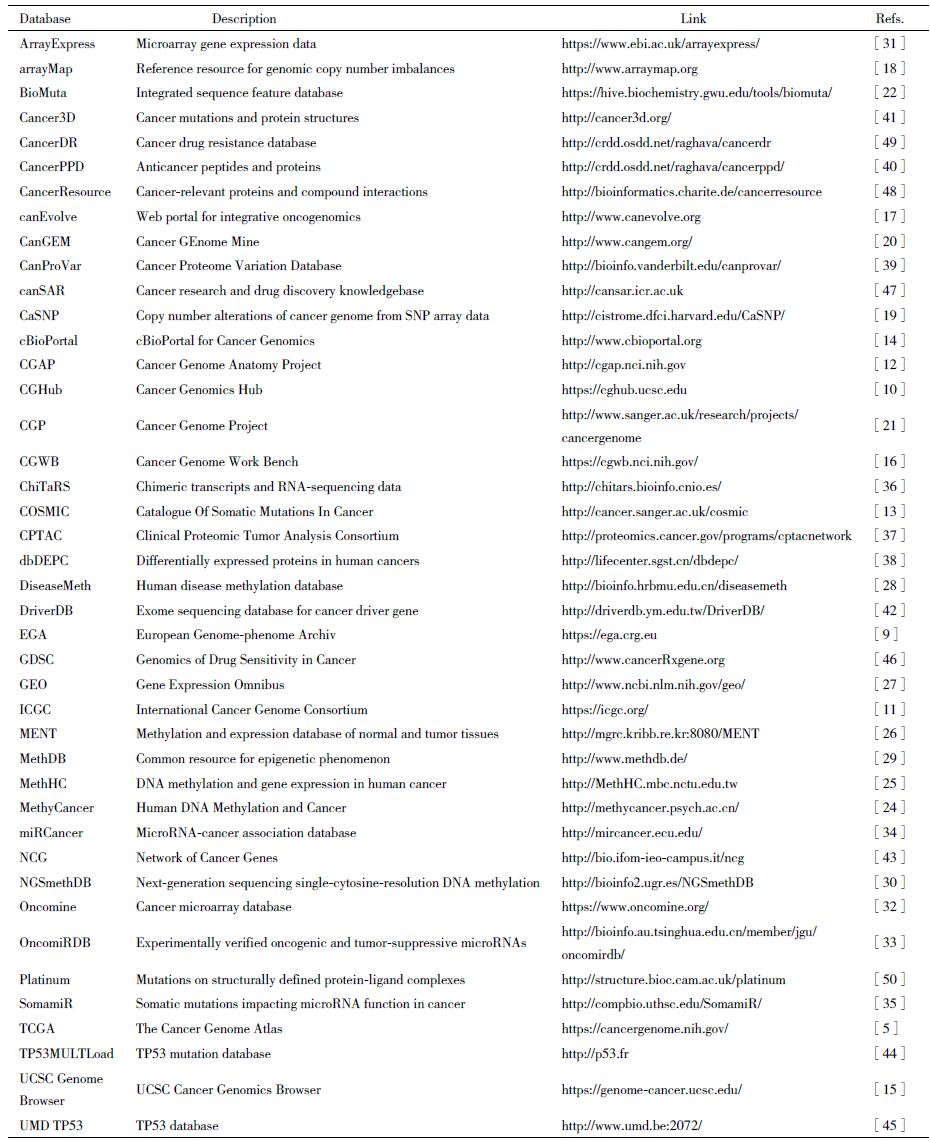
以上的分类并不是很严谨,不同类型的数据库会有交叉。此外,还有一些特有癌症的数据库,如乳腺癌, BCI (Breast Cancer Information Core, NIH); BRCA Share (UMD-BRCA1/2 Mutations Database, BRCA GGC Consortium) 等等。
分类只是为了更清晰,有一个整体印象,不必每一个都熟悉。实际上工作中最常用到的数据库就以下几个,有需求再去查:
- TCGA
- Oncomine
- ICGC
- cBioPortal
- COSMIC
- GEO
- 1KGP
Ref:
肿瘤数据库,可不只有TCGA、GEO!
资源|肿瘤数据库汇总
突变相关数据库教程
1000genomies ESP ExAC比较
杨健, 蔡浩洋. 肿瘤生物信息学数据库[J]. 生物技术通报, 2015, 31(9): 89-101