自然语言处理在医学领域中的应用
1.总述
近年来医疗数据挖掘发展迅速,然而目前医疗数据结构化处于起步阶段,更多的医疗数据仍然以自然语言文本形式出现。自然人的学习能力有限,因此学者们尝试通过自然语言处理(Natural Language Processing,NLP)辅助完成汇总医学领域知识的过程,将知识提炼出来,提取其中有用的诊疗信息,最终形成知识本体或者知识网络,从而为后续的各种文本挖掘任务提供标准和便利。
2.具体应用
2.1 文本挖掘
1)研究背景:生物医学文本挖掘可以帮助人们从爆炸式增长的生物医学自然语言文本数据中抽取出特定的事实信息( 主要是生物实体如基因、蛋白质、药物、疾病之间的关系) ,对整个生物知识网络的建立、生物体关系的预测、新药的研制等均具有重要的意义。
2)典型应用及应用方法
2.2.1命名实体识别
1)研究背景
生物命名实体识别,就是从生物医学文本中识别出指定类型的名称,比如基因、蛋白质、核糖核酸、脱氧核糖核酸、疾病、细胞、药物的名称等[1]。由于生物医学文献的规模庞大,各种专有名词不断涌现,一个专有名词往往有很多同义词,而且普遍存在大量的缩写词,人工识别费时费力,因此如何对命名实体进行识别就变得尤为重要。命名实体识别是文本挖掘系统中的一个重要的基础步骤,命名实体识别的准确程度是其他文本挖掘技术如信息提取或文本分类等的先决条件。
2)典型应用及应用方法
目前,使用比较多的生物命名实体识别的研究方法主要有以下几种:基于启发式规则的方法[2]、词典匹配的方法[3]以及机器学习的方法,如支持向量机(SVM)[4]、最大熵[5]、条件随机场 (CRF)[6]以及隐马尔科夫(HMM)[7]等。
(1)基于启发式规则的方法
Fukuda等人[2] 最早利用基于规则的系统判定文档中的蛋白质名称;Tsuruoka等人[8]采用启发式规则以最小化相关术语的歧义性和变化性,实现了术语名称的标准化进而提高了查找字典的效率。
优点:利用启发式信息产生识别命名实体的规则可以灵活地定义和扩展
缺点:规则对领域知识的依赖性很强,修改它们需要 该领域专家参与并花费大量时间。 另外,由于命名实体类型多样,且新类型的命名实体还在不断涌现,这使得人们很难建立一套一致的规则。
目前,基于规则的方法一般被整合到基于机器学习的方法的后期处理过程中[5]。
(2)基于字典的方法
最早采用的方法是基于字典的方法,1998年,Proux等人[9]第一次应用英语词典来识别基因和蛋白质。
优点:简单且实用。
缺点:新的命名实体不断出现,并且很多命名实体的长度较长甚至存在变体,难以建立一个完整的的生物医学命名实体字典。
因此,基于字典的方法通常以字典特征的形式被整合到基于机器学习的方法中[10]。
(3)基于机器学习的方法
基于机器学习的方法是目前主流的方法,它们利用统计方法从大量数据中估算相关参数和特征进而建立识别模型。
优点:客观、移植性好。
缺点:需要大量的训练数据且训练过程相当耗时。
命名实体识别可以看做是词的分类问题,因此可以采用基于分类的方法如贝叶斯模型和支持向量机[4]等;同时,它也可以看做是序列分析问题(每个词语作为序列中的一个词被打上标签),因此可采用条件随机域[6]、隐马尔可夫模型[7]等基于马尔可夫的模型。基于机器学习的方法包括特征选择、分类方法和后期处理等几个步骤。
2.2.2 关系抽取
1)研究背景
关系抽取( Relationship extraction,RE) 的目标是检测一对特定类型的实体之间有无预先假设的关系[39]。生物医学文本挖掘抽取的就是基因、蛋白质、药物、疾病、治疗之间的关系。
2)典型应用及应用方法
主要有基于模版的方式( 手动、自动) 、基于统计的方式和基于自然语言处理的方式。基于自然语言的方法就是把自然语言分解为可从中提取出关系的结构[11]。Friedman[12]等人通过提出了GENIES系统,它从生物学文献中提取和构建关于细胞途径的信息。
2.2.3 文本分类
1)研究背景
文本分类( Text classification) 就是将文本自动归 入预先定义好的主题类别中,是有监督的机器学习 方法,主 要应用于自动索引、文本过滤、词义消歧 ( WSD) 和 Web 文档分类等。
2)典型应用及应用方法
目前,文本分类的方法有很多,典型且效果较好 的有朴素贝叶斯分类法( Na Bayes) 、K 最近邻( K - NN) 、支持向量机( SVM) 、决策树等,还有基于关联的分类( CBA) 及基于关联规则的分类( ARC) 。Eskin E[13]使用 SVM 算法和基因序列 kernel 预测蛋白质在细胞质中的位置,达到了 87 % 的查准率和 71% 的 查全率。
2.2.4 文本聚类
1)研究背景
文本聚类( Text clustering) 是根据文本数据的特征将一组对象集合按照相似性归纳为不同类的过 程,与文本分类的区别是分类的对象有类别标记。
2)典型应用及应用方法
常见的聚类算法可归纳为平面划分法( 如 K - 均值算法、K - 中心点算法) ,层次聚类法( 可分为凝 聚层 次 聚 类 和 分 割 聚 类) ,基 于 密 度 的 方 法 ( 如 DBSCAN 算法) ,基于网格的方法( 如 STING 算法) ,基于 模 型 的 方 法。
Groth P 等[14]根据显型的描述,利用文本聚类 将基因聚类成簇,利用这些簇预测基因功能,采用客观标准选择一个子类团,从生物过程次本体中预测GO-术语注释,得到了 72. 6% 的查准率和 16. 7% 的 查全率。
2.2.5 共现分析
1)研究背景
共现( Co-occurrence) 分析主要是对隐性知识的挖掘,在生物医学领域主要用于诸如 DNA 序列的数据分析、基因功能相似聚类、基因和蛋白质的功能信息提取、提高远程同源性搜索、基因与确定疾病关系预测等[15]。如果在大规模语料( 训练语料) 中,两个词经常共同出现( 共现) 在同 一窗口单元( 如一定词语间隔、一句话、一篇文档等)中,则认为这两个词在语义上是相互关联的。而且, 共现的频率越高,其相互间的关联越紧密。
2)典型应用及应用方法
基于共现关系的假定,通过对训练语料的统计,计算得到词与词之间的互信息( Mutual information) ,就可以对词与词之间的相关性进行量化比较,获得对文本词汇 语义级别的关联认识。如Pub-Gene系统使用共现方法建立了一个包含基因和基因交互关系的数据库[16],实验结果达到了60%的精确率和51%的召回率。当仅考虑5篇或5篇以上文章中的基因对关系时,精确率上升到72%。[]16]
2.2 决策支持系统[17]
1)研究背景
在医学临床实践中,对于医务人员来说,作为一个理智、情感共存的个体,在医学实践中难免会犯错,这导致了医患双方关系的紧张、甚至生命健康的负面影响。为了降低出错的概率以及提高工作效率,临床决策支持系统应运而生,它可以对医务人员进行诊疗方面的指导。
2)典型应用及应用方法
医疗决策支持系统的建立主要分为以下三个步骤:
2.1 知识库的建立
词库是自然语言处理的基础,首先应建立词库。使用医学专业词汇、频率极高的谓词、量词等词汇、医疗文书词汇的常用组合及常用语句等,加上基本的语法库,形成用于医学语言处理的知识库。
另外,作为临床支持系统,还需要建立作为比较条件的知识库,使患者的各种诊疗要素形成一定倾向性的结果输出。
2.2 语言处理
按照中文自然语言处理的一般步骤,进行分句、分词、语义分析、形成文本摘要。
2.2.1 分句
分为基本单句的分割,和句群的分割。分句主要以基本的标点符号作为分隔符对语言进行计算机子句分割,完成分句处理。中文主要以句号、问号、省略号等为句群结束符,而医疗文书基本上都是陈述句,故多以句号为句群结束符。
2.2.2 分词
目前主流的分词算法主要有三种,分别为基于字符串匹配的分词算法、基于理解的分词算法和基于统计的分词算法。从词库中词条或习惯搭配短语的最大长度开始,逐渐缩短,对基本分句进行匹配词库中的词条。最后把医疗文书分割为一个个词汇或短语。
2.2.3 语义分析、文本摘要
根据汉语基本语法,对词汇进行重组,剔除意义不大的部分,形成摘要。以上述病程记录进行分句、分词为例:
第一步、分句:句群:今日查房,患者诉头昏乏力减轻,腹泻停止,进软食。 分句:今日查房 患者诉头昏乏力减轻 腹泻停止 进软食
第二步、 分词: 今日 \ 查房 患者 \ 诉\ 头昏\ 乏力\ 减轻 腹泻\停止 进\ 软食
2.3 临床决策支持系统
以临床诊疗指南、操作规范为参考,在对医疗文书进行语言处理后进行推理、分析,找出其中存在的问题。分析模型是其中的关键。如图1所示,以上述病程记录为例:依次输入词汇、短语。
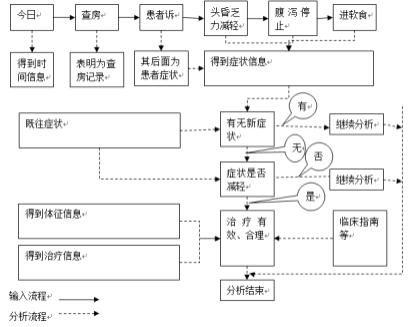
图1 决策支持系统模型
在分析模型中,比照的是临床诊疗指南、操作规范,所以在建立此知识库时,所用的词汇、短语应该与语言处理所用的知识库相对应,否则会增加建立分析模型的难度和复杂性。
2.3 信息提取
1)研究背景
信息抽取(Information Extraction,IE)是指从文本中抽取指定的一类事实信息,形成结构化的数据储存在数据库中,以供用户对信息的查询或进一步分析利用的过程。[18]如一位生物医学科学家要从海量的生物医学文献中寻求关于某种疾病的新的治疗方案,借助于信息抽取系统抽取出的蛋白质、基因或药物等的交互关系信息,就有可能从中发现有价值的治疗线索或方法。
2)典型应用及应用方法
信息抽取技术在电子病历中的应用
由哥伦比亚大学的Carol Friedman等人设计的MEDLEE系统也是一个很成功的医学信息抽取系统,作为临床信息系统(CIS)的一个独立模块在纽约长老会医院使用,它将文本形式的病历报告转换成编码数据以促进乳腺癌研究,有利于病人看护质量的提高[19]。息抽取技术在电子病历中的成功,将克服临床决策支持、临床路径管理等前沿医疗信息发展所面临的诸多瓶颈问题,提升我国医疗信息技术产业的核心竞争力。
信息抽取技术在医学文献中的应用
国内对生物医学文献信息抽取研究相对较多,极大地促进了生物医学的现代化进程,如从中药复方的临床文献进行复方名称的抽取[20];利用信息抽取技术从Web形式的中医药文献资料中抽取结构化中医临床诊疗信息的中医临床诊疗垂直搜索系统TCMVSE[21]。
信息抽取技术在生物医学网络资源中的应用
针对网络上分布散乱的生物医学资源,可以用基于HTML结构的信息抽取方法实现对生物医学资源的抽取,将其转换成结构化的数据存储到数据库中。具体过程见下图2。
![]()
图2 HTML文件转换成解析树示意图
北京中医药大学在1989年完成了“中医方剂信息智能分析支援系统”,收集了对40余万条方剂信息的解释,可产生800余万相关数据,并于1997年得到国家教育部博士点学科专项基金的支持,用Wed_db技术,将方剂数据库移植到Oracle7for UNIX平台,在Internet网上实验性地实现了方剂数据库的查询和分析处理[22]。
2.4 自动问答系统
1)研究背景
随着大数据时代的到来,对于传统的信息检索来说,由于医学专业的特殊性,面对网络上质量参差不齐的医学信息,非医学专业人员在查找、理解及获取方面存在诸多困难和障碍。而基于自动问答的医学信息搜寻模式作为更智能的医学信息资源获取工具,不仅对海量数据资源的有效利用具有重大意义,而且在一定程度上可缓解医患之间信息不对称、提高医疗资源利用效率,同时能更好地体现“以病人为中心”服务理念的转变。
2)典型应用及应用方法
2.1 基于传统搜索技术的问答系统
基于传统搜索技术的问答系统,在问题分析中将问题的关键词和数据资源中的关键词进行匹配,进而获取可能相关的答案片段。典型的医学领域自动问答应用具体见表1。

表1 基于传统检索技术的自动问答系统相关研究
应用方法如下:
基于传统搜索技术的问答系统的核心技术包括三个主要组成模块:问题处理、信息检索和答案抽取。
2.1.1 问题处理
(1)问题类型识别[23]
主要有启发式算法(基于规则的算法)、基于机器学习的算法等。
(2)提取问题关键词
可根据词语的词性、tfidf值或对不同重要程度的词语赋予权重等方法筛选出关键词。
(3)问题关键词拓展[24]
主要有基于词典的方法、基于统计的方法和相关反馈的方法。
- 基于词典的方法可用Wordnet(用于英文问答系统)、Hownet(用于中文问答系统)或其他同义词词典来扩展关键词。
- 基于统计的方法需要大量的问题和预料来训练。每一类问题所对应的答案一般有某种共同的特性,如对于询问地点的问题,答案中经常会出现“在、位于、地处”等关键词。所以通过统计,我们找到这些词后就可以把它们加到问句中。
- 相关反馈的方法是用检索返回的相关文档对关键词进行扩展。
2.1.2 信息检索
问答系统中的信息检索模块利用问题处理模块输出的关键词以及其拓展来搜索相关的段落。
主要有基于统计的方法和基于语义的方法。
基于统计的方法主要根据用户查询与数据全集中数据的统计量来计算相关性。目前较流行的有:布尔模型、概率模型和向量空间模型。[25]
基于语义的方法是对用户查询和数据全集中的数据进行一定程度的语法语义分析,也就是在对用户查询和数据全集中的内容进行理解的基础上进行两者的相关计算。
2.1.3 答案抽取[26,27]
主要有根据命名实体、推理、上下文的方法。
2.2 基于语义技术的问答系统
基于语义技术的问答系统,对自然语言问题进行语义处理,实现从语义层面理解用户提出的问题。相关的应用研究如表2,但目前相关的应用研究较少。

表2 基于语义技术问答系统相关研究
应用方法如下:
基于语义技术的问答系统在基于传统搜索技术的问答系统的基础上,可在问题处理模块和答案抽取模块加入对句子的结构进行分析(即句法分析)的方法。
在问题处理模块里需要通过对问句结构进行分析,根据问句的结构确定问句的类型,同时抽取句子关键词。
在答案抽取阶段,可对答案的候选句子进行结构分析,进行句子相似度的计算,去除重复或相近的候选答案,最后根据问题类型抽取出答案实体。
2.5 医学影像的信息提取和分析
1)研究背景
医学影像报告是电子健康病历 (electronic health record,EHR)中包含大量数字信息的重要组成部分。医学影像中使用NLP的总体目标是挖掘诊断报告中结构化信息,并将其应用于临床诊治过程。
2)典型应用及应用方法
根据信息提取的对象和目的不同,NLP可用于患者个体信息分析、患者群体信息分析和医学影像流程信息分析等。
1.患者个体影像诊断信息提取和分析,对患者个体疾病处理提供帮助
(1)提示“危急发现(critical findings)”:NLP检出影像报告中描述的、可能导致严重后果的影像征象,提醒处理该患者的医师注意[28]。目前NLP可提示的危急情况有阑尾炎、急性肺损伤、肺炎、血栓栓塞性疾病及各类潜在恶性病变等[29]。
(2)提示随访建议:NLP检出报告中应提示临床进行后续操作的内容,自动生成随访建议,提示后续检查或治疗[30]。
2.患者群体影像诊断信息提取和分析,构建患者队列,用于流行病学研究、行政管理等
(1)流行病学研究队列的构建:使用NLP可高效率地分析大数量、患者群体的影像报告,得到群体的特征性数据,从而提高流行病学研究效率,为循证影像医学研究提供帮助[31-35]。
3.医学影像流程信息的提取和分析,用于医学影像报告质量评价和改进
(1)报告质量评价和报告规范的建立:NLP可识别医学影像学的流程和质量指标,判断影像报告是否符合相关指南或诊断规则[36]。同时可用于评价报告的完整性和规范,是否给出正确的建议,是否及时进行危急情况的预警,报告信息是否用于疾病的诊断等方面[37-39]。
(2)影像检查全流程的改进:NLP可对各类影像的综合信息进行分析,将报告中的检查结果和建议等信息与全面的临床信息相互关联,如检查适应证、疾病种类、患者年龄、性别、申请 科室、申请医师及患者类型(住院或门诊)等[40]。这种大规模的数据分析在经过验证后,可得到预测模型,形成适合本地情况的临床决策支持系统(clinical decision support system,CDSS),应可应用到计算机医嘱系统(computerized physician order entry,CPOE)中去[41]。
[1]彭春艳, 张晖, 包玲玉,等.基于条件随机域的生物命名实体识别[J].计算机工程, 2009, 35(22):197-199.
[2]Fukuda K, Tamura A, Tsunoda T, et al. Toward information extraction: identifying protein names from biological papers.[C]// Pacific Symposium on Biocomputing. Pacific Symposium on Biocomputing. Pac Symp Biocomput, 1998:707-718.
[3]Tuason O, Chen L, Liu H, et al. Biological nomenclatures: a source of lexical knowledge and ambiguity.[J]. Pacific Symposium on Biocomputing Pacific Symposium on Biocomputing, 2004:238.
[4]Bakir G, Hofmann T, Schölkopf B, et al. Support Vector Machine Learning for Interdependent and Structured Output Spaces[C]// International Conference on Machine Learning. ACM, 2004:104.
[5]Lin Y F, Tsai T H, Chou W C, et al. A maximum entropy approach to biomedical named entity recognition[C]// International Conference on Data Mining in Bioinformatics. Springer-Verlag, 2004:56-61.
[6]Su J, Su J. Named entity recognition using an HMM-based chunk tagger[C]// Meeting on Association for Computational Linguistics. Association for Computational Linguistics, 2002:473-480.
[7]Li Y, Lin H, Yang Z. Incorporating rich background knowledge for gene named entity classification and recognition[J]. Bmc Bioinformatics, 2009, 10(1):1-15.
[8]Tsuruoka Y, Mcnaught J, Ananiadou S. Normalizing biomedical terms by minimizing ambiguity and variability.[J]. Bmc Bioinformatics, 2008, 9(3):1-10.
[9]Proux D, Rechenmann F, Julliard L, et al. Detecting Gene Symbols and Names in Biological Texts: A First Step toward Pertinent Information Extraction.[C]// CiteSeer, 1998:248-255.
[10]Mcdonald R, Pereira O. Identifying gene and protein mentions in text using conditional random fields[C]// BMC Bioinformatics. 2005:S6.
[11]Cohen AM,Hersh WR.Hersh.A Survey of Current Work in Biomedical Text Mining [J].Brief Bioinform ( S1467-5463 ),2005,6(1) : 57-71.
[12]Friedman C, Kra P, Yu H, et al. GENIES: a natural-language processing system for the extraction of molecular pathways from journal articles[J]. Bioinformatics, 2001, 17 Suppl 1(suppl_1):S74.
[13]Eskin E,Agichtein E. Combining text mining and sequence analysis to discover protein functional regions [C]. Altman RB,Dunker AK,Hunter L,et al. Pac Symp Biocomput,2004: 288 - 299.
[14]Groth P,Weiss B,Pohlenz HD,et al. Mining phenotypes for genefunction prediction [J / OL ]. BMC Bioinformatics (S 1471-2105),2008,9: 136.[2009-08-20]. http: / / www.biomedcentral. com / content / pdf /1471-2105-9-136. pdf.
[15]Erhardt RA,Schneider R,Blaschke C. Status of text - mining tech -niques applied to biomedical text[J].Drug Discov Today(S 1359-6446) ,2006,11( 7 /8) : 315-325.
[16]齐彬,吕婷.共现分析技术在生物医学信息文本数据挖掘中的应用[J].中华医学图书情报杂志,2009,18( 3) :41-43.
[17]刘坤尧, 杨渝沙. 基于自然语言处理的临床决策支持系统[J]. 医学信息, 2014(7).
[18]Pazienza M T. International Summer School on Information Extraction: A Multidisciplinary Approach to an Emerging Information Technology[C]// International Summer School on Information Extraction: A Multidisciplinary Approach to an Emerging Information Technology. Springer-Verlag, 1997:425–426.
[19]李莹 . 文本病历信息抽取方法研究 [D].杭州: 浙江大学,2009.
[20]周雪忠 . 文本挖掘在中医药 中的若干研究 [D] .杭州 :浙江大学 ,2009.
[21]庄 力. 中医临床诊疗垂直搜索系统研究 [D] .北京 :北京交通大学,2009.
[22]任廷革,刘晓峰,高剑波,等.“中医药基础数据库系统”介绍[J].中国中医药信息杂志,2001,8(11):90-92.
[23]张亮, 陈肇雄, 黄河燕. 问题分类的计算型研究[J]. 计算机科学, 2006, 33(4):9-12.
[24]郑实福, 刘挺, 秦兵,等. 自动问答综述[J]. 中文信息学报, 2002, 16(6):46-52.
[25]周丽霞. 网络信息检索研究综述[J]. 情报科学, 2004, 22(4):395-399.
[26]Voorhees E M, Buckland L P. The {Eleventh Text Retrieval Conference{(TREC 2002)[J]. 2002.
[27]Aliod D M, Berri J, Hess M. A real world implementation of answer extraction[C]// International Workshop on Database and Expert Systems Applications. IEEE Computer Society, 1998:143.
[28]Lakhani P, Kim W, Langlotz C P. Automated extraction of critical test values and communications from unstructured radiology reports: an analysis of 9.3 million reports from 1990 to 2011[J]. Radiology, 2012, 265(3):809.
[29]Chapman W W, Fizman M, Chapman B E, et al. A comparison of classification algorithms to automatically identify chest X-ray reports that support pneumonia.[J]. Journal of Biomedical Informatics, 2001, 34(1):4.
[30]Zingmond D,Lenert LA.Monitoring free-text data using medical language processing[J].Comput Biomed Res,1993,26(5):467-481.
[31]Sada Y, Hou J, Richardson P, et al. Validation of Case Finding Algorithms for Hepatocellular Cancer From Administrative Data and Electronic Health Records Using Natural Language Processing.[J]. Medical Care, 2013, 54.
[32]Carrell D S, Halgrim S, Tran D T, et al. Using natural language processing to improve efficiency of manual chart abstraction in research: the case of breast cancer recurrence[J]. American Journal of Epidemiology, 2014, 179(6):749.
[33]Do B H, Wu A, Biswal S, et al. Informatics in radiology: RADTF: a semantic search-enabled, natural language processor-generated radiology teaching file[J]. Radiographics, 2010, 30(7):2039-48.
[34]Chang E K, Yu C Y, Clarke R, et al. Defining a Patient Population With Cirrhosis: An Automated Algorithm With Natural Language Processing.[J]. Journal of Clinical Gastroenterology, 2016, 50(10):889.
[35]Masino A J, Grundmeier R W, Pennington J W, et al. Temporal bone radiology report classification using open source machine learning and natural langue processing libraries[J]. Bmc Medical Informatics & Decision Making, 2016, 16(1):65.
[36]Dutta S, Long W J, Brown D F, et al. Automated detection using natural language processing of radiologists recommendations for additional imaging of incidental findings.[J]. Annals of Emergency Medicine, 2013, 62(2):162-169.
[37]Ip I K, Mortele K J, Prevedello L M, et al. Focal cystic pancreatic lesions: assessing variation in radiologists' management recommendations.[J]. International Journal of Medical Radiology, 2011, 259(1):136-41.
[38]Jr D R, Nossal M, Schofield L, et al. Physician documentation deficiencies in abdominal ultrasound reports: frequency, characteristics, and financial impact[J]. Journal of the American College of Radiology, 2012, 9(6):403-408., Prevedello L M, et al. Repeat abdominal imaging examinations in a tertiary care hospital.[J]. American Journal of Medicine, 2012, 125(2):155-161.
[39]Ip I K, Mortele K J, Prevedello L M, et al. Repeat abdominal imaging examinations in a tertiary care hospital.[J]. American Journal of Medicine, 2012, 125(2):155-161.
[40]Dang P A, Kalra M K, Blake M A, et al. Natural language processing using online analytic processing for assessing recommendations in radiology reports.[J]. Journal of the American College of Radiology, 2008, 5(3):197-204.
[41]Patel T A, Puppala M, Ogunti R O, et al. Correlating mammographic and pathologic findings in clinical decision support using natural language processing and data mining methods[J]. Cancer, 2017, 123(1):114.