kmeans法(K均值法)是麦奎因提出的,这种算法的基本思想是将每一个样本分配给最靠近中心(均值)的类中,具体的算法至少包括以下三个步骤:
1.将所有的样品分成k个初始类;
2.通过欧氏距离将某个样品划入离中心最近的类中,并对获得样品与失去样品的类重新计算中心坐标;
3.重复步骤2,直到所有的样品都不能在分类为止
kmeans法与系统聚类法一样,都是以距离的远近亲疏为标准进行聚类的。但是两者的不同之处也很明显:系统聚类对不同的类数产生一系列的聚类结果,而K均值法只能产生指定类数的聚类结果。具体类数的确定,离不开实践经验的积累。有时也可借助系统聚类法,以一部分样本(简单随机抽样)为对象进行聚类,其结果作为K均值法确定类数的参考。
kmeans算法以k为参数,把n个对象分为k个聚类,以使聚类内具有较高的相似度,而聚类间的相似度较低。相似度的计算是根据一个聚类中对象的均值来进行的。kmeans算法的处理流程如下:随机地选择k个对象,每个对象初始地代表了一个簇的平均值或中心;对剩余的每个对象,根据其与各个聚类中心的距离将其赋给最近的簇;重新计算每个簇的平均值作为聚类中心进行聚类。这个过程不断重复,直到准则函数收敛,得到最终需要的k个类,算法结束;准则函数通常采用最小平方误差准则:
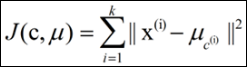
关于k具体数值的选择,在实际工作大多数是根据需求来主观定(如衣服应该设计几种尺码),在这方面能够较直观的求出最优k的方法是肘部法则,它是绘制出不同k值下聚类结果的代价函数,选择最大拐点作为最优k值。
而在Python与R中都各自有实现K-means聚类的方法,下面一一介绍:
Python
Python的第三方包中可以用来做Kmeans聚类的包有很多,本文主要介绍Scipy和sklearn中各自集成的方法;
1.利用Scipy.cluster中的K-means聚类方法
scipy.cluster.vq中的kmeans方法为kmeans2(data,n),data为输入的样本数据矩阵,样本x变量的形式;n为设定的聚类数。
这里我们分别生成5个100x10的高维正态分布随机数,标准差均为0.8,均值分别为1,2,3,4,5,并将其拼接为500x10的矩阵,并按行打乱顺序进行聚类,鉴于维度为10大于2,为了在二维平面上进行可视化,我们使用sklearn包中的降维方法TSNE来对样本数据进行10维至2维的降维以可视化,具体代码如下:
import numpy as np from scipy.cluster.vq import * import matplotlib.pyplot as plt from sklearn.manifold import TSNE '''生成示例数据''' set1 = np.random.normal(1,0.8,(100,10)) set2 = np.random.normal(2,0.8,(100,10)) set3 = np.random.normal(3,0.8,(100,10)) set4 = np.random.normal(4,0.8,(100,10)) set5 = np.random.normal(5,0.8,(100,10)) '''将两个列数相同的矩阵上下拼接,类似R中的rbind()''' data = np.concatenate((set1,set2,set3,set4,set5)) '''按行将所有样本打乱顺序''' np.random.shuffle(data) '''进行kmeans聚类''' res, idx = kmeans2(data,5) '''为不同类的样本点分配不同的颜色''' colors = ([([0.4,1,0.4],[1,0.4,0.4],[0.4,0.4,0.4],[0.4,0.4,1.0],[1.0,1.0,1.0])[i] for i in idx]) '''对样本数据进行降维以进行可视化''' data_TSNE = TSNE(learning_rate=100).fit_transform(data) '''绘制所有样本点(已通过聚类结果修改对应颜色)''' plt.scatter(data_TSNE[:,0],data_TSNE[:,1],c=colors,s=12) plt.title('K-means Cluster')
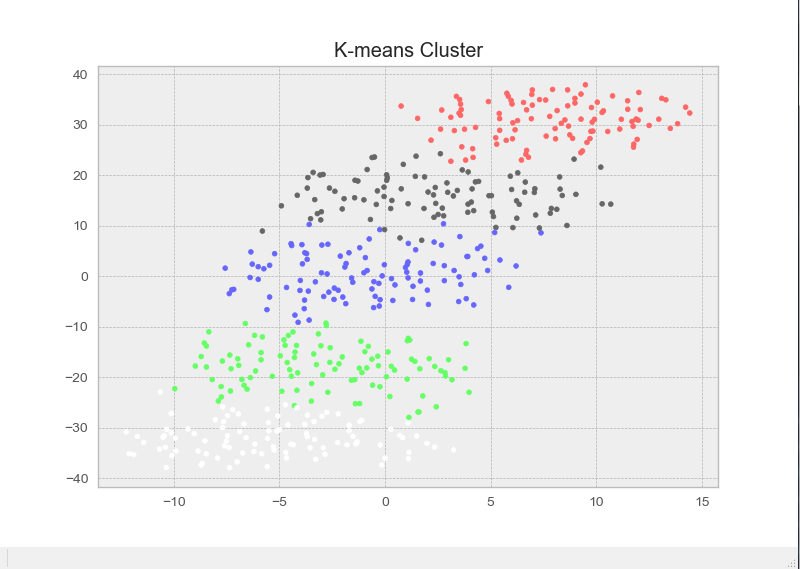
可以看出,我们通过kmeans顺利的将这些数据分到五个类中(有一类颜色为白色),足以见得kmeans在对常规数据的聚合上效果较好,下面我们假装事先不知道样本数据准确的分类数目,利用肘部法则来选取最优k值:
import numpy as np from scipy.cluster.vq import * import matplotlib.pyplot as plt from sklearn.manifold import TSNE from scipy.spatial.distance import cdist from matplotlib.ticker import MultipleLocator xmajorLocator = MultipleLocator(1) '''生成示例数据''' set1 = np.random.normal(1,0.5,(100,10)) set2 = np.random.normal(8,0.5,(100,10)) '''将两个列数相同的矩阵上下拼接,类似R中的rbind()''' data = np.concatenate((set1,set2)) '''按行将所有样本打乱顺序''' np.random.shuffle(data) K = range(1,5) cost = [] '''记录代价变化情况''' for k in K: res, idx = kmeans2(data, k) cost.append(sum(np.min(cdist(data,res,'euclidean'),axis=1))/data.shape[0]) '''绘制代价变化图''' ax = plt.subplot(111) plt.plot(K,cost) ax.xaxis.set_major_locator(xmajorLocator) plt.title('cost change')

可以看出,在k=2的时候,我们找到了对应的‘肘部’,这与真实的类数相同,下面我们进行真实类数较多时的k值选择:
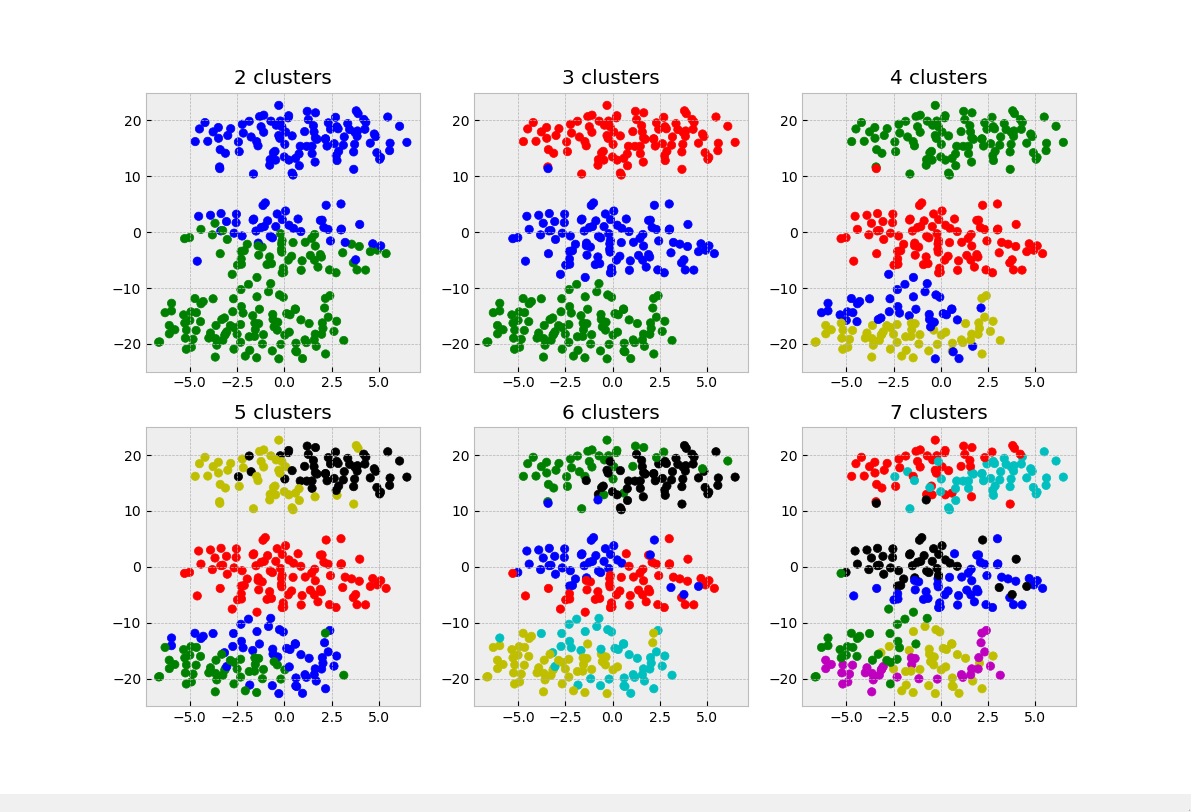
import numpy as np from scipy.cluster.vq import * import matplotlib.pyplot as plt from sklearn.manifold import TSNE from scipy.spatial.distance import cdist from matplotlib.ticker import MultipleLocator from matplotlib import style xmajorLocator = MultipleLocator(1) '''生成示例数据''' set1 = np.random.normal(1,1,(100,10)) set3 = np.random.normal(2,1,(100,10)) set4 = np.random.normal(3,1,(100,10)) set6 = np.random.normal(4,1,(100,10)) '''将两个列数相同的矩阵上下拼接,类似R中的rbind()''' data = np.concatenate((set1,set3,set4,set6)) '''按行将所有样本打乱顺序''' np.random.shuffle(data) K = range(2,6) cost = [] '''记录代价变化情况''' plt.figure(figsize=(12,8)) for k in K: res, idx = kmeans2(data, k) '''为不同类的样本点分配不同的颜色''' colors = ([['red','blue','black','yellow','green'][i] for i in idx]) '''对样本数据进行降维以进行可视化''' data_TSNE = TSNE(learning_rate=100).fit_transform(data) '''绘制所有样本点(已通过聚类结果修改对应颜色)''' plt.subplot(219+k) style.use('bmh') plt.scatter(data_TSNE[:, 0], data_TSNE[:, 1], c=colors, s=12) plt.title('K-means Cluster of {}'.format(str(k))) cost.append(sum(np.min(cdist(data,res,'euclidean'),axis=1))/data.shape[0]) '''绘制代价变化图''' ax = plt.subplot(111) plt.plot(K,cost) ax.xaxis.set_major_locator(xmajorLocator) plt.title('cost change')
以下是不同类数时的聚类图以及代价函数变化图:
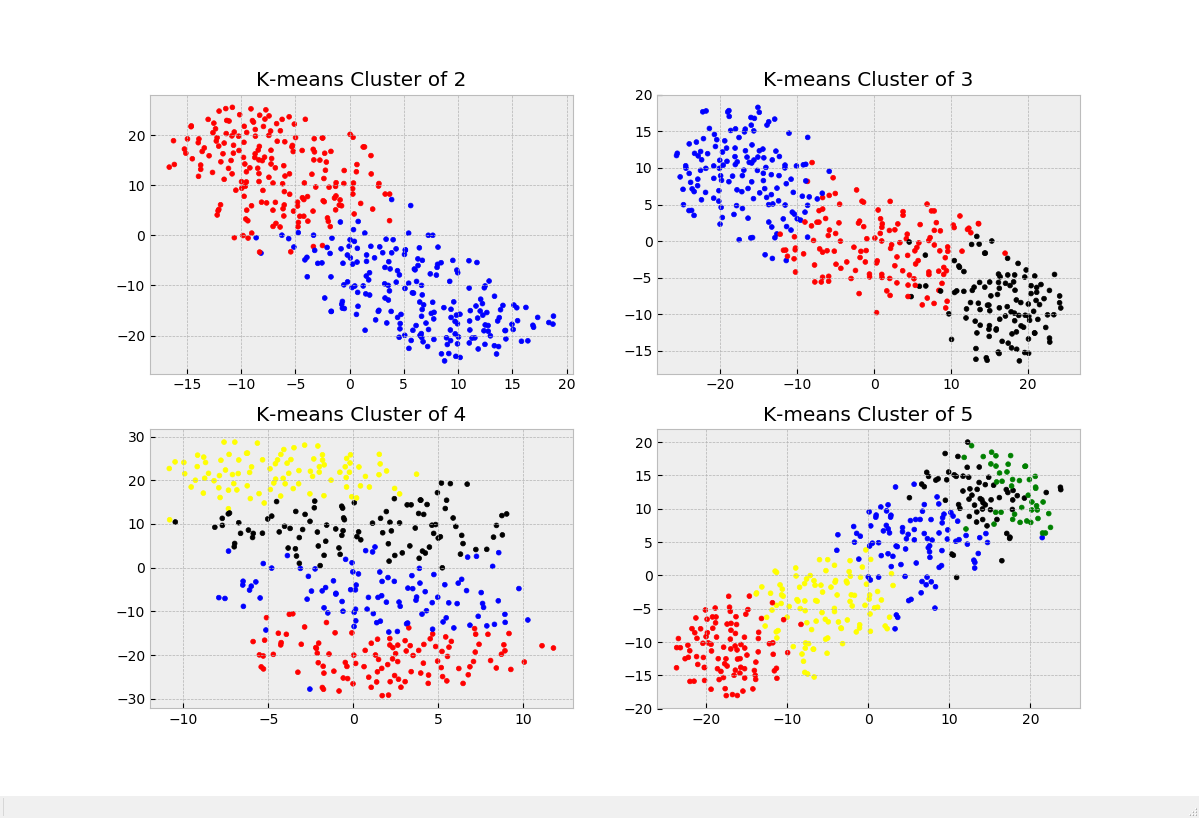
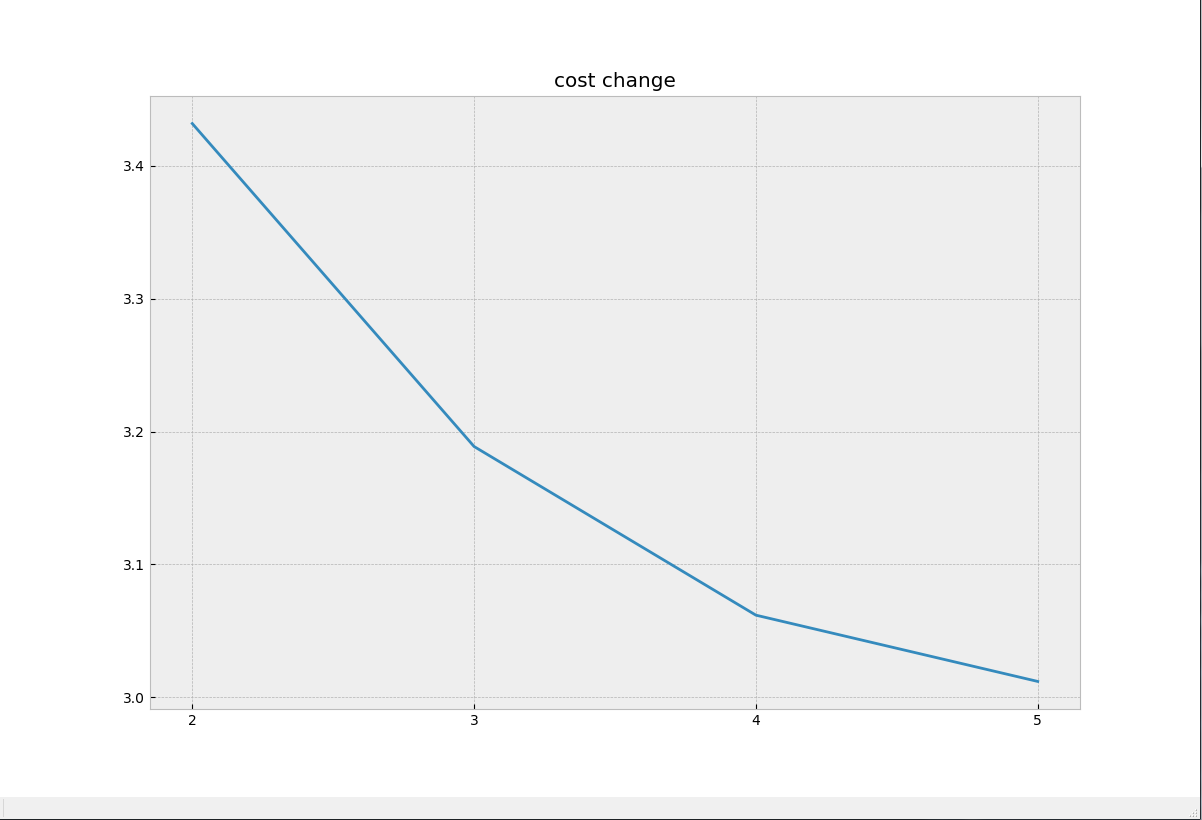
可以看出,在各个类的真实分类较为均匀的时候,肘部法则就失去了意义,因为这时我们无法分辨代价函数的减小是得益于k选的好还是k值的增大。
2.利用sklearn中的方法进行K-means聚类
作为Python中赫赫大名的机器学习包,sklearn中封装的kmeans算法也非常成熟稳定,sklearn.cluster中的KMeans(n_clusters=n,init,n_jobs).fit(data):n_clusters表示设定的聚类个数k,默认为8;init表示初始选择簇中心的方法,有‘kmeans++’与‘random’;n_jobs用来控制线程,-1为CPU全开
from sklearn.manifold import TSNE
from sklearn.cluster import KMeans
import numpy as np
import matplotlib.pyplot as plt
'''生成聚类样本数据'''
set1 = np.random.normal(1,0.7,(100,10))
set2 = np.random.normal(2,0.7,(100,10))
set3 = np.random.normal(3,0.7,(100,10))
data = np.concatenate((set1,set2,set3))
np.random.shuffle(data)
'''生成可视化需要的2维降维数据'''
data_tsne = TSNE(learning_rate=100).fit_transform(data)
colors = ['b', 'g', 'r', 'y', 'k', 'c', 'm', 'chartreuse']
plt.figure(figsize=(12,8))
'''对不同的k值设定进行聚类并绘图'''
for i in range(2,8):
kmeans_model = KMeans(n_clusters=i,init='random',n_jobs=-1).fit(data)#sklearn中的kmeans方法
color = [colors[k] for k in kmeans_model.labels_]
plt.subplot(229+i)
plt.scatter(data_tsne[:,0],data_tsne[:,1],color=color)
plt.title('{} clusters'.format(str(i)))
从主观上看,k=3时效果最好,这也与真实样本的分布类数一致。
R
在R中做K-means聚类就非常轻松了,至少不像Python那样需要安装第三方包,在R中自带的kmeans(data,centers,iter.max)可以直接用来做K-means聚类,其中data代表输入的待聚类样本,形式为样本x变量,centers代表设定的聚类簇数量,iter.max代表算法进行迭代的最大次数,一般比较正常的数据集不会消耗太多次迭代;下面针对低维样本与高维样本分别进行K-means聚类:
一、低维
这里我们生成两类正态分布随机数据,分别是0均值0.7标准差,和3均值0.7标准差,将其拼接在一起,共10000x2的矩阵作为输入变量,设置k分别等于2,3,4,5来看看聚类结果的不同:
#kmeans聚类法 library(RColorBrewer) data1 <- matrix(rnorm(10000,mean=0,sd=0.7),ncol=2) data2 <- matrix(rnorm(10000,mean=5,sd=0.7),ncol=2) data <- rbind(data1,data2) pch1 <- rep('1',5000) pch2 <- rep('2',5000) #设置颜色 cols<-brewer.pal(n=5,name="Set1") par(mfrow=c(2,2)) for(i in 2:5){ cl <- kmeans(data,centers = i,iter.max = 10) plot(data,col=cols[cl$cluster],pch=c(pch1,pch2),cex=0.9) title('K-means Result') points(cl$centers,col='yellow',pch='x',cex=1.5) }
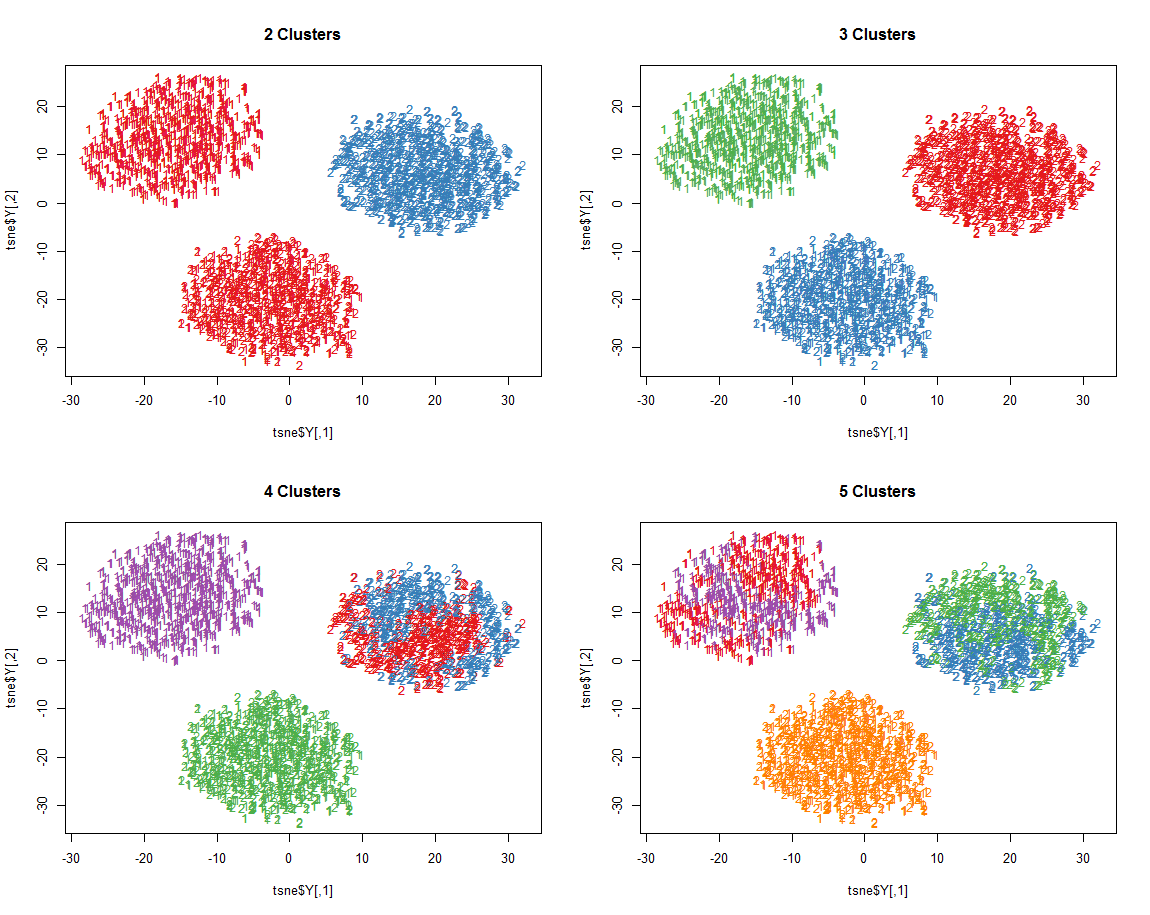
聚类结果可视化如下:

二、高维
当样本数据的维度远远大于3时,就需要对其进行降维至2维以进行可视化,和前面所说的TSNE类似,R中也有同样功能的降维包Rtsne,下面我们就对一个维度较高的(10维)的样本数据集进行聚类及降维可视化:
#kmeans聚类法 library(Rtsne) library(RColorBrewer) data1 <- matrix(rnorm(10000,mean=0,sd=0.7),ncol=10) data2 <- matrix(rnorm(10000,mean=5,sd=0.7),ncol=10) data3 <- matrix(rnorm(10000,mean=10,sd=0.7),ncol=10) data <- rbind(data1,data2,data3) #设置对应样本的点型 pch1 <- rep('1',length(data[,1])*0.5) pch2 <- rep('2',length(data[,1])*0.5) pch3 <- rep('3',length(data[,1])*0.5) #设置颜色 cols<-brewer.pal(n=5,name="Set1") #数据的降维(降至2维) tsne <- Rtsne(data) #自定义代价函数计算函数 Mycost <- function(data,centers_){ l <- length(data[,1]) d <- matrix(0,nrow=l,ncol=length(centers_[,1])) for(i in 1:l){ for(j in 1:length(centers_[,1])){ dd <- 0 for(k in 1:length(data[1,])){ dd <- dd + (data[i,k]-centers_[j,k])^2 } d[i,j] <- sqrt(dd) } } mindist <- apply(d,1,min) return(sum(mindist)) } opar <- par(no.readonly = TRUE) #初始化代价函数记录向量 zb <- c() par(mfrow=c(2,2)) #分别根据不同的k进行聚类并可视化 for(i in 2:5){ cl <- kmeans(data,centers = i,iter.max = 10) plot(tsne$Y,col=cols[cl$cluster],pch=c(pch1,pch2,pch3),cex=0.9) title(paste(as.character(i),'Clusters')) zb[i-1] <- Mycost(data,cl$centers) } par(opar)
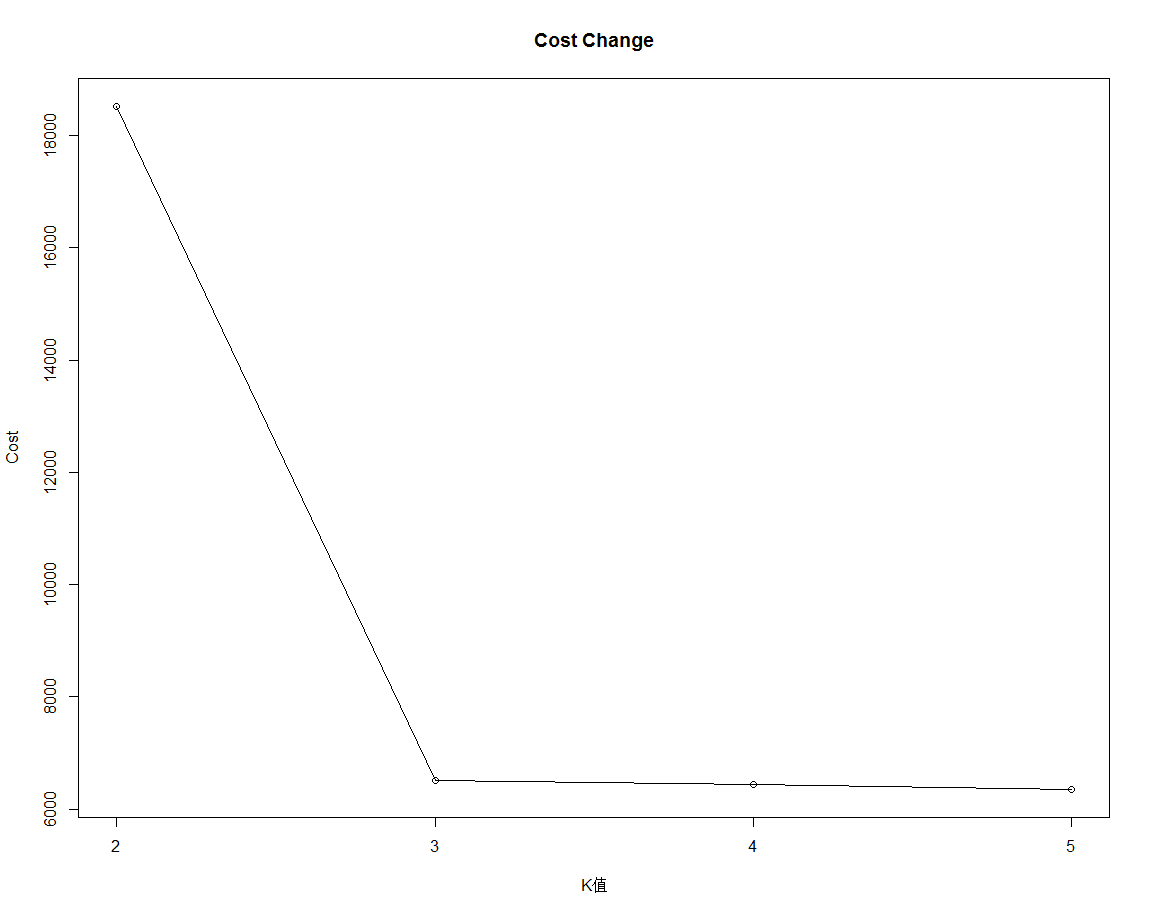
绘制代价函数变化图:
#绘制代价函数随k的增加变化情况 plot(2:5,zb,type='o',xaxt='n',xlab='K值',ylab='Cost') axis(1,at=seq(2,5,1)) title('Cost Change')
总结:Python与R在K-means上各有各的有点,Python方法众多,运算速度快,只是一些细节不够到位;R非常专业,过程也很简洁,只是在运算速度上稍逊一筹,如果让笔者以后实际工作选择的话,我还是更倾向于R,因为其kmeans函数非常“听话”,严格按照设置的类去分,虽然有时会出现一些不合理的地方;Python则没那么“听话”,有些时候,如果被它的算法内部判定为不可再分,则它会在设定的K值下产生一些任性的空类。
如有发现笔者写错的地方,望多多指出。