Datta, K., Gururaj, K., Naik, M., Narvaez, P. & Rutar, M. GenomicsDB: storing genome data as sparse columnar arrays. https://www.intel.com/content/dam/www/public/us/en/documents/white-papers/genomics-storing-genome-data-paper.pdf (2017).
考虑到VCF等基因组数据格式将基因组数据存储为片段,而非对整条染色体都进行记录,作者开发了一款支持分布式存储的数据库系统GenomicsDB。该系统的核心存储采用了TileDB,作者开发了GenomicsDB以执行从生物基因组数据向TileDB数据的转换、对分布式存储的支持等特性。为了便于用户使用,作者还为GenomicsDB开发了Java、Python和Scala(供Apache Spark分布计算时使用)语言的接口。

背景
现在(作者发稿时),高通量测序能够在几天内高通量且便宜地测出人类基因组的序列。进而,全世界每年都有成百万的病人被测序。他们的基因组被用来预测癌症、糖尿病等多种应用。这些进步拥有着从根本上改变我们原本所认为的医疗服务的潜力。
接下来,作者介绍了GATK软件在测序数据上的处理流程。
在GWAS的广泛使用之中,存在着许多常见的挑战:
- 变异数据十分巨大且在持续增长
- 需要可扩展且高效的数据检索
- 需要高效的数据格式转换
针对这些问题,常见的解决思路有:
- 采用可扩展的文件系统或对象存储系统
- 创建合并的、经索引的VCF/gVCF文件
- 使用数据库引擎
因此,作者开发了GenomicsDB,其主要贡献有:
- 高性能的阵列数据存储管理
- 提供了快速高效的C++库,来向TileDB中写入来自许多样本的VCF/gVCF数据
- 提供了快速高效的C++库,来从TileDB中读出数据,并传递给基因组分析工具,如GATK
- 为Apache Spark提供了接口以分布式地处理大数据集
存储细节
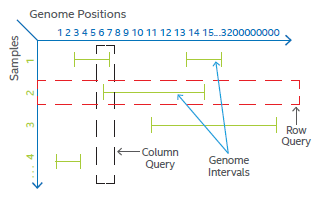
首先,变异数据是天然稀疏(sparse)的。作者将变异数据存储在了一个二维阵列数据结构中。下图展示了两条VCF记录。作者在将VCF向TileDB存储时,由于索引的差异,需要将基因组物理位置(自1起计数)的值减一,作为TileDB的列索引值(自0起计数)。

下图为稀疏的变异数据在TileDB中存储的示意图。每行代表一个样本,每列代表一个位点。绿色的代表一条条基因组片段。
作者描述,其基于如下理由采用了TileDB:
- 变异数据是稀疏的
- 变异数据可以存储为二维阵列(2D array)
- TileDB采用了“柱状机制”(columnar mechanisms)来存储。
- 即TileDB将阵列的每项属性(each attribute of the array,应该指每一列)存储在不同的文件,因此只有相关的文件会被读取。相较于按行排序的存储系统如PostgreSQL,这种策略降低了磁盘访问次数。
- 用户可以指定array中cell的存储顺序(采用按行排序、按列排序、hilbert排序等)
- TileDB提供了高效的存储和检索
文件导入与查询
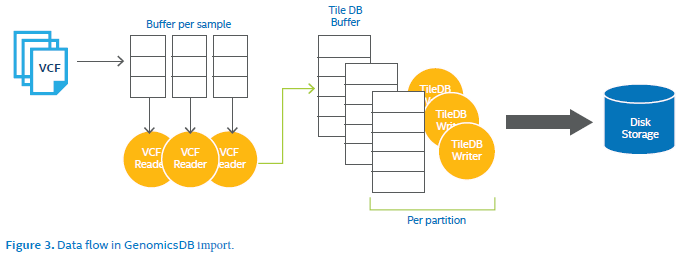
在文件导入中,GenomicsDB支持以多线程形式并行读入VCF文件,并填充到TileDB缓冲区中。TileDB缓冲区直接被传递到TileDB写入接口中,并被序列化为array cell、写入磁盘。通过设置参数,用户可以调整TileDB buffer大小。
文件导入过程还支持批处理。例如1000个样本可划分为500个样本分两次导入。
作者接着讨论了关于TileDB分布式存储的细节。
随后,作者解释了重要参数tile size的设定思路,介绍了批处理特性和分布式存储时的分区管理策略。其中,分区管理策略上分为了按行分区和按列分区。(不同分区存储在不同机器上)
接着,作者表示一些“Mapping Data”将会被存储在关系型数据库上。这些数据包括VCF Header信息、合并方法A combination function (median, concatenation or mean) used while creating a combined VCF from all samples、从基因组物理位置向TileDB列索引的映射关系等。
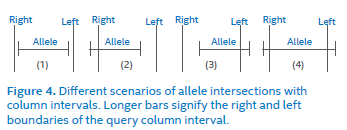
在查询策略上,作者举例了下图四种情形,阐述了cell的边界搜索策略。
性能评估
作者用四页纸的图表文字介绍了GenomicsDB在一些读写性能评估项目上的表现。测定的因素包括:
- 单个tile包含不同cell数
- 并行读取
- 不同segment长度
- 压缩级别
- 分区数量
- 样本数