rasmol软件简介及详细使用说明:
目前在结构生物学领域有许多图形显示的程序,每个都有自己不同的特点。可能很多人知道rasmol,除了图形界面中的一些功能外,该程序的命令行方式有着很强大的功能。下面将介绍一些常用的使用方法。
Rasmol(http://www.openrasmol.org/)程序有多种版本,有unix, windows, Mac等。另外还有一个windows和linux版本http://www.geneinfinity.org/rastop/,该版本将一些原来rasmol菜单下没有的命令加入菜单,比原来的版本方便。
在unix系统下,读入结构文件可以直接用命令的方式,如 rasmol 1crn.pdb. 在windows下,可以先打开raswin, 然后在File的菜单下读入结构文件。Rasmol 所识别的文件有下面几种:
pdb: Brookhaven Protein Databank,来源于 www.rcsb.org
nmrpdb NMR multi-pdb file format
mopac mopac file format; either cartesian or z-matrix format。
mdl Molecular Design Limited's MOL file format
mol2 Tripos' Sybyl Mol2 file format。
xyz MSC's XMol XYZ file format。
alchemy Tripos' Alchemy file format。
charmm CHARMm file format。
如果想读入Charmm软件包的结构文件,用命令行的方式是:rasmol –charmm 1crn.crd.
在打开rasmol后,会出现两个窗口,一个是图形窗口,另外一个是命令行窗口。可以在图形窗口中进行一系列的结构操作,但是有些的操作还需要命令行来补充。下面就将常用的命令进行一下总结。
restrict protein: 在图形窗口中去除所有的非蛋白质原子。
restrict lys: 在图形窗口中去除所有的非lys残基。
select all: 选择所有的原子。
select protein: 选择蛋白质原子。
select hetero: 选择非蛋白质,非DNA原子。
一、常用的选择命令:
1. 结构文件中链的选择:
每条链都有一个字母或数字表示。选择一条链时,必须用:或*说明字母代表链。比如:
select :d 选择d链的所有原子
select *d 选择d链的所有原子
select :d,:e 选择d或e链的所有原子
select glu:2 选择2号链的所有glu。
2. 通过残基名称选择
PDB文件中每个残基都有1-3个字符串的名称。所有的氨基酸用3字符表示,DNA,RNA用单字符表示。水分子用HOH表示。其他的配体的名称可以用文本编辑器打开PDB文件搜索,配体原子对应的坐标在文本中由HETATM开头,而蛋白质或DNA的原子是以ATOM开头,见下面的例子:
ATOM 1902 O GLY R 62 -32.180 -32.765 46.907 1.00 38.84
HETATM 1955 O HOH 1 -26.069 -22.429 17.059 1.00 53.88
有些基团的名称含有数字,如SO3,PO4。在选择这些残基时,残基需要加上方括号,如select [SO3]. 钙原子或其他金属原子一般用2字符表示,如CA, MN,MG, ZN. 如果需要选择钙原子,可以用”select ca”. 钙原子ca的表示与蛋白质的a碳原子表示冲突,因此如果PDB结构中有钙原子,只想选择蛋白质的a碳原子,可以用select protein and *.ca. 另外一些例子有:
select lys:a 选择A链的所有lys.
Select (lys,arg) and :b 选择B链的lys或arg。
3. 通过残基的数字选择
每个残基都有一个数字相对应,下面是一些选择的例子:
Select 32 选择每条链的32号残基以及32号杂原子
Select 19-32 选择每条链的19-32号残基以及19-32号杂原子
select 19-32 and not hetero 选择每条链的19-32号残基
select 19-32 and hetero 选择19-32 号杂原子
select 19-32:y 选择Y链的19-32号残基
select asp47 选择所有链的47号位置的asp
4. 原子的选择
PDB文件中每个原子都有一个序列号对应,可以在图形界面上点击,查看序列号。如果想选择原子可以用:
Select atomno = 131 选择第131个原子
Select atomno = 217, atomno = 1426 选择第217和1426号原子
select atomno >= 195 and atomno <= 277 选择195-277号原子
PDB原子名称,PDB文件中所有的原子的命名采用标准命名,CA表示a-碳原子CB表示b-碳原子,以此类推。CG, CD, CE, CZ, CH (gamma, delta, epsilon, zeta, eta)。N7 (7th nitrogen in a residue), O2P (second oxygen on a phosphorus), OE2 (second oxygen on an epsilon carbon), HD1 (1st hydrogen on a delta carbon). 可以在图形界面中点击原子,然后在命令行窗口上读取原子信息。其他实例:
select *.cg 选择所有g位的碳原子。
select lys.cg 选择所有赖氨酸的g位的碳原子
select :a.cg 选择A链所有g位的碳原子
select lys:a.cg 选择A链所有赖氨酸的g位的碳原子
select 27-42:a.cg 选择A链从27到42位氨基酸的g位的碳原子
select *.h? 选择所有2字符的氢原子
select *.h??? 选择所有的氢原子。
元素名称,键入全名,如magnesium, iron, sulfur等。
另外一些选择实例:
用户可以自定义一些区域进行操作,比如:
define activesite(15,67,109)
select activesite
color green
另外可以通过下面的命令选择某个残基周围的其他原子:
select within(4.5, ser72) 选择ser72周围4.5Å内的原子。
如果不想选择该范围内的某个残基,可以用:
select within(4.5, ser72) and not lys80
二、进一步的操作:
在选择了一些基团 后,就可以对它们进行进一步的操作,比如修改残基的表示方法,可以变成球棍模型,空间堆积模型等等。也可以对它们进行不同的着色。rasmol提供了多种图形显示方法,对原子的显示有wireframe,spacefill,sticks,ball and stick,对于二级结构的显示有ribbons,strands和cartoons。另外还有backbone的显示方法。
在选择了一个残基后,可以有下列操作:
select 172:A
color green 将该残基着绿色
wireframe 0.5 数字表示一个相对值,此时可以看见该残基变粗了。
此时可以在图形界面中的display菜单中选择sticks ,spacefill或ball and stick的方式。然后在命令行窗口可以进行参数设定,比如,在将残基变成ball and stick方式后,在键入 spacefill 0.3, 可以发现原子的表示比原来要小,如果在键入wireframe 0.1,可以发现,化学键的表示变细了。通过这种方法,可以很容易区分所感兴趣的残基。
1. Backbone,ribbons,strands,trace
backbone可以将多肽链表示为通过C-碳原子相互连接的方式。Backbone加上数字可以控制化学键的粗细。如果想将化学键表示为虚线,可以用backbone dash的命令。可以用color backbone yellow将其着黄色。
与backbone类似的命令是trace,该命令将backbone表示进行了圆滑处理。Trace temperature的命令,可以用不同粗细来标示结构中温度因子的大小,温度因子越大,标示越粗。
Ribbons,和strands是二级结构的不同表示方法。Ribbons,strands后跟参数可以控制其宽度。
2. Background
可以用 background yellow将图形界面的背景着黄色。
3. Hbonds和Ssbonds
用该命令要求rasmol搜索氢键,rasmol可以报告氢键的数目。可以用hbonds on或hbonds off控制氢键在图形界面中的显示。也可以将氢键表示换成不同的颜色,用color hbonds yellow可以将默认的红色该为黄色。
Ssbonds用于表示二硫键,用法与hhbonds类似。
另外用color hbonds type,可以用不同的颜色表示不同距离范围内的氢键,比如用红色表示螺旋中的氢键,而黄色表示折叠间的氢键,而转角的氢键用洋红色表示。
4. Label
在选择某个残基后,要向对其进行标记,可以用label,同时通过Set fontsize来控制字体大小,用Set fontstroke控制比划宽度。比如选择R链40位的lys的NZ原子可以用:
select lys40:R.NZ
label lys40
color label yellow
就可以在NZ原子的位置表上lys40。
另外label后面跟不同的参数可以控制label的内容,比如:
%a 原子名称,如上例,将只显示NZ
%b %t 晶体学中的B-值或温度因子
%c %s 多肽链名
%e 显示元素名,上例就是N原子。
%i 结构文件中对应的原子号。
%n 结构文件中对应的残基名
%r 结构文件中对应的残基号
%M NMR Model Number (with leading "/")
%A Alternate Conformation Identifier (with leading ";")
用color label yellow 将用黄色标记。
5. Renumber
有时PDB文件中N-末端的第一个残基的位置不是从1开始,而是从其他数字开始,为了处理方便,可以用renumber将其该为1号开始。也可以在renumber后加数字来选择不同的起始。
6. Save
在选择了一些感兴趣的基团后,可以用save命令来保存所选择的基团的坐标。操作是save myfile.pdb。还可以跟不同的文件格式将它们保存为不同的结构文件,可以用save mdl(alchemy或xyz) myfile。
7. Script
如果进行了很多操作,最后想保留这些历史,可以用write script或write rasmol来保存,下次只要调用角本文件即可,操作是write script myfile。调用时可以用source myfile或script myfile。
8. Show
Show的功能较多,可以跟不同的参数,比如:
show information: 可以给出结构文件的信息,比如什么蛋白质,多少肽链等。
show phipsi: 给出所有的phi,psi角度。
show RamPrint: 给出拉氏图(rasmachandran plot)
show selected { group | chain | atom }:给出选择的内容。
show sequence: 给出氨基酸,核酸序列。
show symmetry: 给出晶体结构晶胞参数等。
show translation(rotation,zoom): 在图形界面上平移,转动或缩放后,给出这些信息。
9. Structure
用该命令可以要求Rasmol计算二级结构(dssp算法),可以给出螺旋,折叠的个数。如果PDB结构中有这些信息,将直接使用,而不通过计算。根据这些信息,rasmol定义了helix, sheet以及turn,因此可以用select helix等来选择二级结构,然后进行不同的操作。
10. 图形位置控制
a. Centre
控制旋转中心或滚动中心。比如 centre lys140,将中心从原来整个分子的中心转移至lys140上。
b. Rotate,translate
控制旋转角度,平移位置。比如分子绕x轴旋转10度,用rotate x 10,反向则用rotate x -10。translate的使用同rotate。
c. Zoom
控制缩放大小,比如zoom 100。
d. Reset
显示回到初始显示位置
11. 原子颜色的控制
rasmol有默认的原子颜色,比如碳原子用灰色表示。可以用下面的方式将其变为绿色:select carbon, color green.
12. rasmol的默认定义类
rasmol有自己定义的类,比如芳香族氨基酸对应aromatic,即如果想选择所有的芳香族氨基酸,可以直接用select aromatic。其他的还有:
AT Acidic Acyclic
Aliphatic Alpha Amino
Aromatic Backbone Basic
Bonded Buried CG
Charged Cyclic Cystine
Helix Hetero Hydrogen
Hydrophobic Ions Large
Ligand Medium Neutral
Nucleic Polar Protein
Purine Pyrimidine Selected
Sheet Sidechain Small
Solvent Surface Turn
步骤:
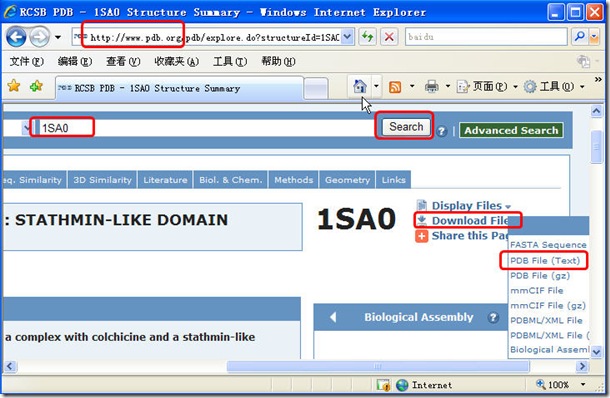
1 下载Rasmol 并安装
2 下载pdb
http://www.pdb.org/pdb/home/home.do
http://www.pdb.org/pdb/results/results.do?qrid=61AE1A7F&tabtoshow=Current
3 用Rasmol 打开下载的pdb文件
REF: