1)将vcf转化为plink格式,假定输入的vcf文件名为:17893893-17898893.vcf,也可以参考链接:将vcf文件转化为plink格式并且保持phasing状态
/vcftools --vcf 17893893-17898893.vcf --plink-tped --out 17893893-17898893
/plink --tfile 17893893-17898893 --recode --out 17893893-17898893
2) 用PLINK确定要研究的位点是否处于连锁的状态;生成blocks和blocks.det两种后缀格式文件;
/plink --file 17893893-17898893 --blocks no-pheno-req --out 17893893-17898893

以上结果说明rs62033246|rs7189274|rs7187782|rs62033247|rs7194607|rs7200159|rs199711091|rs2361776|rs8054992|rs1845376|rs35902958|rs9928657|rs9928743|rs56078865|rs62033249|rs62033250|rs9936044|rs7184460|rs4781927|rs7202082|rs4780669|rs7202439|rs7195939|rs57418698|rs34615631|rs533867711|rs555603620|rs567435894|rs7499772|rs62033251|rs56248612|rs7202488|rs61671028|rs62033253|rs9928974这35个位点是连锁的
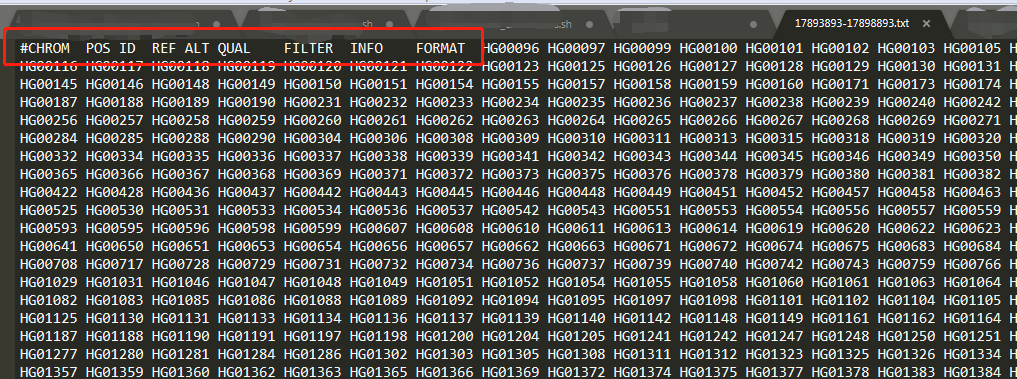
3) 提取这35个位点作为接下来的单倍型网络构建;去掉vcf的头文件,保存为txt格式,见如下图,17893893-17898893.txt:
4)准备17893893-17898893_singstring.txt文件,该文件其实就是去掉 0|0,0|1,1|0,1|1的“|”。

5)接下来,生成两条单链,用R输入如下命令:
install.packages("xlsx")
library(xlsx)
kg_ame<-read.table("E:/17893893-17898893.txt")
kg_ame<-data.frame(kg_ame);
library("stringr");
newdata=c()
for (i in 0:34){
i=i+1
kk=kg_ame[i,6:2509];
kk[which(kk=="0|0")]=paste(kg_ame[i,4],kg_ame[i,4]);
kk[which(kk=="0|1")]=paste(kg_ame[i,4],kg_ame[i,5]);
kk[which(kk=="1|0")]=paste(kg_ame[i,5],kg_ame[i,4]);
kk[which(kk=="1|1")]=paste(kg_ame[i,5],kg_ame[i,5]);
newdata=rbind(newdata,kk)
}
##6:2509指的是2000多个样本对应的碱基;
##0:34指的是35个碱基;这一步是将0|0,0|1,1|0,1|1转化为碱基形式,类似vcf转化为Ped的步骤;
openxlsx::write.xlsx(newdata,file ="E:/17893893-17898893_change.xlsx")
kk=kg_ame[1,7:15];kk
kk[which(kk=="0|0")]=paste(kg_ame[1,4],kg_ame[1,4]);kk
#####分为两条链,转换为ped格式;
library(xlsx)
kg_ame<-read.table("E:/17893893-17898893_singstring.txt")
kg_ame<-data.frame(kg_ame);
library("stringr");
newdata=c()
for (i in 0:34){
i=i+1
kk=kg_ame[i,6:5013];
kk[which(kk=="0")]=kg_ame[i,4];
kk[which(kk=="1")]=kg_ame[i,5];
newdata=rbind(newdata,kk)
}
openxlsx::write.xlsx(newdata,file ="E:/17893893-17898893_changesingstring.xlsx")
se1<-seq(from=1,to=5008,by=2);
se2<-seq(from=2,to=5008,by=2);
stringone<-newdata[,se1];
stringtwo<-newdata[,se2];
openxlsx::write.xlsx(stringone,file ="E:/17893893-17898893_changesingstringone.xlsx")
openxlsx::write.xlsx(stringtwo,file ="E:/17893893-17898893_changesingstringtwo.xlsx")
####以上步骤为转为两条单链
6) 将上述的两条单链生成fas格式;
paste -d " " 17893893-17898893_changesingstringone_firstdot.txt 17893893-17898893_changesingstringone.txt > 17893893-17898893_changesingstringone_dot.fas paste -d " " 17893893-17898893_changesingstringtwo_firstdot.txt 17893893-17898893_changesingstringtwo.txt > 17893893-17898893_changesingstringtwo_dot.fas cat 17893893-17898893_changesingstringone_dot.fas 17893893-17898893_changesingstringtwo_dot.fas >> 17893893-17898893_changesingstring_onetwo_dot.fas #这两条链合并在一起
17893893-17898893_changesingstringone_firstdot.txt 文件格式如下:
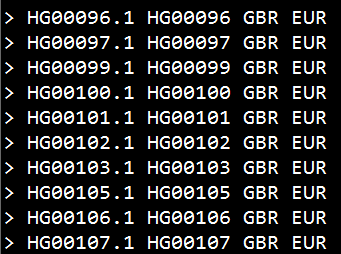
17893893-17898893_changesingstringone.txt的文件格式如下:这个文件就是5)生成出来的其中一条单链。
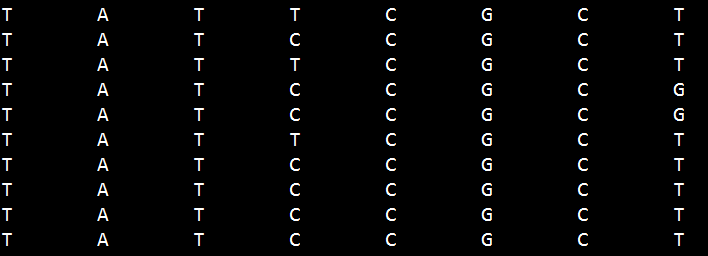
生成的fas文件如下:
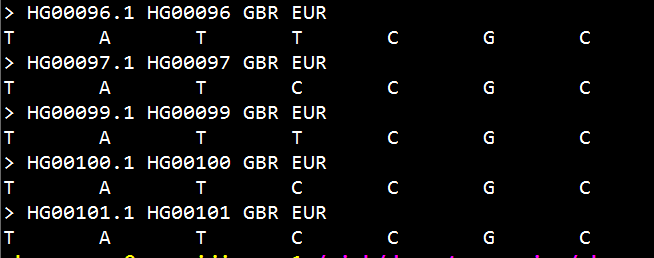
第二条链的处理同上。最后一步,就是把这两条链合并在一起
7)用DnaSP6将fas文件生成nex文件

8)统计nex文件中不同群体的haplotype的数量
准备17893893-17898893_hap.xlsx文件

准备allsample.txt文件
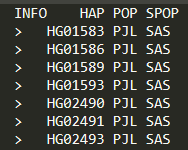
输入如下命令:
#####统计nex生成的所有haplotype所在的样本和群体,这里假定生成了100个haplotype
library(dplyr)
library(xlsx)
model_57hanchip<-read.xlsx("E:/17893893-17898893_hap.xlsx",2)
model_57hanchip<-data.frame(model_57hanchip)
kg_ame<-read.table("E:/allsample.txt")
kg_ame<-data.frame(kg_ame);
newdata=c()
for (i in 0:99){
i=i+1
HAP1<-kg_ame[,2];
df1 <- data.frame(id = HAP1, kg_ame[,3],kg_ame[,4])
HAP2<-model_57hanchip[,i]
df2 <- data.frame(id = HAP2)
k=merge(df1, df2, by = "id",all = FALSE)
j=table(k[,3])
newdata=rbind(newdata,j)
print(i)
}
openxlsx::write.xlsx(newdata,file ="E:/17893893-17898893_hapcount.xlsx")
9)修改生成的nex文件
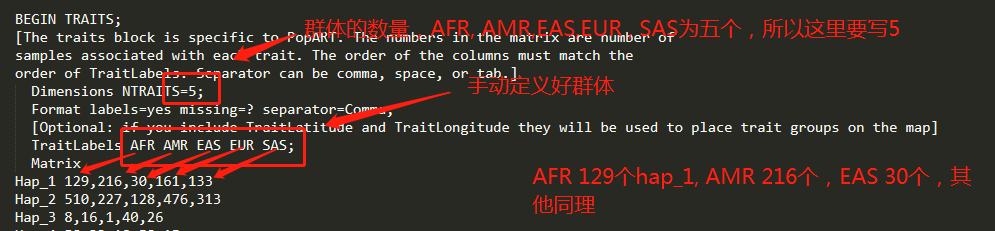
DnaSP6生成的nex文件并没有BEGIN TRAITS的头文件,因此这一步是需要手动修改的。
这一步,除了圈出来的数字和AFR AMR EAS EUR SAS是需要修改,其他照抄。
10)用popart构建单体型网络
导入文件后,选择适合的算法,即可生成。