假设想要计算ATP1A4基因上的外显子碱基覆盖度
首先查询这个基因所有exon的起始和终止位置,查询链接:http://grch37.ensembl.org/Homo_sapiens/Transcript/Exons?db=core;g=ENSG00000132681;r=1:160121360-160156767;t=ENST00000368081
如下图所示,可以看到该基因有6个转录本,假定选择第二个转录本ENST00000470705.1,点击进去,显示该转录本有5个外显子

具体外显子的详细信息如下所示:
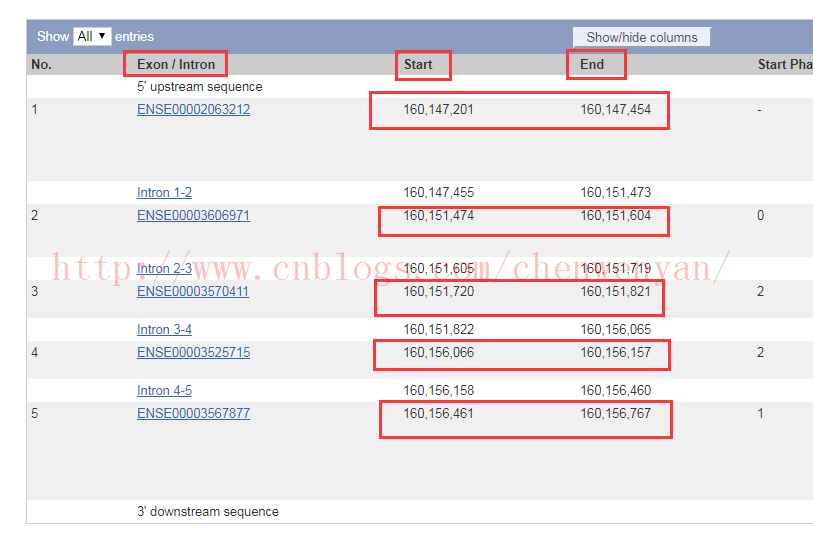
获得该基因的所有外显子的起始终止位置后,就可以用Samtool提取该位置,并且计算碱基覆盖度了,用到的命令如下:
1、先提取外显子的起始终止位置:
/software/samtools view -h file.bam 1:160147201-16014454 > ATP1A4_exon1.sam #提取基因ATP1A4上转录本ENST00000470705.1的第一个外显子片段
2、计算每个exon的碱基覆盖度:
/software/samtools depth ATP1A4_exon1.sam > ATP1A4_exon1_coverage.txt #计算基因ATP1A4上转录本ENST00000470705.1的第一个外显子碱基覆盖度