传统的全基因组关联分析(GWAS)计算的是单个SNP与表型的相关性,除此之外,我们还可以进行SNP之间的互作效应与表型的相关性分析。
本推文主要介绍的是SNP间的上位效应与表型的相关性分析。
上位效应的公式为:Y ~ b0 + b1.A + b2.B + b3.AB + e
Y为表型,A和B分别为两个变异位点,在GWAS中通常指SNP,如果b3为显著,则说明存在上位效应。
1、准备基因型文件
基因型文件:文件格式可以是ped/map或者bed/bim/fam,本教程的格式为ped/map
具体格式准备可参考教程:全基因组关联分析(Genome-Wide Association Study,GWAS)流程(网址 https://www.cnblogs.com/chenwenyan/p/6095531.html)
基因型文件test.ped
基因型文件test.map
2、准备表型文件
表型分为二分类和非二分类表型(多分类或者连续型表型)。
二分类表型文件phenocc.txt: 第一列为FID(可以是群体等),第二列为IID(个体唯一ID),第三列为表型(1是control,2是case)

多分类或者连续型表型文件phenoq.txt: 第一列为FID(可以是群体等),第二列为IID(个体唯一ID),第三列为表型

3、全基因组所有SNP的上位效应分析
二分类表型的命令如下:
/plink-1.07-x86_64/plink --file test --pheno phenocc.txt --epistasis --epi1 1 --noweb --out test
--epi1参数表示设定输出的P的阈值,这里设定为1,指的是输出所有值,PLINK默认输出的P阈值为1e-4。当SNP数量比较多时,建议用PLINK的默认参数或者适当提高阈值,比如1e-5,以此减少输出文件大小。
生成test.epi.cc和test.epi.cc.summary两个文件。
test.epi.cc文件如下所示:

不同列所代表的意思:
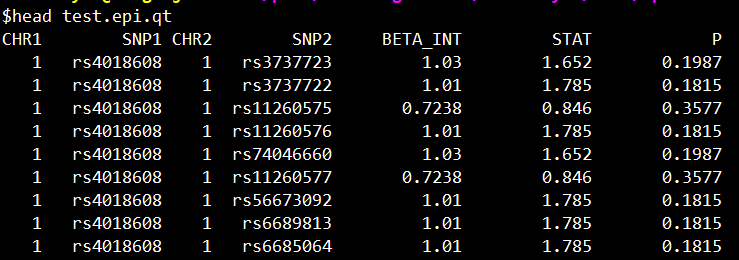
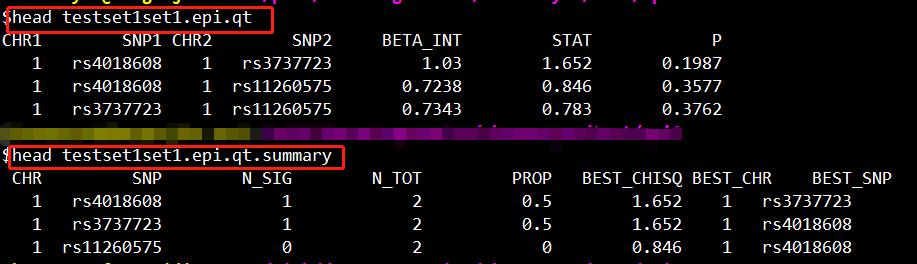
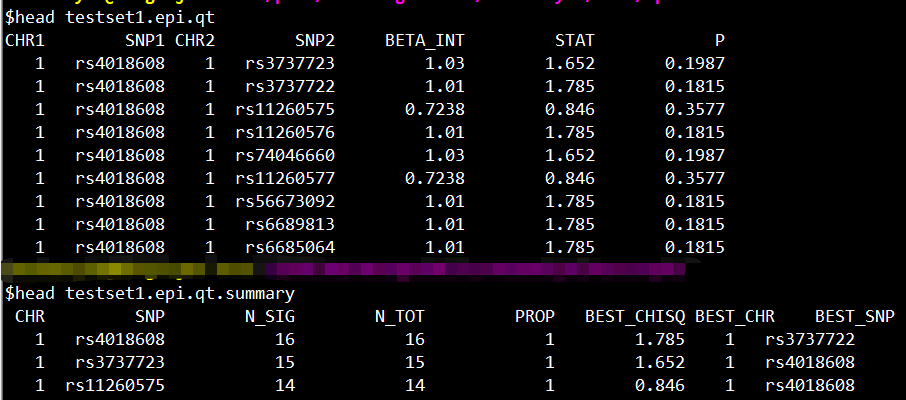
CHR1 Chromosome of first SNP SNP1 Identifier for first SNP CHR2 Chromosome of second SNP SNP2 Identifier for second SNP OR_INT Odds ratio for interaction STAT Chi-square statistic, 1df P Asymptotic p-value
test.epi.cc.summary文件如下所示:
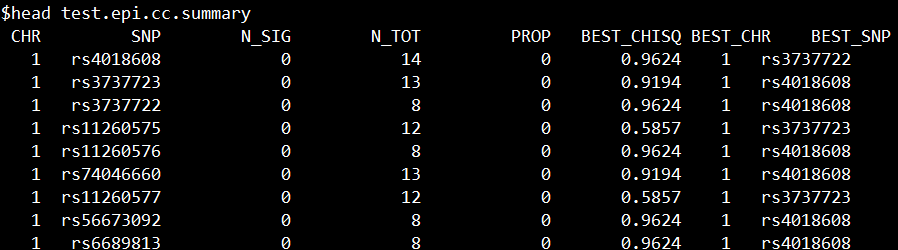
不同列所代表的意思:
CHR Chromosome SNP SNP identifier N_SIG # significant epistatic tests (p <= "--epi2" threshold) N_TOT # of valid tests (i.e. non-zero allele counts, etc) PROP Proportion significant of valid tests BEST_CHISQ Highest statistic for this SNP BEST_CHR Chromosome of best SNP BEST_SNP SNP identifier of best SNP
多分类或连续型表型的命令如下:
/plink-1.07-x86_64/plink --file test --pheno phenoq.txt --epistasis --epi1 1 --noweb --out test
生成test.epi.qt和test.epi.qt.summary两个文件
test.epi.qt文件如下所示:
test.epi.qt.summary文件如下所示:

每列所代表的意思同二分类表型结果文件。
4、指定SNP之间的上位效应分析
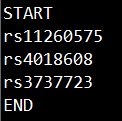
指定三个SNP之间的上位效应分析,假定三个SNP的文件为epi.set1,内容如下:
指定的三个SNP放中间,以START和END作为开头和结尾。
命令如下:
/plink-1.07-x86_64/plink --file test --pheno phenoq.txt --epistasis --set-test --set epi.set1 --epi1 1 --epi2 0.3 --noweb --out testset1set1
--epi2指的是对显著的test进行计数,阈值可以是0.05,0.01等,这里设定为0.3,该值针对N_SIG一列设定的。
示例代码是PLINK的1.07版本,如果是1.9版本,则将--set-test改为set-by-set
生成的结果如下所示:
5、指定SNP与其他SNP的上位效应分析
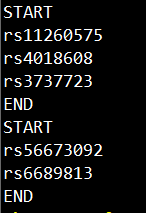
指定rs11260575、rs4018608、rs3737723与rs56673092、rs6689813的上位效应分析,需要文件epi.set2,格式如下:
上位效应分析:
/plink-1.07-x86_64/plink --file test --pheno phenoq.txt --epistasis --set-test --set epi.set2 --epi1 1 --epi2 0.3 --noweb --out testset1set2
结果如下所示:

6、指定SNP与全基因组的上位效应分析
指定SNP与全基因组上位效应分析命令:
/plink-1.07-x86_64/plink --file test --pheno phenoq.txt --epistasis --set-test --set epi.set1 --set-by-all --epi1 1 --epi2 1 --noweb --out testset1
结果如下:
需要用到这批测试数据的公众号“bio生物信息”后台回复“epis”。
