EM算法、GMM - Demo
Intro
这一节学习内容为概率图模型里的一节,因为下午在跑程序手里也没什么事情干,写个EM的demo记录一下。本文也不是来推导过程的,只是方法和代码记录,推导请看其他博客。
GMM
概率图模型跳不过的一章就是高斯混合模型,高斯混合模型是由多个高斯分布以一定权重组成的模型,其概率密度函数等于各高斯模型的概率密度函数加权求和。
一般地,每个高斯分布的概率密度为:
其中k代表第k个高斯分布,因此GMM的概率分布为:
其中(sum_{k=1}^{K}{pi_k} = 1)且(pi_k geq 0).
GMM的一个基本问题就是选用高斯混合模型对数据进行建模,那么模型的参数如何获得呢?
一个GMM的参数仅由一组(、、(pi、mu、sigma))决定,所以我们要估计的参数就是这三个。其涉及的问题是包含隐变量的参数的求解过程,如果模型不含隐变量,直接用最大似然然后求个导就能解决,但是现在模型含有隐变量,也就意味着求导的过程极其复杂,一个比较好的方法是通过EM算法来进行参数估计。
关于GMM的简介就到这里,网上有很多关于GMM的介绍,也非常通俗易懂,不多赘述。
EM
EM算法是一个求解带隐变量的模型的参数常用的方法,其通过最大化训练集的边界似然,迭代更新参数。
过程为:
- 先初始化随机参数
- E步:固定参数求解后验概率
- M步:固定后验概率,优化求解证据下界对应的参数。
- 重复进行直到收敛。
对于GMM模型而言,要求其参数就是按照上面的步骤进行。但在开始之前,我们要先生成给定分布的样本,即采样。采样步骤为:
- 按照(、pi_1、pi_2 dots pi_K)的的分布随机选择一个高斯分布
- 假设选择了第k个高斯分布,按照(N(x|mu_k,sigma_k))的分布采样一个样本(高斯分布的采样计算机可以实现)
E步
固定(mu,sigma),计算后验概率分布(p(z^{(n)}|x^{(n)}))
用上式(gamma_{nk})表示第n个样本对第k个高斯分布的后验概率,其用(N imes K)的矩阵表示。
M步
在已知后验概率的情况下,最大化边际似然(p(x|mu,sigma)),即最大化对数似然:
其中(q(z) = p(z^{(n)}=k)),ELBO通过不等式定义为函数下界,所以我们要求的其实是对数似然的下界。
D为训练集,(gamma)为后验分布。
利用拉格朗日法求解后有如下更新的结论:
其中(N_k = sum_{n=1}^N gamma_{nk})
Coding
'''
@Descripttion: This is Aoru Xue's demo, which is only for reference.
@version:
@Author: Aoru Xue
@Date: 2019-12-30 20:46:28
@LastEditors : Aoru Xue
@LastEditTime : 2019-12-31 00:03:01
'''
from matplotlib import pyplot as plt
import numpy as np
import random
import pickle
# 代码里的循环运算可以矩阵优化,有兴趣且需要请自行优化,高维需要修改部分代码。为保证例子简单,故以一维高斯分布为例。
class GMM():
def __init__(self,pi,mu,sigma,samples): # 生成一个一维高斯混合分布
self.x = np.zeros(shape = (samples,))
for i in range(samples):
idx = np.random.choice(range(pi.shape[0]),p=pi)
self.x[i] = np.random.normal(mu[idx],sigma[idx])
self.mu = np.random.randn(mu.shape[0])
self.sigma = np.ones(sigma.shape[0])
self.pi = np.ones(sigma.shape) / sigma.shape[0]
@staticmethod
def gausian_probablity(x,mu,sigma):
return 1. / np.sqrt(2 * np.pi)/sigma * np.exp(- (x - mu)**2 / 2 / sigma**2)
def EM_method(self,):
post_probability = np.zeros((self.x.shape[0],self.pi.shape[0]),dtype = np.float) # (N,K)
for _ in range(100):
# E
for n in range(post_probability.shape[0]):
for k in range(post_probability.shape[1]):
post_probability[n,k] = self.gausian_probablity(self.x[n],self.mu[k],self.sigma[k]) * self.pi[k]
post_probability = post_probability / np.sum(post_probability,axis = 1,keepdims=True) # 后验概率
# M
N_k = np.sum(post_probability,axis = 0)
for k in range(post_probability.shape[1]):
self.pi[k] = N_k[k] / post_probability.shape[0]
self.sigma[k] = np.sqrt(1./N_k[k] * np.sum(post_probability[:,k] * (self.x - self.mu[k])**2))
self.mu[k] = 1. / N_k[k] * np.sum(post_probability[:,k] * self.x) # (N,1) * (N,1)
return self.pi,self.mu,self.sigma
def draw(pis, mus, sigmas,color="r", tag="real"):
x_min = mus - 3 * mus
x_max = mus + 3 * mus
x_bottom = x_min.min()
x_top = x_max.max()
x_array = np.linspace(x_bottom, x_top, 200)
y_array = np.zeros(200, dtype=float)
for pi, mu, sigma in zip(pis, mus, sigmas):
y_array = y_array + pi * gmm.gausian_probablity(x_array, mu, sigma)
plt.figure(0)
plt.plot(x_array, y_array, color=color, label=tag)
plt.legend()
plt.xlim([x_array.min(), x_array.max()])
if __name__ == "__main__":
pi = np.array([0.1,0.3,0.6])
mu = np.array([-3,1,4])
sigma = np.array([1,1,1])
gmm = GMM(pi = pi,mu = mu,sigma = sigma,samples = 5000)
plt.figure(0)
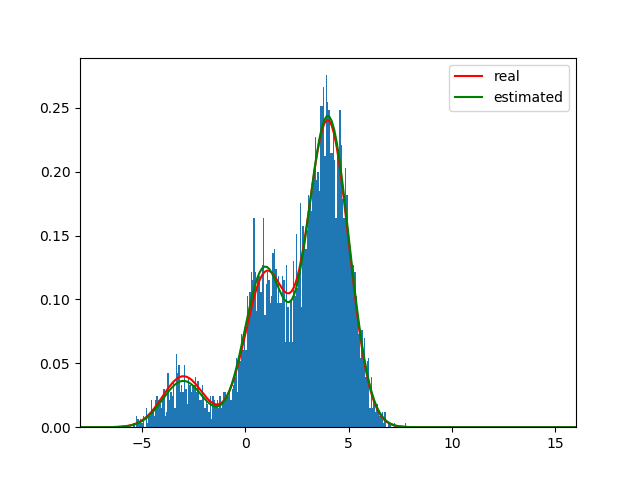
plt.hist(gmm.x, bins=200, density=True)
plt.plot()
draw(pi, mu, sigma, color="r", tag="real")
pi_,mu_,sigma_ = gmm.EM_method()
draw(pi_, mu_, sigma_, color="g", tag="estimated")
plt.show()
Visualization