首先介绍下两种方法:
一、本地分析
1、在STRING数据库下载人的互作文件,如下图,第一个文件
https://string-db.org/cgi/download.pl?sessionId=HGrreq5g8nLI&species_text=Homo+sapiens
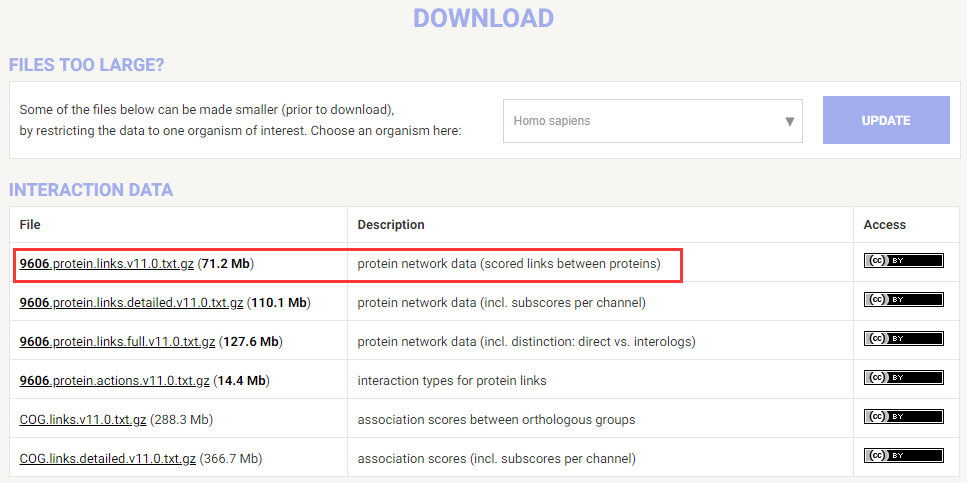
2、在本地电脑上(linux),用grep 检索该蛋白的互作关系:ENSP00000359708 ,发现结果是空
二、直接到STRING数据库进行互作关系分析
1、检索网址
(1)这个支持多个基因ID或者蛋白ID
 https://string-db.org/cgi/input.pl?sessionId=HGrreq5g8nLI&input_page_active_form=multiple_identifiers
https://string-db.org/cgi/input.pl?sessionId=HGrreq5g8nLI&input_page_active_form=multiple_identifiers (2)这个是单个基因ID或者蛋白ID
 https://string-db.org/cgi/input.pl?sessionId=HGrreq5g8nLI&input_page_active_form=single_identifier
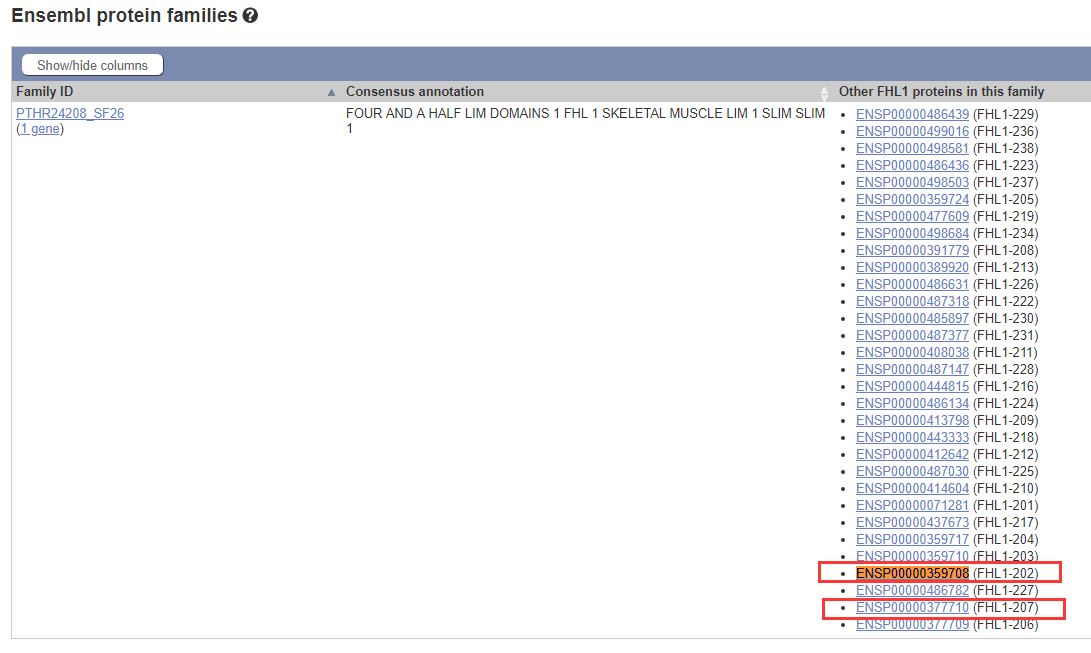
https://string-db.org/cgi/input.pl?sessionId=HGrreq5g8nLI&input_page_active_form=single_identifier 2、输入蛋白ID: ENSP00000359708 ,检索结果如下,该蛋白对应的基因是FHL1 ,点击该节点,会显示该基因与其他基因发生互作关系的蛋白,
发现是这个蛋白:ENSP00000377710 ,这个蛋白和前边那个蛋白是什么关系呢?
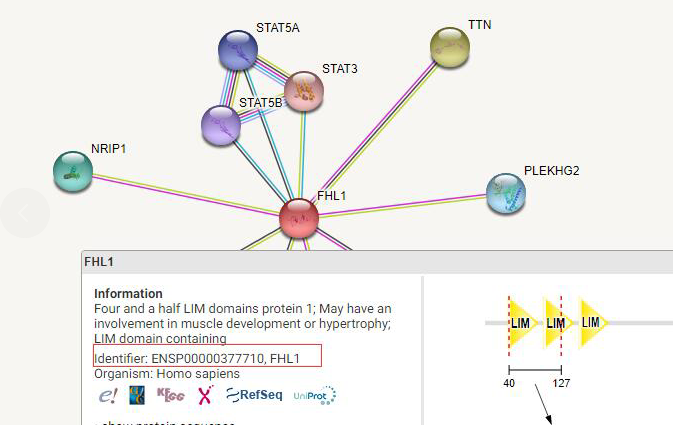
3、通过检索,发现他们同属于FHL1蛋白家族,这样就解释了,为什么在网站中可以检索到了,因为网站展示的基因间的互作,只要改基因对应的蛋白家族中任何一个蛋白与其他基因对应的蛋白家族有互作,就会把该互作关系检索出来。