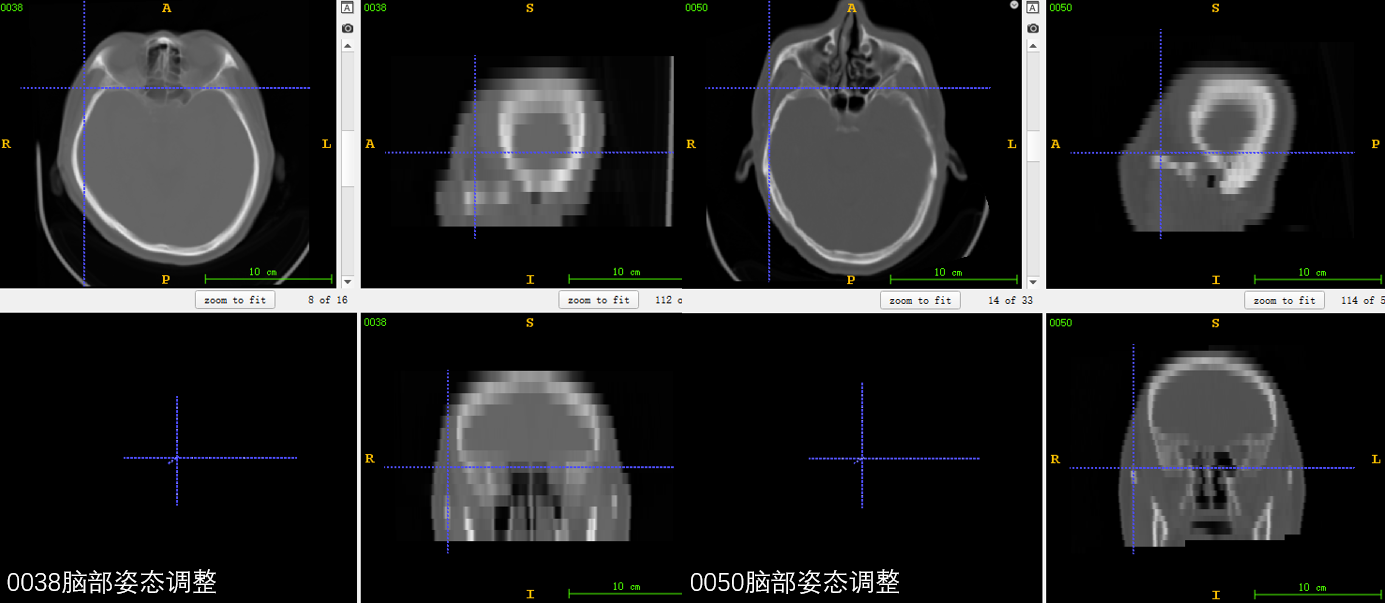
问题描述:项目中需要模型对输入的三维数据中每一层标注的中线mask学习,并对输入的三维数据的每一层进行中线预测。现在预测结果中出现了比数据实际层数还多的mask,根据此预测结果求得三维的中心和中平面,用于后续的旋转等操作会因此导致效果不佳

对于上图中的数据来说,实际一共只有22层,但是却出现了23、24层的预测结果(在mhd文件中层数下标从0开始)
查找原因:由于测试集在进行预测前需要进行和训练集相同的处理,对于resize操作来说,有些数据实际层数不够,我们在resize时是对其采取的补0操作(数据在送入模型前都统一处理成24 * 512 * 512)。模型在预测时按理来说对这些补0的层不会有预测的mask,但我们现在得到的结果中出现了数据实际只有16层却有20层包含mask(对于16层的数据来说,补零了8层,这8层理应不会得到预测结果)
解决方案:将模型的三维输入改为逐层输入,避免模型学到如上所述的先验信息。在数据预处理的时候,将数据的每一层都保存成一个单独的npy文件(之前是将预处理之后的24层一起保存成npy文件)
数据预处理代码:
def readMhd(mhdPath):
'''
根据文件路径读取mhd文件,并返回其内容Array、origin、spacing
'''
itkImage = sitk.ReadImage(mhdPath)
imageArray = sitk.GetArrayFromImage(itkImage) # [Z, H, W]
origin = itkImage.GetOrigin()
spacing = itkImage.GetSpacing()
return imageArray, origin, spacing
def generateHeatmap(mask, sigma):
'''
:param mask: 三维mask
:param sigma: 设置为8,以这两个标记点为中心,生成一个边长为2*sigma+1的正方形,正方形内像素填充为1
:return: 将mask中标记的点扩充成一个区域
'''
for i in range(mask.shape[0]):
if np.max(mask[i, :, :]) == 0:
continue
# 找出当前层不为0的点的坐标,赋值为1,维度顺序Z,Y,X
index = np.where(mask[i, :, :] != 0)
for x, y in zip(index[0], index[1]):
mask[i, max(0, x - sigma):min(511, x + sigma),
max(0, y - sigma):min(511, y + sigma)] = 1.0
return mask
def imageResize(img, mask, H, W):
'''
:param W: 归一化的宽度
:param H: 归一化的高度
:param img: 二维——当前层
:param mask: 二维——当前层
:return: resize成512*512
'''
# print(mask.dtype, mask.min(), mask.max(), mask.shape)
# 使用最近邻插值进行resize
# print('img', img.shape)
if img.shape[0] != 512 or img.shape[1] != 512:
img = transform.resize(img, (H, W), order=0, mode='constant', cval=img.min(),anti_aliasing=True, preserve_range=True)
mask = transform.resize(mask, (H, W), order=0, mode='constant', cval=mask.min(), anti_aliasing=True,preserve_range=True)
#print(img.dtype, img.min(), img.max())
#print(mask.dtype, mask.min(), mask.max())
img = np.array(img, dtype=np.float32)
mask = np.array(mask, dtype=np.uint8)
return img, mask
def preProcess(patientNames, oriDataDir, maskDataDir, targetDataDir):
'''
:param patientNames: 病例号
:param oriDataDir: 数据的外层文件
:param maskDataDir: mask图像的目录
:param targetDataDir: 将要保存的目标目录
:param imageType: 原图的数据类型
:param maskType:
:param replace: 是否需要更新这个目录
:return:
'''
if not os.path.exists(targetDataDir):
os.makedirs(targetDataDir)
# 拼接目标病人文件夹patientTargetDir,生成的原图和mask都放到这个文件夹下
# mask的npy文件后加上_mask
for patientName in patientNames:
print('patientName: ', patientName)
patientTargetDir = os.path.join(targetDataDir, patientName)
# print('patientTargetDir: ', patientTargetDir)
if not os.path.exists(patientTargetDir):
os.makedirs(patientTargetDir)
image, _, _ = readMhd(os.path.join(oriDataDir, patientName + '.mhd'))
mask, _, _ = readMhd(os.path.join(maskDataDir, patientName + '_seg.mhd'))
# print('image_type: ', image.dtype, 'mask_type: ', mask.dtype)
mask = generateHeatmap(mask, 8) # 将mask上的一个像素周围扩充
image, mask = image_resize(image, mask)
# mask = mask.astype(np.uint8) # 将mask转化为uint8类型
# 1.assert用于判断表达式,表达式为False时,触发异常 2.每个numpy数组都具有一个shape属性,它是一个元组,存放的是数组的维数信息# 3.下句若原图和mask维数不匹配就打印异常信息
assert image.shape == mask.shape, "patientName: {}, image shape must be equal to mask shape, but get image {},
mask{}!!!".format(patientName,image.shape,mask.shape)
for layerIdx in range(0, image.shape[0]):
imageSavePath = os.path.join(patientTargetDir, '{}.npy'.format(layerIdx))
# 对每一层resize到512*512
# print('image[Idx].shape: ', image[layerIdx].shape, mask[layerIdx].shape)
image, mask = imageResize(image[layerIdx], mask[layerIdx], 512, 512)
# 将图像按层以numpy的形式保存为.npy格式,存到imageSavePath中(目标病人目录下)
np.save(imageSavePath, image[layerIdx])
# print(imageSavePath, 'saved!')
# 对该层mask图像的二维数组求和,若不为0代表有mask就保存预处理后的mask,没mask就不用保存
if np.sum(mask[layerIdx]):
maskSavePath = os.path.join(patientTargetDir, '{}_mask.npy'.format(layerIdx))
np.save(maskSavePath, mask[layerIdx]) # 保存该层的mask
# print(maskSavePath, 'saved!')
此方案效果:在未解决该问题之前,根据我们对于脑部姿态调整的效果自检程序,未通过我们设定阈值400(脑部姿态调整之后左右信息相减只差大于0.3的像素个数超过400)的50个测试集里有10个数据,使用该方案以后,未通过自检的数据只有4个
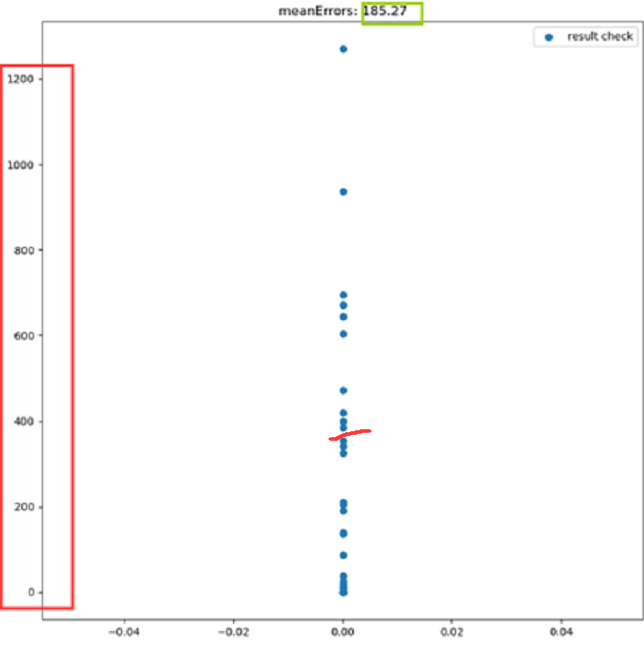
上图是未解决该问题之前,根据自检程序画的散点图,其中纵轴表示左右相减之差大于0.3的像素点个数,横轴在这里无意义,可以看出未通过自检阈值的有10个数据
以下是按照上述方案解决之后,手工检查的未通过的四个数据,主要原因是脑部本身左右就不对称