前言
看了很多,发现这个遗传算法,进化算法是一个非常有用的一个方法。而且可解释性远远强于神经网络。之前写了一篇博文,专门讲解基于DEAP库的python编程,来编写遗传算法,但是那一篇主要偏重代码,出于想要深入理解代码的含义,因此专门记下这篇博文,既是笔记,也是分享。
所有的用python实现的代码,请看这个博文:
GEAP 遗传算法/遗传编程 genetic programming + python(deap库)实现
如果需要还有这两个:
遗传算法GA和遗传编程GP有什么不同?
粒子群算法PSO 和 遗传算法GA 的相同点和不同点
概述
“物竞天择,优胜劣汰”, 达尔文提出了著名的生物进化理论,即所有的动植物都是由较早期、较原始的形式演变而来的。而遗传编程(遗传规划)则在数学和计算机科学领域应用了这一演化过程:从基数较为庞大的原始、粗糙的程序种群中通过评估适应性选择父系、进行遗传操作生成新一代种群,再判断终止条件决定是否再次迭代、生成下一代种群。类比如下:
讲到这里可否理解了,进化算法的本质就是模拟优胜劣汰,下面进入正题
启发式的理解(重点)
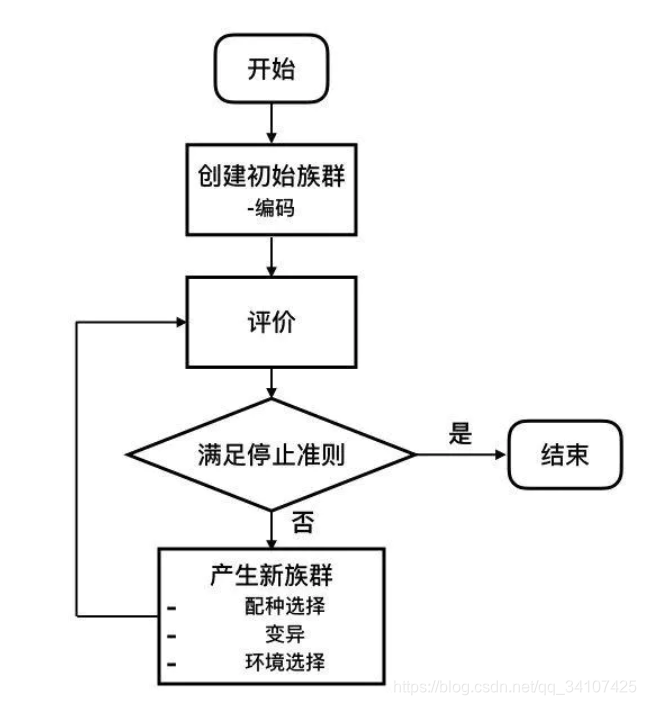
整个包含以下几个方面!一定要看一下
- 个体编码(Individual representation): 将问题的解空间编码映射到搜索空间的过程。常用的编码方式有二值编码(Binary),格雷编码(Gray),浮点编码(Floating-point)等。
简单的说就是你怎么表示一个个体,使用01010101010101来表示一个个体,还是用1238583241来表示一个个体 - 评价(Evaluation): 设定一定的准则评价族群内每个个体的优秀程度。这种优秀程度通常称为适应度(Fitness)。
基于你的个体编码,判断你的个体编码好不好的一个评价函数 - 配种选择(Mating selection): 建立准则从父代中选择个体参与育种。尽可能选择精英个体的同时也应当维护种群的多样性,避免算法过早陷入局部最优。
你要选择哪些个体参与育种,也就是参与下面那一个环节 - 变异(Variation): 变异过程包括一系列受到生物启发的操作,例如重组(Recombination),突变(mutation)等。通过变异操作,父代的个体编码以一定方式继承和重新组合后,形成后代族群。
保留下来的个体,进行重组,突变等等 - 环境选择(Environmental selection): 将父代与子代重组成新的族群。这个过程中育种得到的后代被重新插入到父代种群中,替换父代种群的部分或全体,形成具有与前代相近规模的新族群。
生下来的个体 按道理讲应该是比父类数量少的,一般都会补全到和父类的size相同,补全的方案就叫做环境选择 - 停止准则(Stopping crieterion): 确定算法何时停止,通常有两种情况:算法已经找到最优解或者算法已经选入局部最优,不能继续在解空间内搜索。
咱们看个流程图理解理解:
优化问题的定义
这个不再理解范围内,但是,如果要用python编程进化算法的话,就需要先设置这个问题。
有单优化问题:creator.create('FitnessMin', base.Fitness, weights=(-1.0, ))
- 在创建单目标优化问题时,weights用来指示最大化和最小化。此处-1.0即代表问题是一个最小化问题,对于最大化,应将weights改为正数,如1.0。
- 另外即使是单目标优化,weights也需要是一个tuple,以保证单目标和多目标优化时数据结构的统一。
- 对于单目标优化问题,weights 的绝对值没有意义,只要符号选择正确即可。
和多优化问题:creator.create('FitnessMulti', base.Fitness, weights=(-1.0, 1.0))
- 对于多目标优化问题,weights用来指示多个优化目标之间的相对重要程度以及最大化最小化。如示例中给出的(-1.0, 1.0)代表对第一个目标函数取最小值,对第二个目标函数取最大值。
个体编码
- 实数编码(Value encoding):直接用实数对变量进行编码。优点是不用解码,基因表达非常简洁,而且能对应连续区间。但是实数编码后搜索区间连续,因此容易陷入局部最优。
- 二进制编码(Binary encoding):在二进制编码中,用01两种数字模拟人类染色体中的4中碱基,用一定长度的01字符串来描述变量。其优点在于种群多样性大,但是需要解码,而且不连续,容易产生Hamming cliff(例如0111=7, 1000=8,改动了全部的4位数字之后,实际值只变动了1),在接近局部最优位置时,染色体稍有变动,就会使变量产生很大偏移(格雷编码(Gray coding)能够克服汉明距离的问题,但是实际问题复杂度较大时,格雷编码很难精确描述问题)。

- 序列编码(Permutation encoding):通常在求解顺序问题时用到,例如TSP问题。序列编码中的每个染色体都是一个序列。
- 粒子(Particles):粒子是一种特殊个体,主要用于粒子群算法。相比普通的个体,它额外具有速度、速度限制并且能记录最优位置。
初始族群的创建
- 一般族群:这是最常用的族群类型,族群中没有特别的顺序或者子族群。
toolbox.register('population', tools.initRepeat, list, toolbox.individual) - 同类群(Demes):同类群即一个族群中包含几个子族群。在有些算法中,会使用本地选择(Local selection)挑选育种个体,这种情况下个体仅与同一邻域的个体相互作用。
toolbox.register("deme", tools.initRepeat, list, toolbox.individual)
DEME_SIZES = 10, 50, 100
population = [toolbox.deme(n=i) for i in DEME_SIZES]
- 粒子群:粒子群中的所有粒子共享全局最优。在实现时需要额外传入全局最优位置与全局最优适应度给族群。
creator.create("Swarm", list, gbest=None, gbestfit=creator.FitnessMax)
toolbox.register("swarm", tools.initRepeat, creator.Swarm, toolbox.particle)
评价
评价部分是根据任务的特性高度定制的,DEAP库中并没有预置的评价函数模版。
这里给一个例子
from deap import base, creator, tools
import numpy as np
# 定义问题
creator.create('FitnessMin', base.Fitness, weights=(-1.0,)) #优化目标:单变量,求最小值
creator.create('Individual', list, fitness = creator.FitnessMin) #创建Individual类,继承list
# 生成个体
IND_SIZE = 5
toolbox = base.Toolbox()
toolbox.register('Attr_float', np.random.rand)
toolbox.register('Individual', tools.initRepeat, creator.Individual, toolbox.Attr_float, n=IND_SIZE)
# 生成初始族群
N_POP = 10
toolbox.register('Population', tools.initRepeat, list, toolbox.Individual)
pop = toolbox.Population(n = N_POP)
# 定义评价函数
def evaluate(individual):
return sum(individual), #注意这个逗号,即使是单变量优化问题,也需要返回tuple
# 评价初始族群
toolbox.register('Evaluate', evaluate)
fitnesses = map(toolbox.Evaluate, pop)
for ind, fit in zip(pop, fitnesses):
ind.fitness.values = fit
print(ind.fitness.values)
# 结果:
# (2.593989197511478,)
# (1.1287944225903104,)
# (2.6030877077096717,)
# (3.304964061515382,)
# (2.534627558467466,)
# (2.4697149450205536,)
# (2.344837782191844,)
# (1.8959030773060852,)
# (2.5192475334239,)
# (3.5069764929866585,)
配种选择
锦标赛
deap.tools.selTournament(individuals, k, tournsize, fit_attr = 'fitness')
锦标赛选择顾名思义,就是模拟锦标赛的方式,首先在族群中随机抽取tournsize个个体,然后从中选取具有最佳适应度的个体,将此过程重复k次,获得育种族群。tournsize越大,选择强度(selection intensity)越高,在选择之后留下的育种族群的平均适应度也就越高。比较常用的tournsize是2。
假设tournsize是2,就是在原来的种群中取出来2个,然后再这两个中选一个最适应的,然后重复k次,我们就可以得到k个最适应的,这个就是育种族群,用来后面生孩子重组基因的;
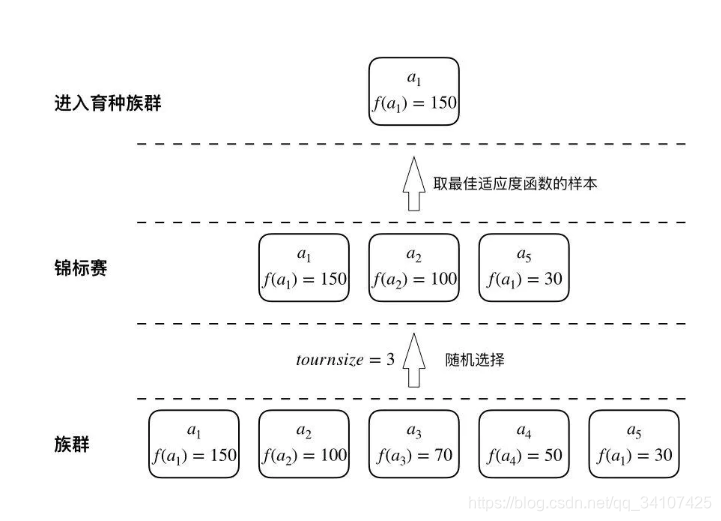
如图,假设这个族群有5个人,tournsize为3,就是选出来3个人,然后在3个人中选取一个最好的
锦标赛选择相比于轮盘赌选择,通常能够有更快的收敛速度,在实际场景中应用较多。
轮盘赌选择
deap.tools.selRoulette(individuals, k, fit_attr = 'fitness')
在轮盘赌选择中,每个个体a被选中的概率P(a)与他的评价函数f(a)成正比
但是如果评价函数出现负数,就不适用
并且很多文章指出来,轮盘赌选择的效果并不好
随机普遍抽样选择
deap.tools.selStochasticUniversalSampling(individuals, k, fit_attr = 'fitness')
随机普遍抽样选择是一种有多个指针的轮盘赌选择,其优点是能够保存族群多样性,而不会像轮盘赌一样,有较大几率对重复选择最优个体。
在与前文相同的例子中使用随机普遍抽样选择,设定指针数k为3,那么指针间距即为,如下图所示:
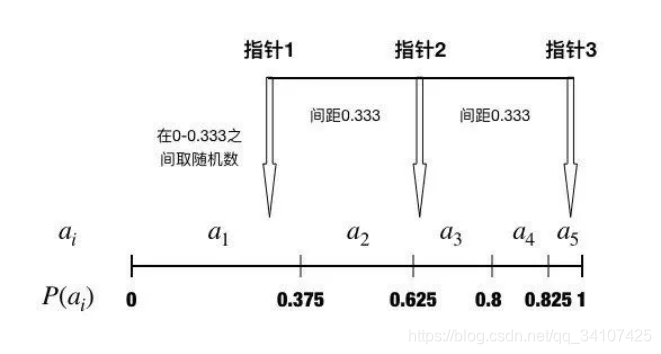
就是我一选就选3个个体,一选选3个,保证了多样性
变异
单点交叉
deap.tools.cxOnePoint(ind1, ind2)
- 最简单的交叉方式,选择一个切口,将两条基因切开之后,交换尾部基因段。尽管该方法非常简单,但是多篇文章指出,该算法在各种实验中性能都被其他交叉算法吊打,因此算是一种不建议使用的loser algorithm

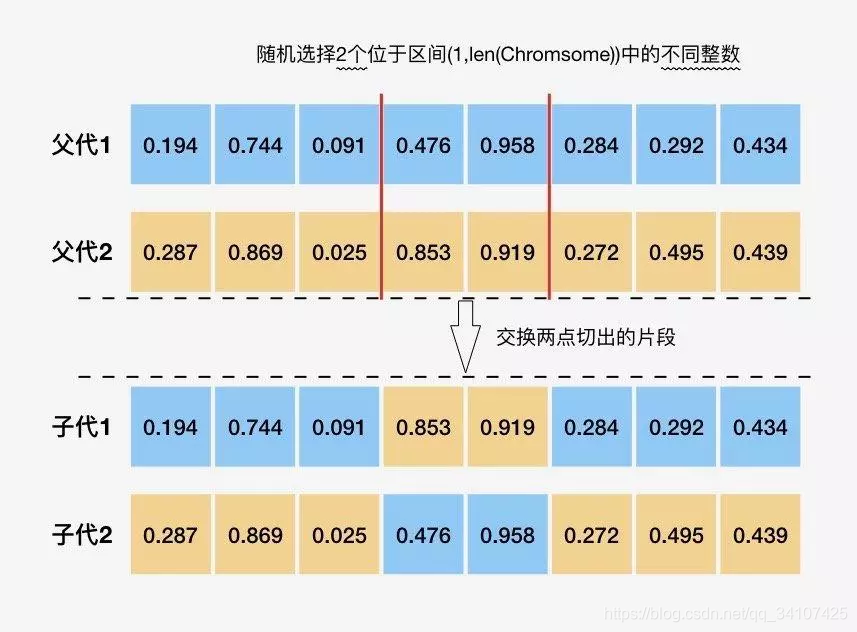
两点交叉
deap.tools.cxTwoPoint(ind1, ind2)
- 用两个点切开基因之后,交换切出来的基因段
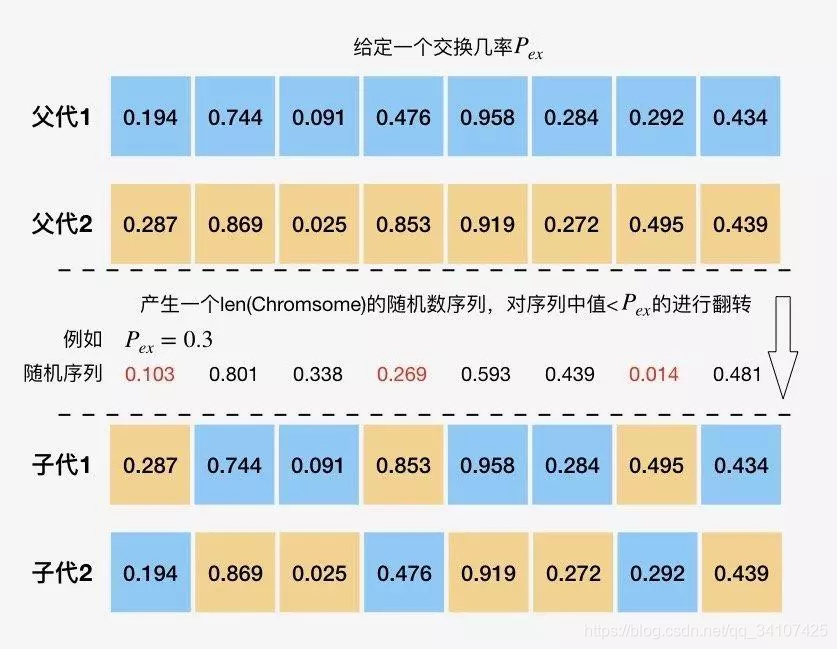
均匀交叉
deap.tools.cxUniform(ind1, ind2, indpb)
- 指定一个变异几率,两父代中的每个基因都以该几率交叉。
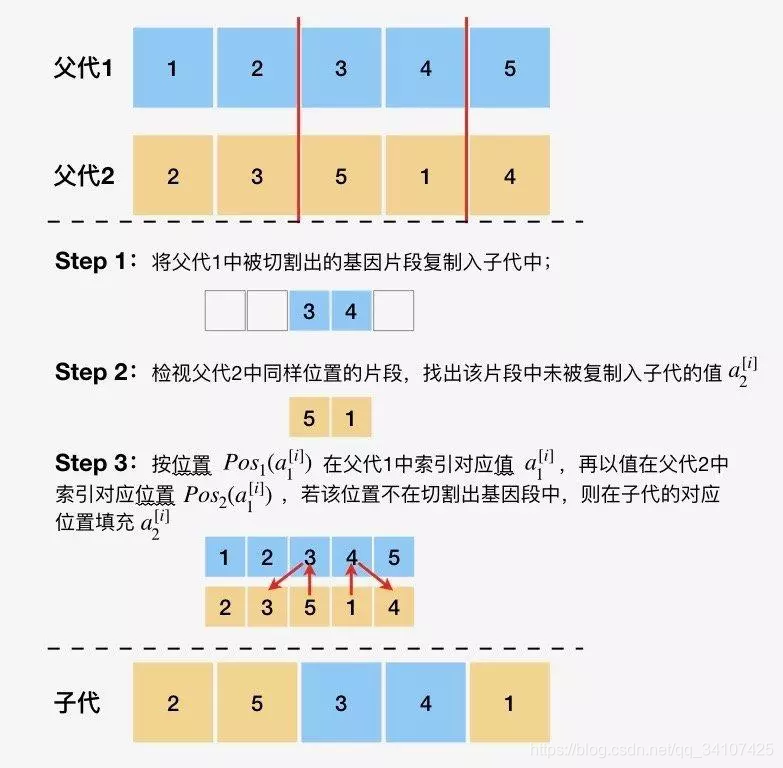
部分匹配交叉
deap.tools.cxPartialyMatched(ind1, ind2)
- 部分匹配交叉主要用于序列编码的个体
- 这个太难叙述了,能看懂就看,看不懂就记住在哪里用就好了,就是上面那句话
突变
高斯突变
tools.mutGaussian(individual, mu, sigma, indpb)
- 对个体序列中的每一个基因按概率变异,变异后的值为按均值为mu,方差为sigma的高斯分布选取的一个随机数。如果不希望均值发生变化,则应该将设mu为0。
乱序突变
tools.mutShuffleIndexes(individual, indpb)
- 将个体序列打乱顺序,每个基因位置变动的几率由indpb给出。
位翻转突变
tools.mutFlipBit(individual, indpb)
对个体中的每一个基因按给定对变异概率取非。
均匀整数突变
tools.mutUniformInt(individual, low, up, indpb)
对序列中的每一位按概率变异,变异后的值为[low, up]中按均匀分布随机选取的一个整数。
环境选择
完全重插入(Pure reinsertion)
- 产生与父代个体数量相当的配种个体,直接用配种个体生成新一代族群。
均匀重插入(Uniform reinsertion)
- 产生比父代个体少的配种个体,用配种个体随机均匀地替换父代个体。
精英重插入(Elitist reinsertion)
- 产生比父代个体少的配种个体,选取配种后代中适应度最好的一些个体,插入父代中,取代适应度较低的父代个体。
精英保留重插入(Fitness-based reinsertion)
- 产生比父代个体多的配种个体,选取其中适应度最大的配种个体形成新一代族群。
通常来说后两种方式由于精英保留的缘故,收敛速度较快,因此比较推荐。
进化算法的python实现
GEAP 遗传算法/遗传编程 genetic programming + python(deap库)实现
进化算法的优缺点
优点
-
泛用性强,对连续变量和离散变量都能适用;
-
不需要导数信息,因此不要求适应度函数的连续和可微性质(或者说不需要问题内在机理的相关信息);
-
可以在解空间内大范围并行搜索;
-
不容易陷入局部最优;
-
高度并行化,并且容易与其他优化方法整合。
-
对于凸优化问题,相对基于梯度的优化方法(例如梯度下降法,牛顿/拟牛顿法)收敛速度更慢;
-
进化算法需要在搜索空间投放大量个体来搜索最优解。对于高维问题,由于搜索空间随维度指数级膨胀,需要投放的个体数也大幅增长,会导致收敛速度变慢;
-
设计编码方式、适应度函数以及变异规则需要大量经验。
简单说最大的缺点就是慢
参考:
GEAP 遗传算法/遗传编程 genetic programming + python(deap库)实现
【遗传编程/基因规划】Genetic Programming