本文转载于https://mp.weixin.qq.com/s?__biz=MzIxNzEzODA5NQ==&mid=2649373408&idx=1&sn=232c2cb36dfde647a5c1f5103d213ddd&chksm=8fe0cc20b897453613450906548a7344d17b3a850be9103cb33b58b0c521212c81de1750e160&mpshare=1&scene=23&srcid=0619cPVePyZ7ifZDQwcCQByc#rd
为了确定潜在新种的分类地位,自然要下载诸多模式种的16S rDNA序列来构建系统发育树,一株一株的手动下载,好累!一个python脚本,可以从LPSN(http://www.bacterio.net/)下载指定属下所有模式种的16S rDNA序列,解放你的双手。
我们先来看一下脚本的参数:
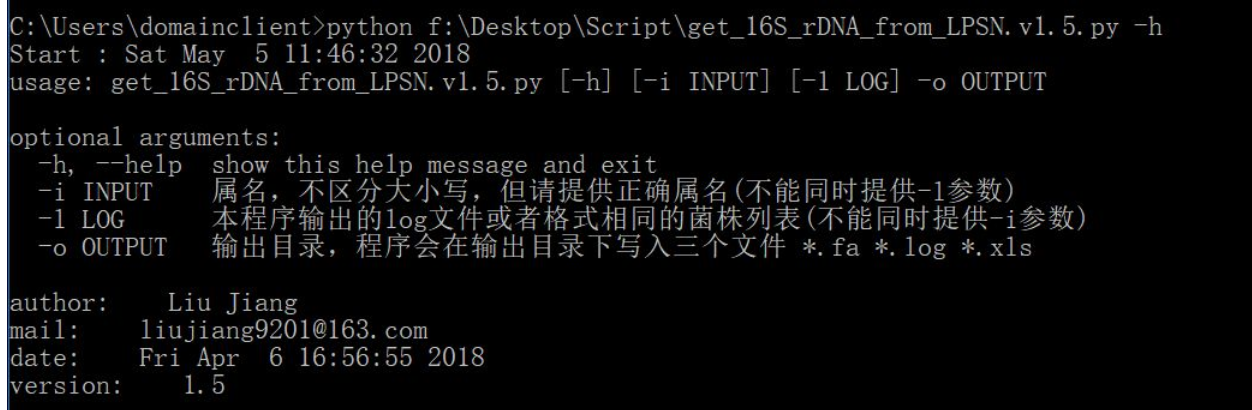
-h显示帮助信息;
-i 指定查询的属名,要求在LPSN中存在;
-l 本脚本输出的log文件,接下来会有详细介绍;
-o 指定输出的文件夹路径,如果文件夹不存在会自动创建。
脚本可以接受的输入包括两种方式:
(1)-i参数指定的属名,脚本会通过查询LPSN获取该属下的所有模式种信息,包括属种名,模式种编号,NCBI登录号,并将这些信息写入“属名.xls“文件中。然后通过NCBI的登录号前往NCBI下载该模式种的16SrDNA序列,并写入“属名.fa“文件中,同时将下载失败的模式种信息写入”属名.log“文件中。
(2)本程序生成的”属名.log”文件可以通过-l参数作为输入,脚本会再次尝试下载log中记录的下载失败的模式种。
需要注意的是,LPSN中给出的某些模式种的NCBI登录号为该模式种的全基因组序列,对于这种情况,脚本不会将序列写入“属名.fa“文件。下面是下载示例:
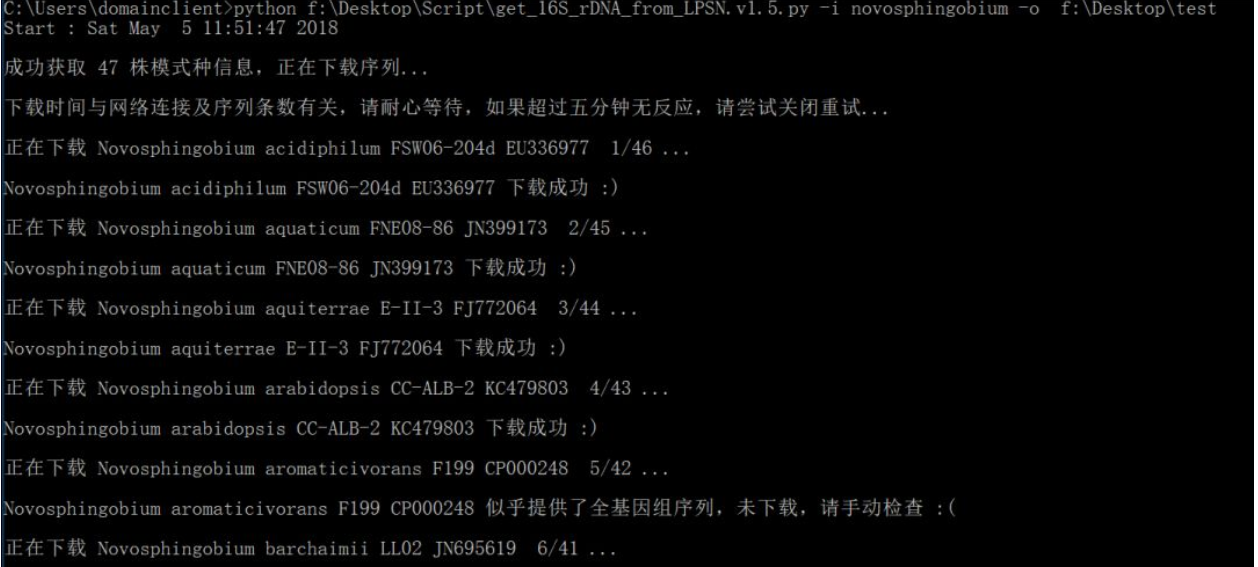
下载完成后会给出一个下载统计报告:
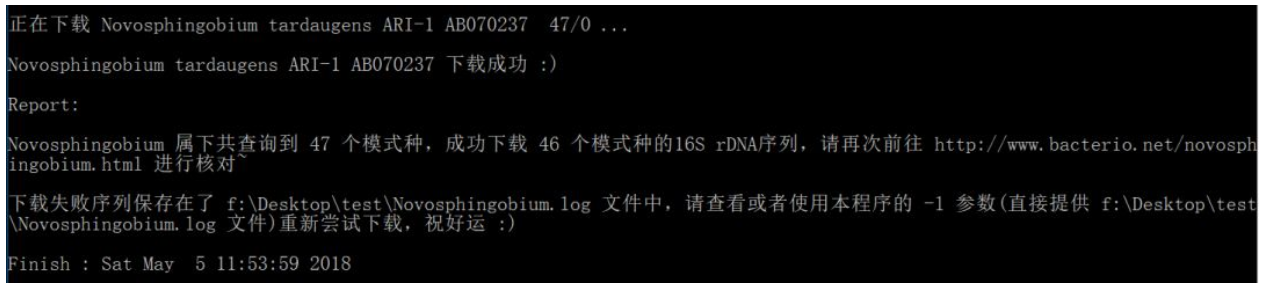
下载速度与网速和模式种个数有关,请耐心等待,另外,NCBI白天的连接速度要好于晚上,不知道这是否是个玄学问题,所以建议白天下载,如果遇到长时间(5分钟)界面无反应的情况可以尝试中断然后重新下载。